Definierte Blockade
© Wiley-VCH, re-use with credit to 'Angewandte Chemie' and a link to the original article.
Enzymatisches Photocaging zur Erforschung der Genregulation durch DNA-Methylierung
Das Anknüpfen und Abspalten von Methylgruppen an die DNA spielt eine wichtige Rolle bei der Genregulation. Um diese Mechanismen genauer erforschen zu können, hat ein deutsches Team eine neue Methode entwickelt, mit der sich Methylierungsstellen gezielt blockieren und zeitlich definiert durch Bestrahlung mit Licht wieder freisetzen lassen (Photocaging). Wie in der Zeitschrift Angewandte Chemie berichtet, wird das benötigte Reagenz in situ enzymatisch hergestellt.
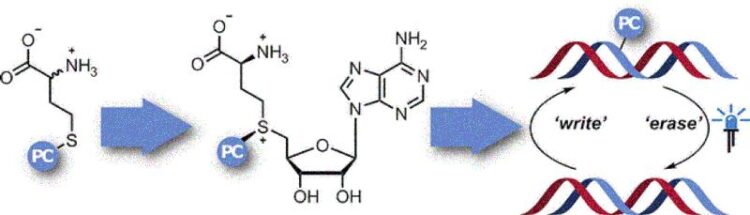
Obwohl sie sehr verschieden aussehen und vollkommen unterschiedliche Aufgaben erfüllen, haben alle Zellen unseres Körpers die identische DNA, verwenden aber nicht die gleichen Gene. Bestimmte Gene werden an-, andere abgeschaltet – je nach Zelltyp und nach Zeitpunkt. Als „Schalter“ fungieren chemische Veränderungen der DNA-Bausteine, sogenannte epigenetische Modifikationen. Ein bedeutender Regulationsmechanismus ist deren Methylierung und Demethylierung, d.h. das Anknüpfen bzw. Abspalten einer Methylgruppe (–CH3). Krebszellen beispielsweise zeigen gegenüber gesunden Zellen veränderte Methylierungsmuster. Bei einer Methylierung übertragen Enzyme, die Methyltransferasen (MTasen), eine Methylgruppe von S-Adenosyl-L-methionin (AdoMet) auf das Zielmolekül.
Um die Aufgabe und Funktionsweise dieser Regulation genauer zu erforschen und Methylierungsmuster zu kartieren, wären „Werkzeuge“ wünschenswert, mit denen sich eine Methylierung spezifisch an bestimmten Zielstellen hemmen und diese Inhibierung zeitlich definiert wieder aufheben ließe. Das Forschungsteam um Andrea Rentmeister wählte dazu eine Methode, die als Photocaging bezeichnet wird: Ein „Photocage“, d.h. eine Molekülgruppe, die bei Bestrahlung zerfällt, etwa eine 2-Nitrobenzyl-Gruppe, blockiert zunächst den Zielort, eine gezielte Bestrahlung mit Licht wirkt dann als „Schalter“, der die Blockade wieder aufhebt.
Die Idee war, AdoMet-Analoga mit einem Photocage zu bestücken, der dann auf die Methylierungsstellen übertragen wird. Allerdings zerfallen die AdoMet-Analoga in wässriger Lösung und können nicht in Zellen eindringen. Daher wollte das Team von der Universität Münster diese in situerzeugen. Im Organismus wird AdoMet durch das Enzym Methionin-Adenosyl-Transferase (MAT) aus der Aminosäure Methionin hergestellt. Zur Synthese der AdoMet-Analoga benötigten sie Methionin mit angeknüpftem Nitrobenzyl-Photocage – und eine MAT, die ein solches verändertes Substrat verwenden kann. Ausgehend von einem MAT-Enzym aus einem Einzeller (Cryptosporidium hominis) gelang es den Forscherinnen und Forschern, durch gezielte Veränderung von Aminosäuren ein Enzym mit einer vergrößerten hydrophoben „Bindungstasche“ zu entwickeln, die ausreichend Platz für die Nitrobenzyl-Gruppe bietet. Eine Kristallstrukturanalyse veranschaulichte, wie das AdoMet-Analogon in der Tasche dieser Photocaging-MAT (PC-MAT) gebunden ist. Ausgehend von diesen Erkenntnissen stellte das Team eine weitere PC-MAT auf Basis eines thermostabilen MAT-Enzyms aus dem Archaeon Methanocaldococcus jannaschii her.
Beide PC-MATs waren mit DNA- und RNA-MTasen kompatibel und ermöglichten ein Anknüpfen von Photocages an alle natürlichen Methylierungsstellen einer Plasmid-DNA. Bestrahlung mit Licht hob die Blockaden wieder auf.
Angewandte Chemie: Presseinfo 22/2020
Autor/-in: Andrea Rentmeister, Universität Münster (Germany), https://www.uni-muenster.de/Cells-in-Motion/de/people/all/rentmeister-a.php
Angewandte Chemie, Postfach 101161, 69451 Weinheim, Germany
Originalpublikation:
https://doi.org/10.1002/ange.202012623
Weitere Informationen:
Media Contact
Alle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Das Mikrobiom verändert sich dynamisch und begünstigt wichtige Funktionen für den Wirt
Ein interdisziplinäres Forschungsteam des Kieler SFB 1182 untersucht am Beispiel von Fadenwürmern, welche Prozesse die Zusammensetzung des Mikrobioms in Wirtslebewesen steuern. Alle vielzelligen Lebewesen – von den einfachsten tierischen und…

Wasser im Boden – genaue Daten für Landwirtschaft und Klimaforschung
Die PTB präsentiert auf der Woche der Umwelt, wie sich die Bodenfeuchte mithilfe von Neutronenstrahlung messen lässt. Die Bodenfeuchte hat nicht nur Auswirkungen auf die Landwirtschaft, sondern ist als Teil…

Bioreaktor- und Kryotechnologien für bessere Wirkstofftests mit humanen Zellkulturen
Medizinische Wirkstoffforschung… Viele Neuentwicklungen von medizinischen Wirkstoffen scheitern, weil trotz erfolgreicher Labortests mit Zellkulturen starke Nebenwirkungen bei Probanden auftreten. Dies kann passieren, wenn zum Beispiel die verwendeten Zellen aus tierischem…





















