Wie funktioniert der kleinste Motor der Welt?

Max-Planck-Wissenschaftler entschlüsseln Nanomechanik einer „molekularen Maschine“, die ATP, den zentralen Energieträger in unserem Körper, produziert
Das Molekül Adenosintriphosphat (ATP) ist als universeller Träger chemischer Energie in allen Organismen von zentraler Bedeutung. Produziert wird es von einem außerordentlich effizienten Enzym, der F-ATPase. Der menschliche Körper verbraucht pro Tag etwa 50 kg ATP, bei großer körperlicher Anstrengung sogar bis zu einer Tonne. Wissenschaftlern vom Göttinger Max-Planck-Institut für biophysikalische Chemie ist es jetzt gelungen, einzelne Phasen der in diesem Enzym ablaufenden Umwandlung mechanischer in chemische Energie mit Hilfe aufwändiger Computersimulationen bis ins atomare Detail aufzuklären (Nature Structural Biology, März 2002). Zwei Jahre Rechenzeit eines Großcomputers waren erforderlich, damit man jetzt in einem Videofilm den Nanomotor in Aktion sehen kann.
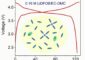
Das Enzym F-ATPase besteht aus zwei Teilen (Abb. 1, links), einem Kopfteil (F1-Teil, grün) und einem Fußteil (F0-Teil, grau). Der „Fuß“ ist direkt in der Membran von Mitochondrien verankert, den „Kraftwerken“ der Zelle. Die Synthese von ATP wird durch eine faszinierende Nano-Mechanik getrieben. Die zur Synthese erforderliche Energie stammt – wie bei einer elektrischen Batterie – aus einer elektrischen Spannung, verursacht durch eine Differenz in der Konzentration geladener Wasserstoffionen (Protonen, H+) zwischen dem Inneren (oben) und dem Äußeren der Mitochondrien (unten). Dieser Ladungsunterschied wird über den Fußteil ausgeglichen (roter Pfeil), wodurch der zylinderförmige Rotor (dunkelgrau) – ähnlich wie bei einem Elektromotor – in Bewegung gesetzt wird. Da der Kopfteil über einen Stator (gelb) fixiert ist, dreht sich die mit dem Fußteil verbundene asymmetrische Achse (orange) innerhalb des Kopfteils. Das ganze Enzym ist nur etwa 25 Millionstel Millimeter groß – das entspricht etwa dem 2000-ten Teil des Durchmesser eines Haares – und damit der kleinste bekannte Motor der Welt.
Der Kopfteil besteht aus sechs Komponenten (Abb. 1, rechts), von denen drei – die sogenannten β-Untereinheiten (grün) – aktiv an der ATP-Synthese beteiligt sind. Jede β-Untereinheit enthält eine Bindungstasche, in der die eigentliche ATP-Synthese aus Adenosindiphosphat (ADP) und Phosphat stattfindet. Erste Hinweise auf den Funktionsmechanismus des Enzyms ergaben sich aus der 1994 aufgeklärten räumlichen Struktur des Kopfteils (Abb. 1, rechts). Eine der drei Bindungstaschen ist in dieser Struktur nämlich leer, die zweite lediglich mit dem Ausgangsmolekül ADP besetzt und die dritte enthält das eigentliche Reaktionsprodukt, das ATP. Die beiden Moleküle in den Bindungstaschen werden also unterschiedlich stark gehalten – und das, obwohl die drei β-Untereinheiten in ihrem chemischen Aufbau völlig identisch sind. Doch sie unterscheiden sich in ihrer räumlichen Struktur (Abb.2, links): Während die untere Hälfte der leeren β-Untereinheit von der Achse weg nach außen geklappt ist (grau), sind die beiden anderen, beladenen β-Untereinheiten geschlossen (farbig, Pfeil).
 Diese Beobachtung ließ vermuten, dass die vom Fußteil bewirkte Drehung der Achse (Pfeil) ein wechselseitiges Öffnen und Schließen der drei β-Untereinheiten erzwingt, in deren Bindungstaschen reihum ein Synthesezyklus stattfindet – vergleichbar den Kolbenbewegungen eines Ottomotors. Die durch die Drehung der Achse übertragene Energie dient dazu, das nach der Synthese in der Bindungstasche fest gebundene ATP-Molekül freizusetzen. Wie die rotierende Achse die Strukturänderungen in den β-Untereinheiten auslöst, war bisher nicht bekannt. Auch wusste man nicht, auf welche Weise Energie mechanisch auf die Bindungstaschen übertragen wird, so dass dort die Bindungsstärke von ATP sinkt. Schließlich war unklar, wie die leere Untereinheit nach der Freisetzung des ATP-Moleküls wieder in ihren geschlossenen Ausgangszustand zurückkehrt.
Diese Beobachtung ließ vermuten, dass die vom Fußteil bewirkte Drehung der Achse (Pfeil) ein wechselseitiges Öffnen und Schließen der drei β-Untereinheiten erzwingt, in deren Bindungstaschen reihum ein Synthesezyklus stattfindet – vergleichbar den Kolbenbewegungen eines Ottomotors. Die durch die Drehung der Achse übertragene Energie dient dazu, das nach der Synthese in der Bindungstasche fest gebundene ATP-Molekül freizusetzen. Wie die rotierende Achse die Strukturänderungen in den β-Untereinheiten auslöst, war bisher nicht bekannt. Auch wusste man nicht, auf welche Weise Energie mechanisch auf die Bindungstaschen übertragen wird, so dass dort die Bindungsstärke von ATP sinkt. Schließlich war unklar, wie die leere Untereinheit nach der Freisetzung des ATP-Moleküls wieder in ihren geschlossenen Ausgangszustand zurückkehrt.
Rainer Böckmann und Helmut Grubmüller vom Max-Planck-Institut für biophysikalische Chemie gelang es nun, die durch die Drehung der Achse ausgelösten Strukturänderungen innerhalb des Enzyms F1-ATPase mit Hilfe aufwändiger Computersimulationen bis hin zu den aktiven Synthesezentren im atomaren Detail zu verfolgen. Für diese Simulationen haben die Wissenschaftler den F1-Teil des Proteins im Computer Atom für Atom (in Abb. 1, links, als Kugeln dargestellt) nachgebaut und mit einer großen Zahl von Wassermolekülen (blaue Punkte) und gelösten Salzionen (rot, gelb) umgeben, so dass sich das virtuelle Protein quasi in seiner natürlichen Umgebung befand. Insgesamt umfasste das Modell etwa 200.000 Atome, deren Bewegungen in einer anschließenden so genannten Molekulardynamik-Simulation genau berechnet wurden. Für die Simulation brauchte ein aus 120 Prozessoren bestehender Hochleistungsparallelrechner der Gesellschaft für Wissenschaftliche Datenverarbeitung Göttingen (GWDG) annähernd zwei Jahre Rechenzeit. Entstanden ist eine Filmsequenz, in der jedes Detail der Strukturänderung am Bildschirm betrachtet und analysiert werden kann.
 Die Simulationen offenbaren eine faszinierende „Nano-Mechanik“ innerhalb der β-Untereinheit, an der hauptsächlich die in Abb. 2 (links) rot gefärbten Teile beteiligt sind. Dabei wird die Kippbewegung der unteren Hälfte der Untereinheit – ähnlich wie bei einem mechanischen Hebelwerk – über eine Reihe von Strukturänderungen, die sich Domino-artig fortpflanzen, in eine auf atomarer Ebene präzise koordinierte Verformung der Bindungstasche umgesetzt. Eine wichtige Rolle spielen dabei drei positiv geladene Aminosäuren (Arginine), die in genau definierter Abfolge von den negativ geladenen Phosphatgruppen des ATP-Moleküls (pinkfarben) weggezogen werden (Pfeile), wodurch die wechselseitige elektrostatische Anziehung verringert wird (Abb. 2, rechts). Auf diese Weise lockert sich der „Griff“ um das gebundene ATP-Molekül, so dass es die Bindungstasche verlassen kann.
Die Simulationen offenbaren eine faszinierende „Nano-Mechanik“ innerhalb der β-Untereinheit, an der hauptsächlich die in Abb. 2 (links) rot gefärbten Teile beteiligt sind. Dabei wird die Kippbewegung der unteren Hälfte der Untereinheit – ähnlich wie bei einem mechanischen Hebelwerk – über eine Reihe von Strukturänderungen, die sich Domino-artig fortpflanzen, in eine auf atomarer Ebene präzise koordinierte Verformung der Bindungstasche umgesetzt. Eine wichtige Rolle spielen dabei drei positiv geladene Aminosäuren (Arginine), die in genau definierter Abfolge von den negativ geladenen Phosphatgruppen des ATP-Moleküls (pinkfarben) weggezogen werden (Pfeile), wodurch die wechselseitige elektrostatische Anziehung verringert wird (Abb. 2, rechts). Auf diese Weise lockert sich der „Griff“ um das gebundene ATP-Molekül, so dass es die Bindungstasche verlassen kann.
Die Simulationen zeigen ferner, dass sich die leere β-Untereinheit nach Drehen der Achse sehr schnell – wie eine zurückschnellende Feder – schließt. Dieser Befund überraschte die Wissenschaftler, da bisher allgemein angenommen wurde, dass die leere β-Untereinheit erst durch die erneute Bindung eines ADP-Moleküls in den geschlossenen Zustand gezwungen wird. Durch diese neue Einsicht wird die Analogie zum Ottomotor noch deutlicher: Auch dort werden die Kolben nicht erst durch das Einströmen des Benzin-Luft-Gemischs nach oben bewegt, sondern bereits vorher und synchronisiert mit der Bewegung der Kurbelwelle.
Dieses Forschungsergebnis zeigt, wie dank fortschreitender Verbesserung der Simulationsverfahren und rasch wachsender Rechenleistung eine zunehmende Zahl wichtiger biologischer Funktionsprozesse auf atomarer Ebene simuliert werden können. Über die traditionelle Bioinformatik hinausgehend, ermöglichen solche Simulationen, biologische Prozesse auf der Grundlage physikalisch-chemischer Gesetzmäßigkeiten zu erklären.
Originalarbeit:
Rainer A. Böckmann und Helmut Grubmüller: Nanoseconds molecular dynamics simulation of primary mechanical energy transfer steps in F1-ATP synthase, Nature Structural Biology, März 2002
Weitere Informationen erhalten Sie von:
Dr. Helmut Grubmüller
Max-Planck-Institut für biophysikalische Chemie, Göttingen
Arbeitsgruppe für theoretische molekulare Biophysik
Tel.: 05 51 / 2 01 – 17 63 oder – 13 34
Fax.: 05 51 / 2 01 – 10 89
E-Mail: hgrubmu@gwdg.de
Media Contact
Alle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Diamantstaub leuchtet hell in Magnetresonanztomographie
Mögliche Alternative zum weit verbreiteten Kontrastmittel Gadolinium. Eine unerwartete Entdeckung machte eine Wissenschaftlerin des Max-Planck-Instituts für Intelligente Systeme in Stuttgart: Nanometerkleine Diamantpartikel, die eigentlich für einen ganz anderen Zweck bestimmt…
Neue Spule für 7-Tesla MRT | Kopf und Hals gleichzeitig darstellen
Die Magnetresonanztomographie (MRT) ermöglicht detaillierte Einblicke in den Körper. Vor allem die Ultrahochfeld-Bildgebung mit Magnetfeldstärken von 7 Tesla und höher macht feinste anatomische Strukturen und funktionelle Prozesse sichtbar. Doch alleine…

Hybrid-Energiespeichersystem für moderne Energienetze
Projekt HyFlow: Leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem für moderne Energienetze. In drei Jahren Forschungsarbeit hat das Konsortium des EU-Projekts HyFlow ein extrem leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem entwickelt, das einen…