Einheitliche Qualitätsstandards für die Virenforschung
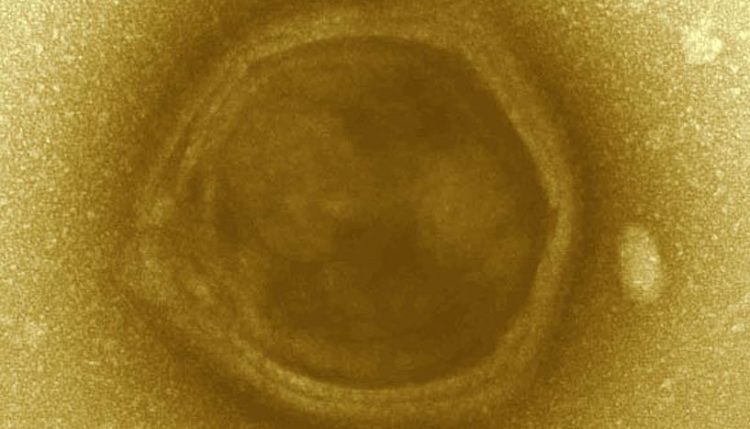
Elektronenmikroskopische Aufnahme eines Klosneuvirus-Partikels, der mit Hilfe genomischer Methoden durch ForscherInnen der Universität Wien in einer Kläranlage in Klosterneuburg entdeckt wurde. Größenskala - 50nm (© Schulz et al.).
Mikroorganismen in auf und um unseren Planeten wird nachgesagt in ihrer Zahl die Anzahl der Sterne in der Milchstraße zu übersteigen. Die Gesamtanzahl der Viren ist jedoch um ein Vielfaches höher. Zahlreiche Arten sind derzeit noch unbekannt und der Forschung nicht zugänglich. Die Anzahl derartiger Datensätze verdreifacht sich jedes Jahr.
Ein internationales Forschungsteam um den Mikrobiologen Thomas Rattei von der Universität Wien hat nun neue Standards zur Einordnung der Forschungsdaten definiert. Die Ergebnisse erscheinen aktuell im Fachjournal Nature Biotechnology.
Um einheitliche Qualitätsstandards in diesem wichtigen Forschungsbereich einzuführen, hat sich vor zwei Jahren ein internationales Konsortium formiert. Seitens der Universität Wien arbeitete Thomas Rattei vom Department für Mikrobiologie und Ökosystemforschung in dieser Gruppe mit.
Das Team um den Mikrobiologen erforscht seit Jahren die Evolution viraler Proteinfamilien und entwickelt dafür spezielle Bioinformatik-Werkzeuge und -Datenbanken.
Rattei argumentiert: „Viren sind nicht nur wichtige Krankheitserreger des Menschen, sondern essenzielle Bestandteile aller Ökosysteme. Deren Erforschung mit Hilfe moderner Genomik-Methoden ist nur möglich, wenn die erzeugten großen Datenmengen einheitlichen Qualitätsstandards gehorchen.“
Das internationale Forschungsteam hat Inhalt und Struktur der minimalen Informationen definiert, die in der Genomforschung unkultivierbarer Viren wichtig sind. Dies umfasst Angaben zu Probenmaterial, Virus-Identifikation, Sequenzierung und Datenqualität.
Die Angabe dieser Informationen wird für künftige Forschungsprojekte obligatorisch sein. Im Rahmen der aktuellen Publikation im Fachjournal Nature Biotechnology werden diese neuen Standards vorgestellt und durch Richtlinien und Praxisempfehlungen ergänzt.
Eine zentrale Rolle nahm bei der Koordination dieses Projekts das „Genomic Standards Consortium (GSC)“ ein. Diese, für alle WissenschafterInnen offene, Gemeinschaft engagiert sich seit vielen Jahren für die Erstellung von Standards in der Genomforschung und hat schon zahlreiche internationale Projekte in diesem Bereich koordiniert. Vom 20. Bis 23. Mai 2019 wird das GSC auch seine Jahrestagung an der Universität Wien abhalten.
„Diese Standards schließen eine wichtige Lücke und sind ein wirksames Instrument, um angesichts des massiven Wachstums der Sequenzdaten von Viren und Mikroorganismen vergleichende Analysen durchführen zu können“, so Rattei und ergänzt: „Sie bieten die Möglichkeit, Barrieren für den Datenaustausch niederzubrechen und die Reproduzierbarkeit der Genomforschung sicherzustellen. Die Notwendigkeiten sehen wir BioinformatikerInnen in unserer täglichen Arbeit immer wieder.“
Publikation in „Nature Biotechnology „
Simon Roux, Evelien M Adriaenssens, Bas E Dutilh, Eugene V Koonin, Andrew M Kropinski, Mart Krupovic, Jens H Kuhn, Rob Lavigne, J Rodney Brister, Arvind Varsani, Clara Amid, Ramy K Aziz, Seth R Bordenstein, Peer Bork, Mya Breitbart, Guy R Cochrane, Rebecca A Daly, Christelle Desnues, Melissa B Duhaime, Joanne B Emerson, François Enault, Jed A Fuhrman, Pascal Hingamp, Philip Hugenholtz, Bonnie L Hurwitz, Natalia N Ivanova, Jessica M Labonté, Kyung-Bum Lee, Rex R Malmstrom, Manuel Martinez-Garcia, Ilene Karsch Mizrachi, Hiroyuki Ogata, David Páez-Espino, Marie-Agnès Petit, Catherine Putonti, Thomas Rattei, Alejandro Reyes, Francisco Rodriguez-Valera, Karyna Rosario, Lynn Schriml, Frederik Schulz, Grieg F Steward, Matthew B Sullivan, Shinichi Sunagawa, Curtis A Suttle, Ben Temperton, Susannah G Tringe, Rebecca Vega Thurber, Nicole S Webster, Katrine L Whiteson, Steven W Wilhelm, K Eric Wommack, Tanja Woyke, Kelly C Wrighton, Pelin Yilmaz, Takashi Yoshida, Mark J Young, Natalya Yutin, Lisa Zeigler Allen, Nikos C Kyrpides & Emiley A Eloe-Fadrosh
Minimum Information about an Uncultivated Virus Genome (MIUViG)
DOI: nbt.4306
Univ.-Prof. Mag. Dr. Thomas Rattei
Department für Mikrobiologie und Ökosystemforschung Research Network Chemistry meets Microbiology
Universität Wien
1090 – Wien, Althanstraße 14 (UZA I)
+43-1-4277-766 80
thomas.rattei@univie.ac.at
Media Contact
Weitere Informationen:
http://www.univie.ac.at/Alle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Neue universelle lichtbasierte Technik zur Kontrolle der Talpolarisation
Ein internationales Forscherteam berichtet in Nature über eine neue Methode, mit der zum ersten Mal die Talpolarisation in zentrosymmetrischen Bulk-Materialien auf eine nicht materialspezifische Weise erreicht wird. Diese „universelle Technik“…

Tumorzellen hebeln das Immunsystem früh aus
Neu entdeckter Mechanismus könnte Krebs-Immuntherapien deutlich verbessern. Tumore verhindern aktiv, dass sich Immunantworten durch sogenannte zytotoxische T-Zellen bilden, die den Krebs bekämpfen könnten. Wie das genau geschieht, beschreiben jetzt erstmals…

Immunzellen in den Startlöchern: „Allzeit bereit“ ist harte Arbeit
Wenn Krankheitserreger in den Körper eindringen, muss das Immunsystem sofort reagieren und eine Infektion verhindern oder eindämmen. Doch wie halten sich unsere Abwehrzellen bereit, wenn kein Angreifer in Sicht ist?…





















