Neue Methode vereinfacht die funktionelle Genomforschung

Ein neues Werkzeug erleichtert Genetikern ihre Arbeit: pGBT-RP2.1, das springende Genkonstrukt, färbt die von ihm inaktivierten Proteine rot. Dadurch hilft es bei der zielgenauen und systematischen Erforschung des Erbguts.
Wichtig ist das neue und vielseitige Werkzeug für das Gebiet der funktionellen Genomforschung (functional genomics). Nun können in Zukunft z.B. Erbkrankheiten schneller dem dafür verantwortlichen Gen zugeordnet werden. Entwickelt wurde das Verfahren im Tiermodell Zebrafisch, es lässt sich aber auch bei anderen Modellorganismen einsetzen.
Beteiligt an der Entwicklung des sogenannten Transposons waren Kölner Genetiker unter Leitung von Professor Matthias Hammerschmidt vom Institut für Entwicklungsbiologie und amerikanische Wissenschaftler von der Mayo Clinic und der Universität von Minnesota. Der Artikel „In vivo protein trapping produces a functional expression codex of the vertebrate proteome“ ist in der aktuellen Ausgabe von Nature Methods erschienen.
„Springende Gene“, sogenannte Transposons, sind frei bewegliche Teile der DNA. Sie werden von Genetikern dazu benutzt, fremde Gene in die DNA eines Organismus einzuschleusen. So ist es möglich, _ einzelne Gene des Fisches auszuschalten. Dabei können die Genetiker beobachten, welche Körperregionen, -funktionen oder Verhaltensweisen des Fisches, sein sogenannter Phänotyp, sich ändern. Die Funktionen und Aufgaben des betroffenen Gens werden auf diese Weise ermittelt.
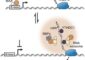
Das entwickelte Transposon wurde von den Wissenschaftlern mit einer „Protein-Falle“, dem „Protein-Trap“ ausgestattet. „Das getroffene Gen erzeugt bei Aktivität dann nicht mehr das ursprüngliche gesunde Protein, sondern ein unfunktionelles und verkürztes“, so Hans-Martin Pogoda, der an der Studie mitgearbeitet hat. „Daran ist nun als Marker das rot-fluoreszierende Protein RFP gekoppelt.“ Unter fluoreszierendem Licht lässt sich die zelluläre Verteilung des Fusionsproteins im Körper des Fisches sichtbar machen und ermitteln (siehe Bild). Das pGBT-RP2.1 Transposon wurde im Labor von Professor Stephen C. Ekker (Mayo Clinic) entwickelt. Die Kölner Genetiker testeten das System erstmals in großem Rahmen im Zebrafisch. „Wir konnten 350 durch pGBT-RP2.1 mutierte Abstammungslinien von Zebrafischen erzeugen und phäno- wie genotypisch charakterisieren“, erklärt Hans-Martin Pogoda.
Die neue Methode birgt große Vorteile. So lassen sich die mutierten Gene leichter identifizieren als bei herkömmlichen Mutageneseverfahren. Auch können die Wissenschaftler bei Ihrer Suche nach relevanten Phänotypen hinsichtlich bestimmter Körperregionen oder Verhaltensweisen eine gezielte Vorselektion treffen. Dank der Beschaffenheit des Transposons lassen sich die betroffenen Gene zudem zu bestimmten Zeiten und in bestimmten Gewebsstrukturen ausschalten. „Auf diese Weise lassen sich jetzt auch beim Zebrafisch Situationen erzeugen, die dem konditionellen Gen-Knock-Out der Maus vergleichbar sind“, sagt Pogoda. „Dies wurde bisher in keinem Modellorganismus außerhalb der Maus erreicht.“
Bei Rückfragen: Professor Matthias Hammerschmidt,
Tel.: 0221/470 5665, E-Mail: mhammers@uni-koeln.de
Dr. Hans-Martin Pogoda,
Tel.: 0221/470 2738, E-Mail: pogodah@uni-koeln.de
Neue Methode vereinfacht die funktionelle Genomforschung
Genetiker markieren durch springendes Gen mutierte Proteine mit fluoreszierendem Farbstoff
Ein neues Werkzeug erleichtert Genetikern ihre Arbeit: pGBT-RP2.1, das springende Genkonstrukt, färbt die von ihm inaktivierten Proteine rot. Dadurch hilft es bei der zielgenauen und systematischen Erforschung des Erbguts. Wichtig ist das neue und vielseitige Werkzeug für das Gebiet der funktionellen Genomforschung (functional genomics). Nun können in Zukunft z.B. Erbkrankheiten __ schneller dem dafür verantwortlichen Gen zugeordnet werden. Entwickelt wurde das Verfahren im Tiermodell Zebrafisch, es lässt sich aber auch bei anderen Modellorganismen einsetzen. Beteiligt an der Entwicklung des sogenannten Transposons waren Kölner Genetiker unter Leitung von Professor Matthias Hammerschmidt vom Institut für Entwicklungsbiologie und amerikanische Wissenschaftler von der Mayo Clinic und der Universität von Minnesota. Der Artikel „In vivo protein trapping produces a functional expression codex of the vertebrate proteome“ ist in der aktuellen Ausgabe von Nature Methods erschienen.
„Springende Gene“, sogenannte Transposons, sind frei bewegliche Teile der DNA. Sie werden von Genetikern dazu benutzt, fremde Gene in die DNA eines Organismus einzuschleusen. So ist es möglich, _ einzelne Gene des Fisches auszuschalten. Dabei können die Genetiker beobachten, welche Körperregionen, -funktionen oder Verhaltensweisen des Fisches, sein sogenannter Phänotyp, sich ändern. Die Funktionen und Aufgaben des betroffenen Gens werden auf diese Weise ermittelt.
Das entwickelte Transposon wurde von den Wissenschaftlern mit einer „Protein-Falle“, dem „Protein-Trap“ ausgestattet. „Das getroffene Gen erzeugt bei Aktivität dann nicht mehr das ursprüngliche gesunde Protein, sondern ein unfunktionelles und verkürztes“, so Hans-Martin Pogoda, der an der Studie mitgearbeitet hat. „Daran ist nun als Marker das rot-fluoreszierende Protein RFP gekoppelt.“ Unter fluoreszierendem Licht lässt sich die zelluläre Verteilung des Fusionsproteins im Körper des Fisches sichtbar machen und ermitteln (siehe Bild). Das pGBT-RP2.1 Transposon wurde im Labor von Professor Stephen C. Ekker (Mayo Clinic) entwickelt. Die Kölner Genetiker testeten das System erstmals in großem Rahmen im Zebrafisch. „Wir konnten 350 durch pGBT-RP2.1 mutierte Abstammungslinien von Zebrafischen erzeugen und phäno- wie genotypisch charakterisieren“, erklärt Hans-Martin Pogoda.
Die neue Methode birgt große Vorteile. So lassen sich die mutierten Gene leichter identifizieren als bei herkömmlichen Mutageneseverfahren. Auch können die Wissenschaftler bei Ihrer Suche nach relevanten Phänotypen hinsichtlich bestimmter Körperregionen oder Verhaltensweisen eine gezielte Vorselektion treffen. Dank der Beschaffenheit des Transposons lassen sich die betroffenen Gene zudem zu bestimmten Zeiten und in bestimmten Gewebsstrukturen ausschalten. „Auf diese Weise lassen sich jetzt auch beim Zebrafisch Situationen erzeugen, die dem konditionellen Gen-Knock-Out der Maus vergleichbar sind“, sagt Pogoda. „Dies wurde bisher in keinem Modellorganismus außerhalb der Maus erreicht.“
Bei Rückfragen:
Professor Matthias Hammerschmidt,
Tel.: 0221/470 5665, E-Mail: mhammers@uni-koeln.de
Dr. Hans-Martin Pogoda,
Tel.: 0221/470 2738, E-Mail: pogodah@uni-koeln.de
Media Contact
Weitere Informationen:
http://www.uni-koeln.deAlle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Neuartiges Material für nachhaltiges Bauen
Innovativer Werkstoff für eine energieeffiziente Architektur: Forschende des Karlsruher Instituts für Technologie (KIT) stellen in der aktuellen Ausgabe der Fachzeitschrift Nature Communications ein polymerbasiertes Material mit besonderen Eigenschaften vor. Das…

Neues Antibiotikum gegen Erreger der Flussblindheit und Lymphatischen Filariose
Prof. Achim Hoerauf, Direktor des Instituts für Medizinische Mikrobiologie, Immunologie und Parasitologie des Universitätsklinikums Bonn (UKB), und seinem Team ist es in Kollaboration mit der Abteilung Pharmazeutische Technologie und Biopharmazie…
Evolutionäre Genomik: Folgen biodiverser Fortpflanzungssysteme
Die Deutsche Forschungsgemeinschaft (DFG) fördert die Einrichtung eines neuen Graduiertenkollegs (GRK) in der Biologie an der Universität Göttingen. Das GRK mit dem Titel „Evolutionary Genomics: Consequences of Biodiverse Reproductive Systems…