Kleine Löcher in der Hülle lassen Bakterien dahinsiechen

Die Haut bildet eine Barriere zwischen dem Körper und der Umwelt. Zusätzlichen Schutz bietet die Immunabwehr über natürliche Antibiotika, die mögliche Krankheitserreger wie Bakterien oder Pilze abtöten können. Eins dieser Antibiotika ist Dermcidin, das in den Schweißdrüsen des Menschen hergestellt und auf der Haut gegen eine ganze Reihe von Mikroorganismen aktiv wird.
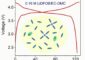
Nun hat ein Forscherteam unter Beteiligung des Universitätsklinikums Tübingen und des Max-Planck-Instituts für Entwicklungsbiologie ein Modell entworfen, wie Dermcidin seine antibiotische Wirkung im schwierigen Milieu des Schweißes entfaltet: Sein aktiver Bestandteil taucht in die Bakterienhülle ein und bildet dort winzige Löcher, die den Bakterien ein langsames, aber sicheres Ende bereiten.
Es ist an sich nicht ungewöhnlich, dass antibiotische Stoffe Mikroorganismen durch die Bildung von Poren in der äußeren Hülle töten. Viele der bekannten natürlichen Antibiotika auf der Körperoberfläche tragen eine positive Ladung und können sich besonders gut an die überwiegend negativ geladene Hüllmembran der Bakterien anlagern. Sie bilden dort Kanäle und zersetzen die Membran, schnell tritt der Tod der Bakterienzelle ein. Bei Dermcidin hatten Forscher bisher vermutet, dass der Wirkmechanismus prinzipiell anders aussehen müsste. Dermcidin wird auf der Haut gespalten und der aktive Bestandteil, DCD-1L genannt, ist insgesamt negativ geladen. Außerdem sterben die angegriffenen Bakterien langsam, sie können sogar noch Notfallprogramme in Gang setzen.
In einer neuen Studie hat ein Forscherteam dies noch einmal überprüft. Dazu gehörten Maren Paulmann, Ines Wanke und Birgit Schittek von der Universitäts-Hautklinik Tübingen und Forscher des Max-Planck-Instituts für Entwicklungsbiologie unter der Leitung von Dirk Linke und Michael Habeck, deren Zusammenarbeit über den an der Universität Tübingen angesiedelten Sonderforschungsbereich 766 „Die bakterielle Zellhülle“ zustande kam, sowie weitere Wissenschaftler vom Forschungszentrum Borstel und dem Karlsruher Institut für Technologie. Bei früheren Untersuchungen mit dem Elektronenmikroskop und speziellen Markierungsexperimenten hatte sich kein Hinweis auf Porenbildung in der Bakterienmembran durch DCD-1L gefunden. Erst elektrische Messungen an künstlichen Membranen ergaben nun, dass geladene Teilchen hindurchkommen, allerdings nur sehr langsam. Das ließ auf sehr kleine Löcher schließen.
Der Ablauf könnte den Ergebnissen der Forscher zufolge so aussehen: Das insgesamt negativ geladene DCD-1L-Molekül hat ein positiv geladenes Ende, das zur Anheftung an die Bakterienmembran ausreicht. Durch die Wechselwirkung mit der Membran im stark sauren und salzigen Schweißmilieu nimmt das bis dahin unstrukturierte DCD-1L eine Schraubenform an, und es können sich mehrere DCD-1L-Moleküle spontan zusammenlagern. Stabilisiert durch positiv geladene Zinkionen, die vermehrt im Schweiß vorkommen, bildet der DCD-1L-Komplex winzige Kanäle durch die Bakterienhülle. Sie sind so schmal, dass einzelne geladene Teilchen gerade hindurchpassen. Doch mit der Zeit bricht durch den Ladungsausgleich das Spannungsgefälle zwischen dem Innern des Bakteriums und der Umgebung zusammen, das die Grundlage für lebenswichtige Transport- und Stoffwechselprozesse bildet. Die Bakterienzelle stirbt.
Körpereigene Antibiotika wie DCD-1L aus Dermcidin werden auch im Zusammenhang mit der Hauterkrankung Neurodermitis erforscht. Diese Patienten leiden unter vermehrt auftretenden Hautinfektionen. Interessanterweise fanden sich bei diesen Patienten geringere Mengen an DCD-1L und verwandten Stoffen aus Dermcidin im Schweiß als bei Gesunden.
Originalpublikation:
Maren Paulmann, Thomas Arnold, Dirk Linke, Suat Özdirekcan, Annika Kopp, Thomas Gutsmann, Hubert Kalbacher, Ines Wanke, Verena J. Schuenemann, Michael Habeck, Jochen Bürck, Anne S. Ulrich, Birgit Schittek: Structure-activity analysis of the dermcidin-derived peptide DCD-1L, an anionic antimicrobial peptide present in human sweat. The Journal of Biological Chemistry, Band 287, Ausgabe 11, 8434-8443, 9. März 2012.
Ansprechpartner:
Prof. Birgit Schittek
Universitäts-Hautklinik Tübingen
Tel.: 07071 29-80832
E-Mail: birgit.schittek(at)med.uni-tuebingen.de
Dr. Dirk Linke
Max-Planck-Institut für Entwicklungsbiologie
Tel.: 07071 601- 357
E-Mail: dirk.linke(at)tuebingen.mpg.de
Media Contact
Alle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Diamantstaub leuchtet hell in Magnetresonanztomographie
Mögliche Alternative zum weit verbreiteten Kontrastmittel Gadolinium. Eine unerwartete Entdeckung machte eine Wissenschaftlerin des Max-Planck-Instituts für Intelligente Systeme in Stuttgart: Nanometerkleine Diamantpartikel, die eigentlich für einen ganz anderen Zweck bestimmt…
Neue Spule für 7-Tesla MRT | Kopf und Hals gleichzeitig darstellen
Die Magnetresonanztomographie (MRT) ermöglicht detaillierte Einblicke in den Körper. Vor allem die Ultrahochfeld-Bildgebung mit Magnetfeldstärken von 7 Tesla und höher macht feinste anatomische Strukturen und funktionelle Prozesse sichtbar. Doch alleine…

Hybrid-Energiespeichersystem für moderne Energienetze
Projekt HyFlow: Leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem für moderne Energienetze. In drei Jahren Forschungsarbeit hat das Konsortium des EU-Projekts HyFlow ein extrem leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem entwickelt, das einen…