Molekulare motorbetriebene Bio-Computer
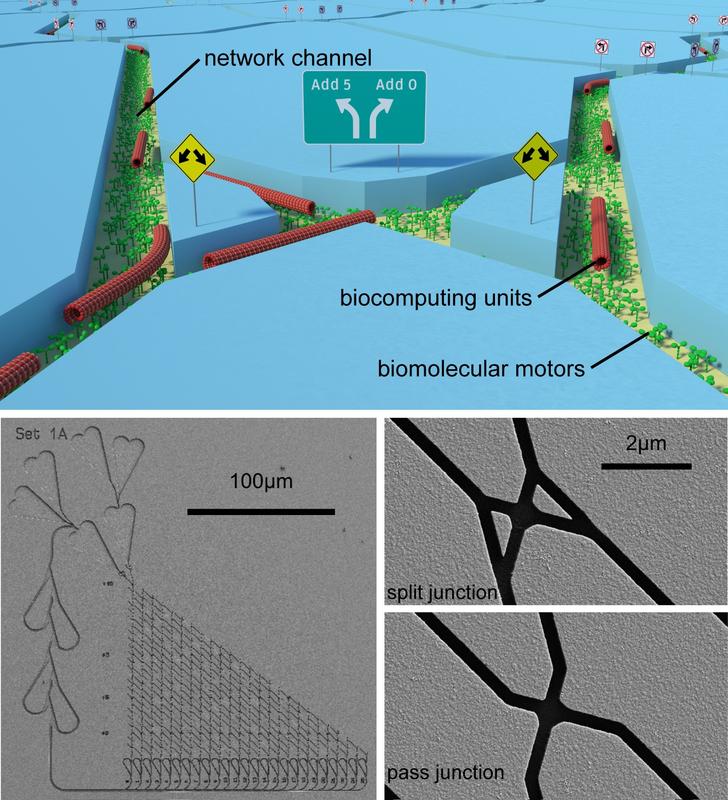
Netzwerkbasierte Bioinformatik. Till Korten, Cornelia Kowol
Fehler in Software oder Computerchips verursachen Abstürze von Computern oder Smartphones und ermöglichen Hackern das Stehlen von Passwörtern. Automatisierte Prüfverfahren könnten diese Probleme vermeiden. Leider steigt die dafür benötigte Rechenleistung exponentiell mit der Größe des Programmes an. Daher sind für herkömmliche Computer der Energieverbrauch, der Kühlungsbedarf und die benötigte Rechenleistung zu hoch, um große Programme zu überprüfen.
Ein vor kurzem gestartetes Forschungsprojekt hat die Entwicklung eines Bio-Computers zum Ziel, der zwei Hauptprobleme der Supercomputer von heute überwinden soll: Zum einen verbrauchen Supercomputer erhebliche Mengen elektrischen Stromes, so dass die Entwicklung mächtigerer Computer vor allem an der Kühlung der Prozessoren scheitert.
Zum anderen sind heutige Computer nicht besonders gut darin, mehrere Aufgaben gleichzeitig zu erledigen. Der Bio-Computer auf Basis von molekularen Motoren verbraucht im Vergleich zu herkömmlichen Computern nur einen Bruchteil der Energie pro Rechenoperation. Außerdem kann er sehr viele Operationen gleichzeitig ausführen und ist daher besonders für Probleme wie die Softwareüberprüfung geeignet, bei der sehr viele Lösungen überprüft werden müssen.
Der Einfluss dieser Forschung beschränkt sich nicht auf das Design fehlerfreier Software: „Praktisch betrachtet können alle wirklich interessanten mathematischen Probleme der heutigen Zeit mit unserer derzeitigen Computertechnologie nicht effizient berechnet werden“, so Dan V. Nicolau, Ph.D. M.D. vom britischen Unternehmen MolecularSense, das die initiale Idee zur Nutzung biomolekularer Motoren als Computer hatte.
Diese Grenze, ab der ein Problem zu schwierig für einen Computer wird, möchte das hier vorgestellte neue Projekt durch die Nutzung biomolekularer Motoren als Recheneinheiten hinausschieben: Die Grundidee ist, dass die – jeweils nur wenige Milliardstel eines Meters (Nanometers) kleinen – biomolekularen Maschinen Probleme lösen, indem sie sich durch ein Netzwerk winziger Kanäle bewegen. Das per Nanofabrikation hergestellte Netzwerk repräsentiert dabei einen mathematischen Algorithmus (siehe Bild 1). Dieser Ansatz wird von den Forschern als „netzwerk-basierter Bio-Computer“ bezeichnet.
Jedesmal, wenn die Biomoleküle eine Kreuzung im Netzwerk erreichen, können sie entscheiden, ob sie eine Zahl addieren oder nicht. Jedes einzelne Biomolekül fungiert so als ein winziger Computer – mit Prozessor und Arbeitsspeicher. Obwohl jedes Biomolekül für sich betrachtet viel langsamer rechnet als ein elektronischer Computer, kann die schiere Anzahl an Molekülen dank Selbstorganisation eine große Rechenleistung entwickeln. Dieser Ansatz funktioniert im kleinen Maßstab auch schon in der Praxis, wie die Forscher in einer Publikation im wissenschaftlichen Magazin PNAS zeigen konnten.
„Wir nutzen die molekularen Motoren von Zellen, die in Milliarden von Jahren der Evolution optimiert wurden, als hocheffiziente Nanomaschinen.” sagt Prof. Stefan Diez, der das teilnehmende Dresdner Forschungsteam leitet. „Die biologischen Recheneinheiten können sich selbst vermehren, um sich der Schwierigkeit des mathematischen Problems anzupassen”, ergänzt Dr. Till Korten von der TU Dresden, Mitkoordinator des Bio4Comp Projektes und gemeinsamer Erstautor der PNAS Publikation.
Das Forscherteam wird sich nun der Entwicklung der Technologie widmen, die zur Hochskalierung netzwerkbasierter Biocomputer benötigt wird. Dabei haben sich die Forscher sich zum Ziel gesetzt andere alternative Computer wie DNA-Computer oder Quantencomputer zu übertreffen.
Das Forscherteam hofft, damit eine größere Gemeinschaft aus Wissenschaft und Wirtschaft anzusprechen, und so ein neues Forschungsfeld zu begründen. Um dies zu realisieren, haben sie eine Forschungsförderung des Future & Emerging Technologies (FET) Programmes der EU in Höhe von 6,1 Millionen Euro erhalten. Das geförderte Projekt ist stark interdisziplinär und bewegt sich im Spannungsfeld zwischen Mathematik, Biologie, Ingenieurwissenschaft und Informatik.
1,1 Millionen Euro der Forschungsförderung gehen an die Gruppe von Stefan Diez, Professor für BioNanoTools am B CUBE, einem Forschungsinstitut der TU Dresden, das sich der Erforschung und Entwicklung biologischer Materialien widmet. Die Gruppe von Stefan Diez wird Motorproteine der Zelle (z. B. Kinesin-1) modifizieren, um sie für ihren Einsatz in Bio-Computern und anderen nanofabrizierten Geräten zu optimieren.
Dabei werden sie von einer engen Zusammenarbeit mit dem Center for Advancing Electronics Dresden (cfaed), einem der aktuellen Exzellenzcluster der TU Dresden, profitieren. „Indem wir Motorproteine optimieren, stellen wir nicht nur ideale Werkzeuge für die Nanotechnologie her – sondern lernen auch viel über ihre Funktionsweise und über ihr Verhalten innerhalb der Zelle”, so Diez. Diese Erkenntnisse werden auch über dieses Forschungsprojekt hinaus nützlich sein, da zum Beispiel auch die Rollen der Motorproteine bei Krankheiten wie Krebs oder Demenz untersucht werden können.
Das Bio4Comp Projekt (2017-2021) wird durch Horizont 2020 gefördert, das EU-Rahmenprogramm für Forschung und Innovation (Zuschussvereinbarung Nr. 732482). Weitere Informationen finden Sie unter http://www.bio4comp.eu.
Informationen für Journalisten:
Stefan Diez, Professor für BioNanoTools
B CUBE – Center for Molecular Bioengineering
Technische Universität Dresden, Dresden, Germany
Tel.: +49 (0) 351 463-43010
stefan.diez@tu-dresden.de
http://www.tu-dresden.de/bcube
Kontaktliste Projektpartner:
Partner 1: Lund University, Lund, Schweden
Heiner Linke, Professor für Nanophysik; Direktor NanoLund
Tel.: +46 (0) 46 222 4245
heiner.linke@ftf.lth.se
Kristina Lindgärde Pressansvarig vid Kommunikation och Samverkan, LTH
Tel.: +46 (0) 46 222 0769
kristina.lindgarde@kansli.lth.se
http://www.nano.lu.se/
Partner 2: Linné-University Kalmar, Kalmar, Schweden
Alf Månsson, professor i fysiologi
Tel.: +46 (0) 70 886 62 43
Annika Sand, pressansvarig
Tel.: +46 (0) 76 830 01 05
https://lnu.se/en/research/searchresearch/the-molecular-motor-and-bionano-group/
Partner 3: Molecular Sense Ltd., Oxford, U.K.
Dan V. Nicolau, PhD. MD.
https://molecularsense.com/
Partner 4: Bar-Ilan University, Ramat Gan, Israel
Dr. Hillel Kugler
Tel.: +972 (0) 3 7384437
kugler.hillel@biu.ac.il
http://www.eng.biu.ac.il/hillelk/
Partner 5: Fraunhofer-Gesellschaft zur Förderung der angewandten Forschung e.V.Prof. Dr. Stefan E. Schulz
Stellvertretender Direktor des Fraunhofer ENAS, Leiter des Geschäftsfelds Mikro- und Nanoelektronik, Bio4Comp-Projektleiter in der Fraunhofer-Gesellschaft
Telefon: +49 (0) 371 45001-232
stefan.schulz@enas.fraunhofer.de
Dr. Martina Vogel
Leiterin Marketing & Public Relations, Fraunhofer ENAS, Chemnitz
Tel.: +49 (0) 371 45001-203
martina.vogel@enas.frauhnofer.de
Marie-Luise Righi
Leiterin Marketing & Kommunikation, Fraunhofer ISC, Würzburg
Tel.: +49 (0) 931 4100-150
marie-luise.righi@isc.fraunhofer.de
https://www.fraunhofer.de/
To PNAS paper: http://www.pnas.org/content/113/10/2591.full?sid=5d9e45c4-6338-461e-9c93-a74c5ca…
To web-site:http://www.bio4comp.eu
Media Contact
Alle Nachrichten aus der Kategorie: Informationstechnologie
Neuerungen und Entwicklungen auf den Gebieten der Informations- und Datenverarbeitung sowie der dafür benötigten Hardware finden Sie hier zusammengefasst.
Unter anderem erhalten Sie Informationen aus den Teilbereichen: IT-Dienstleistungen, IT-Architektur, IT-Management und Telekommunikation.
Neueste Beiträge

Diamantstaub leuchtet hell in Magnetresonanztomographie
Mögliche Alternative zum weit verbreiteten Kontrastmittel Gadolinium. Eine unerwartete Entdeckung machte eine Wissenschaftlerin des Max-Planck-Instituts für Intelligente Systeme in Stuttgart: Nanometerkleine Diamantpartikel, die eigentlich für einen ganz anderen Zweck bestimmt…
Neue Spule für 7-Tesla MRT | Kopf und Hals gleichzeitig darstellen
Die Magnetresonanztomographie (MRT) ermöglicht detaillierte Einblicke in den Körper. Vor allem die Ultrahochfeld-Bildgebung mit Magnetfeldstärken von 7 Tesla und höher macht feinste anatomische Strukturen und funktionelle Prozesse sichtbar. Doch alleine…

Hybrid-Energiespeichersystem für moderne Energienetze
Projekt HyFlow: Leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem für moderne Energienetze. In drei Jahren Forschungsarbeit hat das Konsortium des EU-Projekts HyFlow ein extrem leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem entwickelt, das einen…





















