Narben der Entwicklung
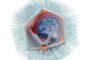
LINNAEUS macht es möglich, die Herkunft jeder Zelle eines Zebrafischs zu verfolgen. Mikroskopische Aufnahme: AG Junker, MDC
Dass Zellen die Bausteine des Lebens sind, steht in jedem Bio-Lehrbuch. Ihre Vielfalt offenbart sich jedoch erst jetzt. Denn nun zeigen Technologien wie die RNA-Sequenzierung, welche Gene in jeder einzelnen Zelle abgelesen werden. Anhand ähnlicher Expressionsprofile kann man sie systematisch ordnen.
„Wann immer wir uns ein Organ oder einen Organismus damit ansehen, finden wir nicht nur bekannte, sondern auch unbekannte und seltenere Zelltypen“, sagt Dr. Jan Philipp Junker, Leiter der Arbeitsgruppe „Quantitative Entwicklungsbiologie“ am Berlin Institute for Medical Systems Biology des Max-Delbrück-Centrums für Molekulare Medizin in der Helmholtz-Gemeinschaft (MDC).
„Die nächste Frage liegt nahe: Woher kommen die verschiedenen Zelltypen?“ Das Team um Junker stellt nun im Fachjournal „Nature Biotechnology“ eine Technik namens LINNAEUS vor, mit der sie sowohl den Zelltyp als auch den Ursprung jeder Zelle analysieren können.
„Wir wollen die Flexibilität in der Entwicklung von Organismen verstehen“, sagt Junker. Passiert während der embryonalen Entwicklung eine Störung, zum Beispiel durch eine Mutation oder durch Umwelteinflüsse, sorgen Reparaturmechanismen mitunter dafür, dass ein Lebewesen später gesund erscheint.
Nur die Herkunft der einzelnen Zellen verrät dann die wahre Geschichte – das Ausmaß der Störung sowie den Reparaturmechanismus. Selbst das Herz eines erwachsenen Zebrafischs kann sich nach einer Verletzung regenerieren.
„Wiederholt sich dabei ein entwicklungsbiologisches Programm oder passiert etwas Neues? Wandeln sich Zellen und übernehmen andere Aufgaben?“, fragt Junker. In anderen Fällen fehlt ein Zelltyp und verursacht damit eine Erkrankung. Aus den Stammbäumen aller Zellen, die mit LINNAEUS möglich werden, können Forscherinnen und Forscher künftig neue Hypothesen zu solchen Fragen ableiten.
Durch das schnelle Flicken entstehen zufällige Narben
Die Technik beruht auf Narben im Erbgut, deren Kombination wie ein Barcode für die Herkunft jeder Zelle funktioniert. Noch während die Zebrafisch-Embryonen im Ein-Zell-Stadium sind, injiziert Junkers Team die Gen-Schere CRISPR-Cas9. Innerhalb der nächsten acht Stunden zerschneidet die Schere immer wieder eine Sequenz, die der Fisch garantiert nie braucht: das Gen für ein rot fluoreszierendes Protein (RFP). Das rote Leuchten des Embryos nimmt ab, die Wunden im Erbgut schließen derweil Tausende verschiedener Narben.
„CRISPR schneidet zwar immer an genau einer Stelle. Aber die Zellen haben für die Reparatur höchstens eine Viertelstunde Zeit bis zur nächsten Zellteilung“, sagt Junker. „Also wird schnell geflickt, die Chromosomenstücke irgendwie zusammengeklebt. Dabei passieren Fehler. Die Narben im Erbgut haben eine zufällige Länge, auch die exakte Position ist variabel.“ Bei der Zellteilung werden die genetischen Narben an die Tochterzellen vererbt. Zellen, die auf einen gemeinsamen Vorfahren zurückgehen, können somit anhand ihrer genetischen Narben erkannt werden.
Während die Einzelzell-RNA-Sequenzierung Tausende Zellen in Zelltypen ordnet, zeigen die Narben Millionen Verbindungen zwischen den Zellen. Die Herausforderungen, aus diesem Wollknäuel von Daten Stammbäume zu rekonstruieren, waren vielfältig. Zum einen sind manche Narben besonders wahrscheinlich.
„Das ist gefährlich. Denn wenn die gleiche Narben-Sequenz in einer Zelle im Herz und im Gehirn geschaffen wird, könnte man fälschlicherweise davon ausgehen, dass sie einen gemeinsamen Vorfahren haben“, sagt Junker. „Wir mussten also wissen, welchen Sequenzen wir nicht trauen können und sie aussortieren.“ Zum anderen seien nicht immer alle Narben in einer Zelle auffindbar, sagt Bioinformatiker Bastiaan Spanjaard, einer der Erstautoren der Studie. „Wir haben also eine Methode entwickelt, die beim Bauen der Stammbäume die fehlenden Daten überbrücken kann.“
In den Datensatz hineinzoomen
Das Ergebnis sind Stammbäume, an deren Verzweigungen jeweils bunte Kuchendiagramme stehen. Jede Verzweigung ist eine Narbe und jede Farbe in den Kuchendiagrammen zeigt, in welchen Zelltypen sie vorkommt. In diese kondensierte Darstellung eines riesigen Datensatzes können Forscherinnen und Forscher so tief hineinzoomen wie sie möchten.
„Im Herzen gibt es zum Beispiel zwei Zelltypen, die kaum unterscheidbar sind. Aber die Stammbäume zeigen, dass sich ihre Entwicklung sehr früh trennt“, sagt Junker. „Wir wollen als nächstes schauen, wo sie im Fischherz vorkommen. Das gibt oft einen ersten Hinweis auf ihre Funktion.“ Sein Labor bleibt bei den Zebrafischen als Modell. Besonderes Potenzial sieht Junker jedoch auch darin, die Technik bei menschlichen Organoiden anzuwenden. Schließlich könne man dort verstehen, welche Mutation eines Patienten die Zellstammbäume nachhaltig stört.
Literatur
Bastiaan Spanjaard et al (2018): „Simultaneous lineage tracing and cell-type identification using CRISPR–Cas9-induced genetic scars“, Nature Biotechnology. Advance Online Publication 09.04.2018, doi:10.1038/nbt.4124.
Abstract: A key goal of developmental biology is to understand how a single cell is transformed into a full-grown organism comprising many different cell types. Single-cell RNA-sequencing (scRNA-seq) is commonly used to identify cell types in a tissue or organ. However, organizing the resulting taxonomy of cell types into lineage trees to understand the developmental origin of cells remains challenging. Here we present LINNAEUS (lineage tracing by nuclease-activated editing of ubiquitous sequences) – a strategy for simultaneous lineage tracing and transcriptome profiling in thousands of single cells. By combining scRNA-seq with computational analysis of lineage barcodes, generated by genome editing of transgenic reporter genes, we reconstruct developmental lineage trees in zebrafish larvae, and in heart, liver, pancreas, and telencephalon of adult fish. LINNAEUS provides a systematic approach for tracing the origin of novel cell types, or known cell types under different conditions.
Über das Max-Delbrück-Centrum für Molekulare Medizin (MDC)
Das Max-Delbrück-Centrum für Molekulare Medizin in der Helmholtz-Gemeinschaft (MDC) wurde 1992 in Berlin gegründet. Es ist nach dem deutsch-amerikanischen Physiker Max Delbrück benannt, dem 1969 der Nobelpreis für Physiologie und Medizin verliehen wurde. Aufgabe des MDC ist die Erforschung molekularer Mechanismen, um die Ursachen von Krankheiten zu verstehen und sie besser zu diagnostizieren, verhüten und wirksam bekämpfen zu können. Dabei kooperiert das MDC mit der Charité – Universitätsmedizin Berlin und dem Berlin Institute of Health (BIH) sowie mit nationalen Partnern, z.B. dem Deutschen Zentrum für Herz-Kreislauf-Forschung (DHZK), und zahlreichen internationalen Forschungseinrichtungen. Am MDC arbeiten mehr als 1.600 Beschäftigte und Gäste aus nahezu 60 Ländern; davon sind fast 1.300 in der Wissenschaft tätig. Es wird zu 90 Prozent vom Bundesministerium für Bildung und Forschung und zu 10 Prozent vom Land Berlin finanziert und ist Mitglied in der Helmholtz-Gemeinschaft deutscher Forschungszentren.
Media Contact
Alle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Diamantstaub leuchtet hell in Magnetresonanztomographie
Mögliche Alternative zum weit verbreiteten Kontrastmittel Gadolinium. Eine unerwartete Entdeckung machte eine Wissenschaftlerin des Max-Planck-Instituts für Intelligente Systeme in Stuttgart: Nanometerkleine Diamantpartikel, die eigentlich für einen ganz anderen Zweck bestimmt…
Neue Spule für 7-Tesla MRT | Kopf und Hals gleichzeitig darstellen
Die Magnetresonanztomographie (MRT) ermöglicht detaillierte Einblicke in den Körper. Vor allem die Ultrahochfeld-Bildgebung mit Magnetfeldstärken von 7 Tesla und höher macht feinste anatomische Strukturen und funktionelle Prozesse sichtbar. Doch alleine…

Hybrid-Energiespeichersystem für moderne Energienetze
Projekt HyFlow: Leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem für moderne Energienetze. In drei Jahren Forschungsarbeit hat das Konsortium des EU-Projekts HyFlow ein extrem leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem entwickelt, das einen…