Wie Bakterien sauerstoffabhängig die Bewegungsrichtung ändern: Molekulare Interaktionen

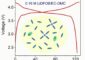
Strukturelle Änderungen in HemAT: Wenn Sauerstoff an die sensorische Domäne bindet (aus methodischen Gründen wurde das Experiment mit Kohlenmonoxid, CO, anstatt mit Sauerstoff durchgeführt), ändert sich die Proteinstruktur in der Umgebung der sensorischen Domäne. Dadurch verschieben sich Helix B und G. Das wiederum beeinflusst die benachbarte H-Helix, die sich bis in die Signal-Domäne fortsetzt. Abbildung: Samir El-Mashtoly<br>
Wie genau einzellige Organismen wie Bakterien es schaffen, auf ihre Umwelt zu reagieren, ist nicht vollkommen verstanden. Neue Erkenntnisse hat Dr. Samir El-Mashtoly vom RUB-Lehrstuhl Biophysik, geleitet von Prof. Dr. Klaus Gerwert, zusammen mit Kollegen aus Japan gewonnen.
Im Journal of Biological Chemistry beschreiben die Forscher die molekularen Interaktionen im Bakterium Bacillus subtilis während der Aerotaxis, also der Änderung in der Bewegungsrichtung abhängig von der Sauerstoffkonzentration in der Umgebung. Das Team untersuchte strukturelle Änderungen des Proteins HemAT. Über eine Signalkette sendet es Kommandos an den Motor der Schwimmgeißel des Bakteriums.
Signalübertragungskette
Zunächst bindet Sauerstoff an die Häm-Gruppe von HemAT; diese Gruppe ist auch aus dem Hämoglobin roter Blutkörperchen bekannt und wird als „sensorische Domäne“ von HemAT bezeichnet. Durch die Bindung ändert sich die Struktur der sensorischen Domäne. Das löst weitere strukturelle Veränderungen in HemAT aus, die letztendlich die Signal-Domäne des Proteins erreichen. Die Signal-Domäne überträgt dann die Information über die steigende Sauerstoffkonzentration an weitere Proteine in der Zelle. Diese Proteine leiten die Botschaft an den Motor der Schwimmgeißel weiter. Das Forscherteam untersuchte, wie die Information innerhalb von HemAT von der sensorischen Domäne zur Signal-Domäne wandert.
Proteinhelices übermitteln die Botschaft
Zu diesem Zweck nutzte Dr. El-Mashtoly die Einrichtung für zeitaufgelöste Ultraviolett-Resonanz-Raman-Spektroskopie am Picobiology Institute in Japan. Mit dieser Methode lassen sich zum Beispiel strukturelle Informationen über das Protein und Interaktionen von Wasserstoffbrückenbindungen verfolgen – auf einer Nano- bis Mikrosekunden-Zeitskala. Die Ergebnisse legen nahe, dass die strukturellen Änderungen in der sensorischen Domäne, also der Häm-Gruppe, zwei Proteinhelices in HemAT verschieben. Diese Verschiebung beeinflusst eine weitere Helix, die bis in die Signal-Domäne hineinragt. Über eine Serie von strukturellen Änderungen erreicht also die Information über die Sauerstoffbindung die Signal-Domäne.
Titelaufnahme
S. El-Mashtoly, M. Kubo, Y. Gu, H. Sawai, S. Nakashima, T. Ogura, S. Aono, T. Kitagawa (2012): Site-specific protein dynamics in communication pathway from sensor to signaling domain of oxygen sensor protein, HemAT-Bs, Journal of Biological Chemistry, doi: 10.1074/jbc.M112.357855
Weitere Informationen
Dr. Samir El-Mashtoly, Lehrstuhl Biophysik, Fakultät für Biologie und Biotechnologie der Ruhr-Universität, 44780 Bochum, Tel. 0234/32-29833
samir.elmashtoly@bph.rub.de
Angeklickt
Biophysik an der RUB
http://www.bph.rub.de/
Redaktion: Dr. Julia Weiler
Media Contact
Alle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Diamantstaub leuchtet hell in Magnetresonanztomographie
Mögliche Alternative zum weit verbreiteten Kontrastmittel Gadolinium. Eine unerwartete Entdeckung machte eine Wissenschaftlerin des Max-Planck-Instituts für Intelligente Systeme in Stuttgart: Nanometerkleine Diamantpartikel, die eigentlich für einen ganz anderen Zweck bestimmt…
Neue Spule für 7-Tesla MRT | Kopf und Hals gleichzeitig darstellen
Die Magnetresonanztomographie (MRT) ermöglicht detaillierte Einblicke in den Körper. Vor allem die Ultrahochfeld-Bildgebung mit Magnetfeldstärken von 7 Tesla und höher macht feinste anatomische Strukturen und funktionelle Prozesse sichtbar. Doch alleine…

Hybrid-Energiespeichersystem für moderne Energienetze
Projekt HyFlow: Leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem für moderne Energienetze. In drei Jahren Forschungsarbeit hat das Konsortium des EU-Projekts HyFlow ein extrem leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem entwickelt, das einen…