Leukämie: Künstliche Intelligenz unterstützt bei der Diagnostik
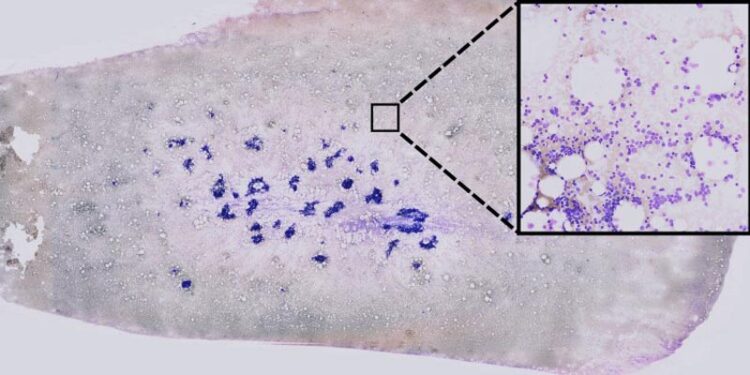
KI-basierte Verarbeitung von Knochenmarkausstrichen zur Unterstützung der Blutkrebsdiagnostik. Aus extrem hochaufgelösten Bilddaten (links) werden mittels sogenannter unüberwachter Lernverfahren Einzelzellbilder (Mitte) extrahiert. Anschließend werden diese Zellbilder unter Verwendung von neuronalen Netzen auf visuelle Auffälligkeiten, welche einen genetischen Ursprung haben könnten, untersucht. Bereiche, die für die Entscheidung des neuronalen Netzwerkes sehr wichtig sind, wurden mit Hilfe einer Methode für sogenannte Explainable AI farblich hervorgehoben (rechts).
© AG Risse
Informatiker und Mediziner entwickeln neues Verfahren zur Erkennung genetischer Veränderungen.
Therapeutische Entscheidungen bei Leukämie-Patienten basieren unter anderem auf einer Reihe bestimmter genetischer Merkmale der Erkrankung. Informatiker und Mediziner der Universität Münster haben nun eine Studie veröffentlicht, in der mittels eines KI-Verfahrens genetische Merkmale aus hochauflösenden mikroskopischen Aufnahmen von Knochenmarkausstrichen vorhergesagt werden können. Die Ergebnisse sind in der Fachzeitschrift „Blood Advances“ veröffentlicht.
Therapeutische Entscheidungen bei Patienten mit Akuter Myeloischer Leukämie (AML) – einer sehr aggressiven Form des Blutkrebses – basieren unter anderem auf einer Reihe bestimmter genetischer Merkmale der Erkrankung. Zum Zeitpunkt der Diagnose liegen diese Informationen jedoch nicht vor. Der Nachweis dieser genetischen Anomalien ist jedoch von entscheidender Bedeutung für eine frühzeitige und gezielte Behandlung der Patienten. Da Gentests teuer und zeitaufwendig sind, besteht nach wie vor ein großer Bedarf an kostengünstigen, schnellen und allgemein zugänglichen Tests zur Vorhersage dieser Anomalien.
Ein Team von Informatikern und Medizinern der Universität Münster hat nun eine Studie veröffentlicht, in der mittels eines Verfahrens, das auf Künstlicher Intelligenz (KI) basiert, diverse genetische Merkmale aus hochauflösenden mikroskopischen Aufnahmen von Knochenmarkausstrichen vorhergesagt werden können. Dadurch könnten künftig bereits am Tag der Diagnose gezielte Therapieentscheidungen getroffen werden, ohne auf genetische Analysen warten zu müssen. Die Ergebnisse sind in der Fachzeitschrift „Blood Advances“ veröffentlicht.

KI-basierte Verarbeitung von Knochenmarkausstrichen zur Unterstützung der Blutkrebsdiagnostik. AG Risse
Bei diesem neuen Verfahren wurden die genetischen Veränderungen direkt aus extrem hochaufgelösten Multi-Gigabyte-Scans von ganzen Knochenmarkausstrichen von mehr als 400 AML-Patienten extrahiert. Ausgehend von dieser riesigen Datengrundlage konnten mehr als zwei Millionen einzelne Zellbilder erstellt werden. „Wir haben ein neuartiges und vollautomatisches Verfahren entwickelt, das mittels der Technik des maschinellen Lernens für eine komplexe Aufgabe trainiert wurde. In unserem Fall kann der zugrunde liegende Algorithmus die genetischen Merkmale und sehr feinen Muster in den großen histologischen Aufnahmen selbstständig erkennen“, erklärt Prof. Dr. Benjamin Risse, der die algorithmischen Entwicklungen aus Sicht der Informatik leitete. Das Verfahren filterte anschließend die Einzelzellbilder in Kategorien von verschiedenen Zelltypen und zeigte zudem genetische Abweichungen auf. Besonders interessant sei dabei, dass es dem menschlichen Betrachter nicht möglich sei, einige dieser Muster zu erkennen. Das liege zum Beispiel daran, dass sie zu schwach ausgeprägt sein können oder extrem feine Texturen betreffen, die dem menschlichen Auge trotz exzellenter Bildgebung verborgen bleiben.
Ein zentraler Vorteil des Verfahrens liegt in der aufeinander aufbauenden KI-Pipeline, die eine Kontrolle der (Zwischen-) Ergebnisse ermöglicht und die für maschinelles Lernen oft benötigten manuellen Vorarbeiten auf ein Minimum reduziert. Ermöglicht wird dies durch eine Kombination von sogenannten unüberwachten, selbst-überwachten und überwachten Lernverfahren. Insbesondere die beiden erstgenannten Verfahren benötigen zum Beispiel keine manuelle Datenselektion, sondern versuchen, relevante Inhalte selbstständig aus den Bilddaten zu gewinnen.
„Mithilfe eines sogenannten inkrementellen Vorgehens haben wir Zwischenschritte zur Überprüfung der Bilder durch einen menschlichen Experten durchgeführt. Das ist zum Beispiel bei problematisch eingestuften Zellbildern nötig“, sagt Dr. Linus Angenendt, Leiter der Arbeitsgruppe Personalisierte Krebstherapie und Digitale Medizin am Universitätsklinikum Münster. Problematische Zellbilder können zum Beispiel durch fehlerhafte Färbungen auftreten. Das trainierte Modell wurde anschließend auf einem unabhängigen Datensatz von weiteren 70 Patienten mit über 440.000 Einzelzellbildern evaluiert – quasi als Testkohorte.
Zwar kann das neue Verfahren die genetischen Analysen nicht ersetzen. Die Methode trägt dennoch dazu bei, sehr früh eine Idee davon zu bekommen, welche genetischen Veränderungen der Leukämie zugrunde liegen könnten. Das würde vor allem bei besonders aggressiven Erkrankungen helfen, bei denen man nicht auf die kompletten genetischen Analysen warten kann.
Die Wissenschaftler sind davon überzeugt, dass digitale Verfahren und künstliche Intelligenz an großen medizinischen Datensätzen immer wichtiger werden, wenn es um personalisierte Therapieempfehlungen für Patienten mit bösartigen Erkrankungen geht. Diese Studie liefert dazu eine wichtige Grundlage, beispielsweise für die Entwicklung ähnlicher Ansätze für andere Knochenmarkserkrankungen.
Die Arbeit erhielt finanzielle Unterstützung der Europäischen Union und der Deutschen Forschungsgemeinschaft.
Originalpublikation
Kockwelp, J., et al (2023). Deep Learning Predicts Therapy-Relevant Genetics in Acute Myeloid Leukemia from Pappenheim-stained Bone Marrow Smears. Blood Advances. Doi: 10.1182/bloodadvances.2023011076
Wissenschaftliche Ansprechpartner:
Prof. Dr. Benjamin Risse
Institut für Geoinformatik
Universität Münster
Telefon +49 251 83-32717
E-Mail: b.risse@uni-muenster.de
Originalpublikation:
Media Contact
Alle Nachrichten aus der Kategorie: Verfahrenstechnologie
Dieses Fachgebiet umfasst wissenschaftliche Verfahren zur Änderung von Stoffeigenschaften (Zerkleinern, Kühlen, etc.), Stoffzusammensetzungen (Filtration, Destillation, etc.) und Stoffarten (Oxidation, Hydrierung, etc.).
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Trenntechnologie, Lasertechnologie, Messtechnik, Robotertechnik, Prüftechnik, Beschichtungsverfahren und Analyseverfahren.
Neueste Beiträge

Neues Wirkprinzip gegen Tuberkulose
Gemeinsam ist es Forschenden der Heinrich-Heine-Universität Düsseldorf (HHU) und der Universität Duisburg-Essen (UDE) gelungen, eine Gruppe von Molekülen zu identifizieren und zu synthetisieren, die auf neue Art und Weise gegen…

Gefahr durch Weltraumschrott
Neue Ausgabe von „Physikkonkret“ beleuchtet Herausforderungen und Lösungen für eine nachhaltige Nutzung des Weltraums. Die Deutsche Physikalische Gesellschaft (DPG) veröffentlicht eine neue Ausgabe ihrer Publikationsreihe „Physikkonkret“ mit dem Titel „Weltraumschrott:…

Wasserstoff: Versuchsanlage macht Elektrolyseur und Wärmepumpe gemeinsam effizient
Die nachhaltige Energiewirtschaft wartet auf den grünen Wasserstoff. Neben Importen braucht es auch effiziente, also kostengünstige heimische Elektrolyseure, die aus grünem Strom Wasserstoff erzeugen und die Nebenprodukte Sauerstoff und Wärme…





















