Innovative Methode: Forscher der TU Kaiserslautern finden neues DNA-Reparatur-Protein
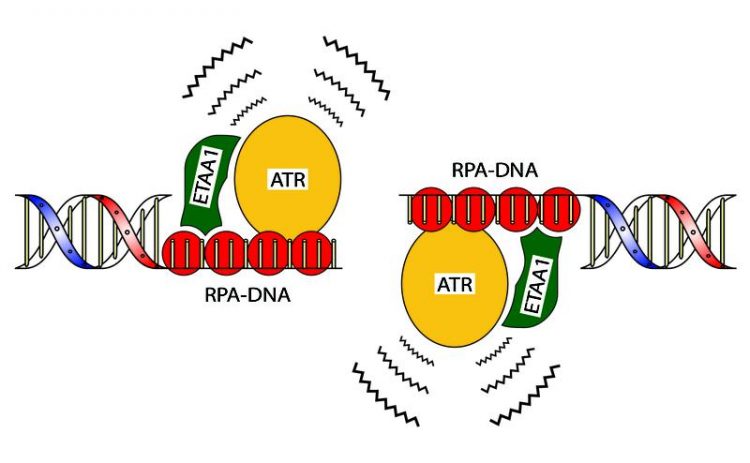
Die Abbildung zeigt die Wirkweise des neu identifizierten Proteins. Es verbindet die ATR-Kinase mit einem Protein namens RPA, das einzelsträngige DNA aufspürt. So können DNA-Schäden behoben werden. Räschle
Teilt sich eine Zelle, gibt sie ihr genetisches Material zu gleichen Teilen an ihre beiden Tochterzellen weiter. Die DNA muss sich dazu zunächst verdoppeln. Dabei sorgt eine Vielzahl von Proteinen dafür, dass alles in geregelten Bahnen verläuft. Zahlreiche Reparaturmechanismen stellen sicher, dass DNA-Schäden während der Verdopplung des Erbgutes sofort entfernt werden. Kommt es hierbei zu Fehlern, können Krebserkrankungen die Folge sein.
„Ein Beispiel ist der genetisch veranlagte Brustkrebs, bei dem ein solcher Reparaturfaktor inaktiviert ist“, sagt Dr. Markus Räschle, der am Lehrstuhl für Molekulare Genetik bei Professorin Dr. Zuzana Storchová und am Lehrstuhl für Biotechnologie und Systembiologie bei Professor Dr. Michael Schroda an der TU Kaiserslautern forscht. „Wir möchten herausfinden, wie viele und welche Proteine an den komplexen Mechanismen der DNA-Reparatur beteiligt sind.“
Dazu erforschen Räschle und seine Kollegen das sogenannte Proteom, die Gesamtheit aller Proteine einer Zelle. Experten sprechen bei diesem Forschungszweig von Proteomik. „Mit Hilfe der Massenspektrometrie ermitteln wir die genaue Menge und Masse von Molekülen“, erklärt der Forscher. „Damit können wir Tausende von Proteinen einer Probe exakt bestimmen.“
In den vergangenen Jahren hat Räschle im Labor von Professor Dr. Matthias Mann – einem der führenden Experten für Proteomik – am Max-Planck-Institut für Biochemie in Martinsried ein Verfahren entwickelt, welches erlaubt, alle Proteine eines beliebigen DNA-Reparaturprozesses gesamtheitlich zu analysieren.
„Mit herkömmlichen Techniken konnten wir bislang nur eine beschränkte Anzahl bekannter Reparaturfaktoren analysieren. Nun können wir auch neue Moleküle entdecken und anschließend untersuchen, wie sie untereinander wechselwirken“, so Räschle weiter, der diese Technik mit seinen Kollegen im letzten Jahr in der Fachzeitschrift „Science“ vorgestellt hat.
In der aktuellen Studie untersuchen sie mit diesem neuen Ansatz, welche Proteine bei der Reparatur von DNA-Doppelstrangbrüchen beteiligt sind. Neben vielen bekannten Reparaturfaktoren haben sie so auch ein bislang wenig charakterisiertes Protein entdeckt, das sogenannte Ewing Tumor-Associated Antigen 1 (ETAA1).
Zusammen mit Kollegen vom Zentrum für Proteinforschung der Universität Kopenhagen um Professor Niels Mailand und Peter Haahr, dem Erstautor der aktuellen Studie, zeigen sie, wie dieses Protein zur Reparatur von DNA-Doppelstrangbrüchen beiträgt. „Es löst bei Fehlern ein Alarmsignal aus und sorgt so dafür, dass Schäden behoben werden“, erklärt Räschle. „Es bindet und aktiviert ein weiteres Protein, die ATR-Kinase, und hilft so, das Signal an andere Reparaturfaktoren in der Zelle weiterzuleiten.“
Erst im vergangenen Jahr haben Wissenschaftler aus Houston entdeckt, dass Mutationen im ETAA1-Gen mit einem erhöhten Pankreaskrebsrisiko einhergehen. „Die Erforschung der DNA-Reparaturprozesse auf molekularer Ebene kann uns so helfen“, so Räschle weiter, „die Ursachen verschiedener Krebs-Erkrankungen besser zu verstehen.“
Die Studie wurde in der renommierten Fachzeitschrift „Nature Cell Biology“ publiziert: „Activation of the ATR kinase by the RPA-binding protein ETAA1“
DOI: 10.1038/ncb3422
Die Studie wurde vom Center For Integrated Protein Science Munich (CIPSM) finanziell unterstützt.
Fragen beantwortet:
Dr. Markus Räschle
Molekulare Genetik
Tel.: 0631 205-5919
E-Mail: markus.raeschle@biologie.uni-kl.de
Media Contact
Weitere Informationen:
http://www.uni-kl.deAlle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Diamantstaub leuchtet hell in Magnetresonanztomographie
Mögliche Alternative zum weit verbreiteten Kontrastmittel Gadolinium. Eine unerwartete Entdeckung machte eine Wissenschaftlerin des Max-Planck-Instituts für Intelligente Systeme in Stuttgart: Nanometerkleine Diamantpartikel, die eigentlich für einen ganz anderen Zweck bestimmt…
Neue Spule für 7-Tesla MRT | Kopf und Hals gleichzeitig darstellen
Die Magnetresonanztomographie (MRT) ermöglicht detaillierte Einblicke in den Körper. Vor allem die Ultrahochfeld-Bildgebung mit Magnetfeldstärken von 7 Tesla und höher macht feinste anatomische Strukturen und funktionelle Prozesse sichtbar. Doch alleine…

Hybrid-Energiespeichersystem für moderne Energienetze
Projekt HyFlow: Leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem für moderne Energienetze. In drei Jahren Forschungsarbeit hat das Konsortium des EU-Projekts HyFlow ein extrem leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem entwickelt, das einen…