Einblicke in verborgene Proteinzustände
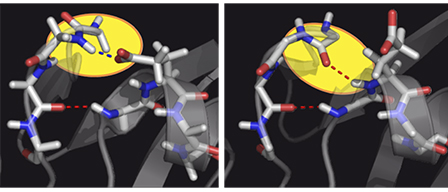
Der "Flip" zwischen zwei Zuständen des Proteins Ubiquitin, bei dem sich ein Teil des Proteins umlagert und über eine Wasserstoffbrücke an die geschwungene Helix des Proteins bindet (rechts). Der Zustand besteht für eine halbe Millisekunde und liegt nur zu etwa 10 Prozent in der Proteinprobe vor. Quelle: Paul Schanda/IBS Grenoble
Proteine erfüllen in allen Organismen lebenswichtige Aufgaben. Häufig verändern sie dabei ihre dreidimensionale Struktur, um mit verschiedenen Bindungspartnern in Aktion zu treten. Ein deutsch-französisches Forscherteam mit Jülicher Wissenschaftlern hat nun eine Methode entwickelt, die solche dynamischen Vorgänge erstmals bei größeren und unlöslichen Proteinen in atomarer Genauigkeit analysieren kann.
Dies könnte zu neuen Erkenntnissen über den Zusammenhang von Form und Funktion bei diesen Proteinen führen und dazu beitragen, die Prozesse bei der Entstehung von Alzheimer und anderen Krankheiten besser zu verstehen. Die Studie wurde im Fachjournal Angewandte Chemie als „Very Important Paper“ vorgestellt.
Proteine sind im Körper notwendig für zahlreiche grundlegende Funktionen wie den Stoffwechsel und die Immunabwehr, leiten als Rezeptoren Signale weiter und steuern biochemische Reaktionen in der Zelle. So vielfältig wie diese Aufgaben sind auch die räumlichen Strukturen, die Proteine annehmen können.
Eine der wichtigsten Methoden, um die jeweilige Form der langen, dreidimensional gefalteten Aminosäureketten zu entschlüsseln, ist die NMR (engl. „nuclear magnetic resonance“)-Spektroskopie, mit der sich die Position der einzelnen Atome im Molekül bestimmen lässt. „Manche funktionalen Zustände kann man so jedoch nicht abbilden, weil sie, bezogen auf die Gesamtzahl der Proteine in einer Probe, sehr selten auftreten und nur für Sekundenbruchteile bestehen“, erklärt Prof. Dieter Willbold, Direktor des Institute of Complex Systems, Bereich Strukturbiochemie (ICS-6) am Forschungszentrum Jülich.
Erste Erfolge, dynamische Vorgänge sichtbar zu machen, erzielte eine Forschergruppe mit Jülicher Beteiligung kürzlich bereits mithilfe einer Weiterentwicklung der Flüssig-NMR-Spektroskopie. Diese Methode ist allerdings nur auf kleinere und in Lösung befindliche Proteine anwendbar. Ein Team von Wissenschaftlern des Institut de Biologie Structurale (IBS) des Commissariats à l'Energie Atomique in Grenoble und des Forschungszentrums Jülich hat nun eine neues Verfahren auf Basis der Festkörper-NMR entwickelt, das diese Limitierung aufhebt.
Die Methode erlaubt direktere Einblicke als bisher, indem sie Informationen über die Winkel zwischen den einzelnen Verbindungen innerhalb des Proteinmoleküls liefert. „Wenn man eine große Zahl solcher Winkel kennt, kann man die Struktur rekonstruieren, auch wenn diese nur eine kurze Lebensdauer hat und zu einem kleinen Prozentsatz in der Probe vorliegt“, sagt Paul Schanda, Forschungsgruppenleiter für Festkörper-NMR am IBS.
Die Zuverlässigkeit des Verfahrens wurde am gut erforschten und häufig vorkommenden Protein Ubiquitin geprüft, welches an viele andere Proteine bindet. In früheren Studien war aufgefallen, dass an einer bestimmten Stelle von Ubiquitin zwei leicht unterschiedliche Strukturen zu existieren scheinen. „Mit bisherigen Methoden erhielt man mal die eine, mal die andere Struktur. Mit dem neuen Verfahren konnten wir zeigen, dass in der Probe stets beide Strukturen vorliegen: Ubiquitin springt ständig zwischen den beiden Zuständen hin und her“, erklärt Paul Schanda.
Das Verfahren ließe sich beispielsweise auf Enzym- und Membranproteine anwenden, die zu den wichtigsten Angriffspunkten für medizinische Wirkstoffe gehören. Ebenso nützlich könnte es für die Alzheimerforschung werden, die einen der Schwerpunkte der Jülicher Forscher darstellt. Bei dieser Krankheit verklumpen eigentlich harmlose Proteine im Hirn zu schädlichen Aggregaten. „Wahrscheinlich stehen am Anfang zwei oder mehr A-beta-Proteine, die in einem seltenen Faltungszustand aneinander binden und einen Aggregationskeim bilden. Vorgänge wie diese können wir jetzt wesentlich besser erforschen“, sagt Dieter Willbold. „Das ist ein wichtiges Ergebnis der sehr fruchtbaren und seit mehr als fünf Jahren funktionierenden Kooperation zwischen dem Forschungszentrum Jülich und dem IBS in der strukturbiologischen Forschung“, betont der Wissenschaftler.
Peixiang Ma et al.: Probing Transient Conformational States of Proteins by Solid-State R1ρ Relaxation-Dispersion NMR Spectroscopy. Angew. Chem. Int. Ed., 53: 4312–4317. doi: 10.1002/anie.201311275
Prof. Dr. Dieter Willbold
Institut for Complex Systems, Strukturbiochemie (ICS-6)
Forschungszentrum Jülich
Institut für Physikalische Biologie, Heinrich-Heine-Universität Düsseldorf
Tel.: +49 2461 61-2100
E-Mail: d.willbold@fz-juelich.de
Dr. Paul Schanda
Institut de Biologie Structurale. Grenoble, Frankreich
Tel.: +33 457 428-659
E-Mail: paul.schanda@ibs.fr
Annette Stettien
Tel.: +49 2461 61-2388
E-Mail: a.stettien@fz-juelich.de
Media Contact
Alle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Diamantstaub leuchtet hell in Magnetresonanztomographie
Mögliche Alternative zum weit verbreiteten Kontrastmittel Gadolinium. Eine unerwartete Entdeckung machte eine Wissenschaftlerin des Max-Planck-Instituts für Intelligente Systeme in Stuttgart: Nanometerkleine Diamantpartikel, die eigentlich für einen ganz anderen Zweck bestimmt…
Neue Spule für 7-Tesla MRT | Kopf und Hals gleichzeitig darstellen
Die Magnetresonanztomographie (MRT) ermöglicht detaillierte Einblicke in den Körper. Vor allem die Ultrahochfeld-Bildgebung mit Magnetfeldstärken von 7 Tesla und höher macht feinste anatomische Strukturen und funktionelle Prozesse sichtbar. Doch alleine…

Hybrid-Energiespeichersystem für moderne Energienetze
Projekt HyFlow: Leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem für moderne Energienetze. In drei Jahren Forschungsarbeit hat das Konsortium des EU-Projekts HyFlow ein extrem leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem entwickelt, das einen…