Einblicke in die Struktur der molekularen Spleißmaschine

In menschlichen Zellen ist der genetische Bauplan von Proteinen in langen Desoxyribonukleinsäure (DNA)-Molekülen verschlüsselt. Um die Information für die Proteinherstellung nutzbar zu machen, müssen im Zellkern zunächst Ribonukleinsäure-(RNA)-Kopien der DNA erstellt werden. Diese RNA-Kopien können jedoch erst dann für die Proteinherstellung verwendet werden, wenn durch präzisen Zuschnitt einige nicht benötigte interne Bereiche (sog. „Introns“) entfernt und die informationsrelevanten Bereiche („Exons“) neu verbunden worden sind.
Das Entfernen der Introns wird in Anlehnung an das Trennen und Verknüpfen von Seilenden in der Seefahrt als „Spleißen“ bezeichnet. Komplexe Makromoleküle, die so genannten Spleißosomen, führen diese Aufgabe aus. Sie bestehen ihrerseits aus über 150 verschiedenen Proteinen sowie einigen RNA-Molekülen, die sich für jede Runde von Schnitt und Verknüpfung auf der RNA zusammenbauen müssen. Dazu werden im menschlichen Zellkern aus der Vielzahl dieser Einzelkomponenten zunächst eine Reihe vorgefertigter Komplexe (sog. snRNPs) bereitgestellt, die dann den Zusammenbau des vollständigen Spleißosoms in wenigen Schritten ermöglichen. Spleißosomen wählen die zu verknüpfenden Exons gewebespezifisch aus und erhöhen somit dramatisch die Proteinvielfalt im menschlichen Körper. Die Bedeutung dieses Vorgangs erschließt sich aus zahlreichen, mit Fehlfunktionen des Spleißens assoziierten Erkrankungen, darunter viele bösartige Tumorkrankheiten, in denen die Präzision der Exon-Verknüpfung gestört ist.
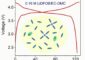
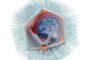
Wissenschaftlern des Max-Planck-Instituts für biophysikalische Chemie ist es jetzt gelungen, die dreidimensionale (3D) Struktur der größten Untereinheit des Spleißosoms (des sog. „U4/U6.U5 tri-snRNP“) mit Hilfe eines hochauflösenden Elektronenmikroskops zu entschlüsseln und so dessen Aufbau in einer präzisen Karte darzustellen. Eine Schlüsselkomponente der korrekten Exonerkennung ist das im tri-snRNP vorkommende RNA-Mokekül „U5 snRNA“, das nun einem kleinen zentralen Bereich des nur 30 Millionstel Millimeter großen tri-snRNP direkt zugeordnet werden konnte.
Interessanterweise ist der tri-snRNP wiederum aus zwei Untereinheiten, dem U4/U6 snRNP und dem U5 snRNP aufgebaut, die zusätzlich jeweils separat aus menschlichen Zellkernen gewonnen und in ihrer 3D Struktur untersucht werden konnten. Dabei konnte gezeigt werden, dass die molekularen Maschinen auch in Abwesenheit eines RNA-Substrates ständig in Bewegung sind und sich das U5 snRNA-Molekül in einem hochdynamischen Teil des tri-snRNP befindet. Dieser Teil wird beim Zusammenbau des tri-snRNPs aus seinen Komponenten von einer gekrümmten in eine längliche Form „verbogen“, wodurch seine Beweglichkeit eingeschränkt, aber nicht vollständig aufgehoben wird. Zukünftige Studien sollen nun die strukturelle Grundlage der Exon-Erkennung näher untersuchen.
Bei der hier verwendeten Methode der 3D Kryo-Elektronenmikroskopie werden die Makromoleküle vor den schädigenden Einflüssen des Elektronenstrahls geschützt, indem sie umgeben von Eis auf -180°C abgekühlt werden. Dann werden, ähnlich wie bei der medizischen Computertomographie, Projektionsbilder der Moleküle erstellt, aus denen anschließend mittels aufwendiger Rechenverfahren die Struktur bestimmt wird. In Zusammenarbeit mit Wissenschaftlern der Abteilung für Zelluläre Biochemie von Prof. Reinhard Lührmann und der „Bio-Future“-Forschungsgruppe für 3D Kryo-Elektronenmikroskopie von Dr. Holger Stark konnten Dr. Björn Sander, Dr. Monika Golas und Dr. Berthold Kastner die Molekülkomplexe aus Zellkulturen isolieren und erstmals für die 3D Kryo-Elektronenmikroskopie verfügbar machen. Hierbei waren in der Arbeitsgruppe entwickelte, neue Verfahren zur Probenaufbereitung und Strukturberechnung entscheidend, die eine Auflösung der tri-snRNP Struktur von 2 Millionstel Millimetern ermöglichten.
Originalveröffentlichung:
Bjoern Sander, Monika M. Golas, Evgeny M. Makarov, Hero Brahms, Berthold Kastner, Reinhard Lührmann, and Holger Stark: Organization of Core Spliceosomal Components U5 snRNA Loop I and U4/U6 Di-snRNP within U4/U6.U5 Tri-snRNP as Revealed by Electron Cryomicroscopy. Molecular Cell 24, 267-278 (20.Oktober 2006). DOI 10.1016/j.molcel.2006.08.021
Weitere Informationen:
Dr. Holger Stark, Max-Planck-Institut für biophysikalische Chemie, FG 3D Kryo-Elektronenmikroskopie, Am Fassberg 11, 37077 Göttingen, Tel: 0551 201-1305, Fax: -1197
Media Contact
Weitere Informationen:
http://www.mpibpc.mpg.de/groups/stark/ http://www.mpibpc.mpg.de/groups/pr/PR/2006/06_22/Alle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Diamantstaub leuchtet hell in Magnetresonanztomographie
Mögliche Alternative zum weit verbreiteten Kontrastmittel Gadolinium. Eine unerwartete Entdeckung machte eine Wissenschaftlerin des Max-Planck-Instituts für Intelligente Systeme in Stuttgart: Nanometerkleine Diamantpartikel, die eigentlich für einen ganz anderen Zweck bestimmt…
Neue Spule für 7-Tesla MRT | Kopf und Hals gleichzeitig darstellen
Die Magnetresonanztomographie (MRT) ermöglicht detaillierte Einblicke in den Körper. Vor allem die Ultrahochfeld-Bildgebung mit Magnetfeldstärken von 7 Tesla und höher macht feinste anatomische Strukturen und funktionelle Prozesse sichtbar. Doch alleine…

Hybrid-Energiespeichersystem für moderne Energienetze
Projekt HyFlow: Leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem für moderne Energienetze. In drei Jahren Forschungsarbeit hat das Konsortium des EU-Projekts HyFlow ein extrem leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem entwickelt, das einen…