Die kleinste Angelrute der Welt
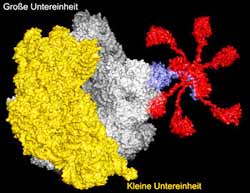
Funktionelle Untereinheiten eines Ribosoms. Die kleine und große Untereinheit fügen sich zum Gesamtribosom zusammen. In einer zentralen Höhlung findet die Proteinsynthese statt. Hier kommen auch Translationsfaktoren zu liegen. Ein Modul zur Rekrutierung und Schaltung der Translationsfaktoren (blaue und rote Proteine) befindet sich in unmittelbarer Nachbarschaft. Die Köpfe der roten Molekülketten bilden Andock- und Schaltstellen für die Translationsfaktoren. Bild: MPI für biophysikalische Chemie
Wissenschaftlern gelingt tiefgreifender Einblick in die Schaltstation der Proteinsynthese-Maschinerie
Ein Konsortium von Wissenschaftlern des Max-Planck-Instituts für biophysikalische Chemie in Göttingen, einer Max-Planck Arbeitsgruppe am DESY in Hamburg, der Universität Witten-Herdecke und der Moskauer Lomonosov Universität hat die atomare Struktur eines wichtigen Regulationszentrums der Proteinsynthesemaschinerie in lebenden Zellen aufgeklärt und ist dem Verständnis seiner Funktionsweise damit einen entscheidenden Schritt näher gekommen. In der aktuellen Ausgabe der Zeitschrift Cell (1. Juli 2005) beschreiben die Wissenschaftler, mit welchen Strukturen das Ribosom nach den so genannten Translationsfaktoren „fischt“ und diese molekularen Schalter betätigt.
Die überwiegende Zahl der biochemischen Vorgänge und Funktionen in lebenden Zellen, wie z.B. Katalyse von Reaktionen, Gerüstbildung, Kommunikation und Transport, werden von Proteinen unterhalten. Die Bauanleitungen für diese Proteine sind in der Erbinformation (DNS) einer jeden Zelle festgeschrieben. Sie werden abgerufen, indem zunächst Blaupausen der DNS in Form der Boten-Ribonukleinsäuren (RNS) angelegt und anschließend in eine Kette von Aminosäuren, die Proteinbausteine, übersetzt werden. Für diese Übersetzung („Translation“) ist das Ribosom zuständig.
Mit einem Durchmesser von ca. 25 Milliardstel Millimetern stellen bereits die einfachsten Ribosomen aus Bakterien auf molekularer Ebene riesige Aggregate dar. Sie bestehen aus über fünfzig Proteinkomponenten und drei langen RNS-Molekülen, die zu einer großen und einer kleinen ribosomalen Untereinheit zusammengesetzt sind (siehe Bild). In Aufbau und Funktionsweise ist ein Ribosom einer Miniatur-Maschinerie vergleichbar: Die Boten-RNS wird wie ein Fließband durch diese Maschine hindurchgeschleust. Dabei wird das fadenförmige Botenmolekül Schritt für Schritt abgetastet; zu jedem Nukleinsäuretriplett existiert ein passendes Adaptermolekül, eine Transport-RNS (t-RNS), die eine bestimmte Aminosäure transportiert. Die Aminosäuren werden nacheinander zu einer Kette zusammengefügt und ergeben schließlich ein neues Proteinmolekül.
Für jede Teilaufgabe, wie z.B. die Auswahl der passenden t-RNS, das Zusammenfügen der einzelnen Proteinbausteine oder das Entsorgen entladener t-RNS, ist ein spezielles Modul des Ribosoms zuständig. Um Fehler bei der Synthese der Proteine, von denen einige mehrere tausend Bausteine umfassen, weitestgehend zu vermeiden, müssen die einzelnen Module und ihre Arbeitsgänge genau aufeinander abgestimmt sein. Dazu bedient sich das Ribosom einer Reihe von Kontrollproteinen, so genannter Translationsfaktoren, die nur zu bestimmten Zeitpunkten an die zentrale Maschinerie andocken. Einige der Translationsfaktoren funktionieren dabei als molekulare Schalter. Sie tragen kleine, energiereiche Moleküle, die während eines Arbeitsganges chemisch gespalten werden. Diese Spaltung zieht eine Formveränderung der Faktoren nach sich, die vom Ribosom wahrgenommen wird und den Startschuss zur Einleitung des nächsten Arbeitsschrittes gibt. Das Einholen der Translationsfaktoren und das Umlegen der molekularen Schalter werden von einer speziellen Schaltzentrale am Ribosom koordiniert. Obwohl die Bestandteile dieser Schaltzentrale seit längerem bekannt waren, wusste man bisher wenig über die Art und Weise ihrer Funktion.
Um dieser Funktion auf die Schliche zu kommen, hat die Arbeitsgruppe von Markus Wahl vom Max-Planck-Institut für biophysikalische Chemie zunächst ein detailliertes, dreidimensionales Bild dieses Ribosomenbereiches erstellt. Sie züchteten Kristalle von Teilen des Schaltzentrums und untersuchten deren Streuung im Röntgenlicht. Aus den Streudaten konnten sie die atomare Struktur dieser Komponenten auf 0,2 Nanometer genau berechnen. Frank Schlünzen und Jörg Harms am DESY ermittelten über ähnliche Verfahren die Verankerung des Schaltzentrums an der großen ribosomalen Untereinheit. Wie in einem dreidimensionalen Puzzlespiel passten die Forscher dann alle Teilstrukturen in Hüllen des Ribosoms ein, die mithilfe der Elektronenmikroskopie im Arbeitskreis von Holger Stark bei etwa zehnfach niedrigerer Auflösung sichtbar gemacht wurden. „Von der großen ribosomalen Untereinheit in unmittelbarer Nähe der Stelle, an der die Translationsfaktoren zu liegen kommen, erstreckt sich ein langer, beweglicher Fortsatz“, beschreibt Markus Wahl das Bild. „An diesem Fortsatz sind bis zu sechs flexible Molekülketten aufgehängt, jede mit einem kugelförmigen Kopf.“ (Abb. 1).
Im Einklang mit früheren Arbeiten, die darauf hindeuteten, dass die Köpfe die ersten Andockstellen für die Translationsfaktoren darstellten, ähnelte die Struktur einer molekularen Angelrute mit sechs Schnüren und je einem Köder, mit denen das Ribosom nach Translationfaktoren „fischen“ konnte. Die Forscher vermuteten außerdem, dass die Köpfe auch an die Ribosom-gebundenen Faktoren heranreichen könnten, um deren Schalter umzulegen. Das Labor von Marina Rodnina in Witten testete diese Hypthesen durch gezielte Veränderungen an der „Angelrute“: Zunächst wurden die „Köder“ durch genetische Verfahren abgeschnitten. Wie erwartet waren die Angelschnüre ohne Köder erfolglos beim „Fischen“ nach Faktoren. Auch die Schaltprozesse waren erwartungsgemäß etwa 1000-fach verlangsamt. Dann veränderten die Biochemiker gezielt Oberflächenbausteine der Köpfe, die mit den Translationsfaktoren in Berührung kommen konnten, und störten somit ihre Funktionsweise. „Unsere Ergebnisse zeigten, dass eine ganze Reihe solcher Bausteine gemeinsam für das Einholen der Faktoren und das Umlegen der Schalter verantwortlich sind“, erklärt Marina Rodnina.
Da die Fähigkeit zur Proteinsynthese eine elementare Grundlage für alles Leben auf unserer Erde ist, kommen Ribosomen in allen Organismen, vom Bakterium bis zum Menschen, vor und ähneln sich in ihrem Aufbau. Die bakteriellen Ribosomen weisen jedoch Detailunterschiede zu den Ribosomen höherer Organismen auf. So verhindern z.B. einige Antibiotika die Proteinsynthese in Bakterien, aber nicht bei Menschen, Tieren oder Pflanzen. Auch die Schaltzentrale des Ribosoms weist Unterschiede zwischen Bakterien und höheren Organismen auf. Eine genauere Kenntnis der verschiedenen Translationsprozesse könnte daher eine Grundlage zur Neuentwicklung beispielsweise von Medikamenten gegen Infektionskrankheiten bieten.
Media Contact
Weitere Informationen:
http://www.gwdg.deAlle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Neue universelle lichtbasierte Technik zur Kontrolle der Talpolarisation
Ein internationales Forscherteam berichtet in Nature über eine neue Methode, mit der zum ersten Mal die Talpolarisation in zentrosymmetrischen Bulk-Materialien auf eine nicht materialspezifische Weise erreicht wird. Diese „universelle Technik“…

Tumorzellen hebeln das Immunsystem früh aus
Neu entdeckter Mechanismus könnte Krebs-Immuntherapien deutlich verbessern. Tumore verhindern aktiv, dass sich Immunantworten durch sogenannte zytotoxische T-Zellen bilden, die den Krebs bekämpfen könnten. Wie das genau geschieht, beschreiben jetzt erstmals…

Immunzellen in den Startlöchern: „Allzeit bereit“ ist harte Arbeit
Wenn Krankheitserreger in den Körper eindringen, muss das Immunsystem sofort reagieren und eine Infektion verhindern oder eindämmen. Doch wie halten sich unsere Abwehrzellen bereit, wenn kein Angreifer in Sicht ist?…





















