Pfad der RNA enthüllt – Satelliten-Navigation für Biomoleküle

Im Rahmen des Exzellenzclusters Nanosystems Initiative Munich (NIM) haben sie eine Methode entwickelt, mit der sich die Moleküle an der Spitze der RNA beim Transkriptionsprozess nanometergenau verfolgen lassen.
Bei dem in der aktuellen Ausgabe der Fachzeitschrift „Proceedings of the National Academy of Sciences“ (PNAS) vorgestellten Verfahren wird unter Ausnutzung des Energieübertrags zwischen Fluoreszenzfarbstoffen die Distanz des RNA-Moleküls zu mindestens drei fest verorteten Molekülen gemessen und daraus wie bei der Satelliten-Navigation die Position des RNA-Moleküls bestimmt. Diese Beobachtungsmöglichkeit bietet eine wichtige Grundlage für das Verständnis von Mechanismen der Genregulation.
Damit eine Zelle neue Proteine erzeugen kann, holt sie sich den Bauplan für die neuen Eiweißstrukturen aus der DNA. Dabei werden die erforderlichen Gensequenzen der DNA zunächst auf eine Boten-RNA kopiert, auf Englisch „messenger-RNA“, kurz mRNA. Der Kopiervorgang von der DNA zur mRNA geschieht in einem darauf spezialisierten Protein, der RNA-Polymerase. Die kann man sich als winzigen Kopierautomaten vorstellen. An einem Ende wird die DNA eingeschleust, am anderen Ende kommen DNA und mRNA heraus. Die Produktion der gewünschten und von der DNA vorgegebenen Zellbausteine geschieht im Anschluss.
Dieser Ablauf ist schon recht lange bekannt. Um aber genau nachvollziehen zu können, wie die RNA nach dem Verlassen der Polymerase weiterbehandelt wird, muss man wissen, auf welchem Weg die RNA aus der Polymerase herauskommt. Mit den bisher angewandten Messmethoden, etwa der Röntgenstrukturanalyse, lässt sich zwar gut der Weg der RNA im Polymerase-Molekül verfolgen. Sobald die RNA aber die Polymerase verlassen hat, versagt die Methode, weil sich die RNA-Moleküle dann flexibel zwischen mehreren Positionen hin und her bewegen können. Eine solche Dynamik lässt sich mit der Röntgenstrukturanalyse nicht abbilden. Man bekäme als Messergebnis nur eine ungenau bestimmte mittlere Position.
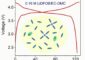
Einen genaueren Einblick verschafft eine neue Methode, die in den Arbeitsgruppen von Michaelis und Cramer im Center for NanoScience (CeNS) der LMU entwickelt wurde. Grundlage ist die Messung des Fluoreszenz-Resonanz-Energie-Transfers (FRET) mit einem Fluoreszenz-Mikroskop. Dabei überträgt ein angeregtes Fluoreszenzfarbstoffmolekül einen Teil seiner Energie auf ein zweites Farbstoffmolekül. Die übertragene Energie und damit die Intensität des gemessenen FRET-Signals hängt sehr empfindlich vom Abstand der beiden Farbstoffmoleküle ab. So lassen sich Entfernungen im Nanometerbereich bestimmen. Diese Methode kombinierten Michaelis, Cramer und ihre Mitarbeiter mit einem Verfahren, das aus der Satelliten-Navigation (Globales Positionierungs-System GPS) bekannt ist. Um den Ort eines Objekts präzise zu bestimmen, benötigt man nur seine Distanz zu drei bekannten Positionen. So peilt ein GPS-Empfänger in einem Navigationsgerät drei Satelliten an, misst die Distanz zu diesen und errechnet daraus die eigene Position.
Um die Position von RNA-Molekülen zu bestimmen, markierten die Münchener Wissenschaftler zwei bekannte Stellen auf der zu kopierenden DNA und noch zwei weitere Stellen im RNA-Polymerase-Molekül mit unterschiedlichen Fluoreszenz-Farbstoffen, die in Analogie zum GPS als „Satelliten“ betrachtet werden können. Ein weiterer Farbstoff kam ans vordere Ende der RNA. Aus den Distanzen der vier Satelliten zum RNA-Molekül konnten sie nun dessen genaue Position ermitteln. „Wir haben so gewissermaßen ein Nano-Positionier-System (NPS) entwickelt und damit für mehrere verschieden lange RNA-Sequenzen deren Lage nach Austritt aus der Polymerase bestimmt“, sagt Michaelis. Im Prinzip ist das Farbstoffmolekül auf der RNA also ein Beobachter an der Spitze eines Zuges, an den immer mehr Wagen angehängt werden. Es schaut aus dem Fenster und beobachtet, wie sich seine Umgebung und seine Position verändert, je länger der Zug wird.
Ein erster Schritt ist damit getan. Als Nächstes wollen die Wissenschaftler untersuchen, was passiert, wenn die RNA-Produktion ins Stocken gerät. Dies kann zum Beispiel durch einen Fehler in der abgelesenen DNA verursacht werden. Um die Polymerase-Maschine dann wieder zum Laufen zu bringen, muss dieser Fehler zunächst repariert werden. Aber wie genau funktioniert das? Die Forscher erhoffen sich von ihrer Arbeit weitere Erkenntnisse über diesen Reparaturprozess. Solche Erkenntnisse könnten möglicherweise zur Entwicklung von Behandlungsmethoden für Krankheiten führen, bei denen eine Störung der DNA-Reparatur vorliegt.
Die aktuell in den PNAS vorgestellten Arbeiten entstanden im Rahmen der Exzellenz-Cluster „Nanosystems Initiative Munich“ (NIM) und „Center for Integrated Protein Science Munich“ (CiPSM). NIM hat es sich zum Ziel gesetzt, funktionale Nanostrukturen für Anwendungen in der Medizin und in der Informationsverarbeitung zu entwickeln, zu erforschen und zum Einsatz zu bringen. Der Schwerpunkt der Forschung in CiPSM zielt auf das fundamentale Verständnis der Funktion von Proteinen in lebenden Systemen.
Publikation:
„Single-molecule tracking of mRNA exiting from RNApolymerase II“,
Joanna Andrecka, Robert Lewis, Florian Brueckner, Elisabeth Lehmann, Patrick Cramer and Jens Michaelis, PNAS am 27.12.2007
Ansprechpartner:
Professor Jens Michaelis
Department für Chemie und Biochemie der LMU
Geschäftsführender Direktor, Max-Planck-Institut für Quantenoptik
Tel.: 089 / 2180-77561
Fax: 089 / 2180-99 77561
E-Mail: michaelis@lmu.de
Web: www.cup.uni-muenchen.de/pc/michaelis
Professor Patrick Cramer
Department für Chemie und Biochemie sowie Genzentrum der LMU
Tel.: 089 / 2180-76951
Fax: 089 / 2180-76999
E-Mail: cramer@lmb.uni-muenchen.de
Dr. Peter Sonntag
Nanosystems Initiative Munich (NIM)
Presse- und Öffentlichkeitsarbeit
Tel.: 089 / 2180-5091
Fax: 089 / 2180-5694
E-Mail: peter.sonntag@lmu.de
Media Contact
Weitere Informationen:
http://www.uni-muenchen.de/Alle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Hybrid-Energiespeichersystem für moderne Energienetze
Projekt HyFlow: Leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem für moderne Energienetze. In drei Jahren Forschungsarbeit hat das Konsortium des EU-Projekts HyFlow ein extrem leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem entwickelt, das einen…
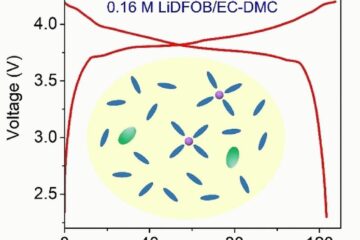
Für kostengünstigere, nachhaltigere Akkus
Ultraniedrig konzentrierter Elektrolyt für Lithium-Ionen-Batterien Lithium-Salze machen Akkumulatoren leistungsfähig, aber teuer. Ein ultraniedrig konzentrierter Elektrolyt auf Basis des Lithium-Salzes LiDFOB könnte eine kostengünstige und dabei nachhaltigere Alternative sein. Zellen mit…

Chemischer Seiltrick auf molekularer Ebene
Mechanismusforschung hilft, wenn „Trial & Error“ versagt. In den meisten industriellen chemischen Reaktionen verbinden sich Katalysatoren mit den Ausgangsstoffen und begleiten sie über Zwischenstufen zum Produkt. Dieser Weg wird in…