Methodenentwicklung an BESSY II: Automatische Auswertung beschleunigt die Suche nach neuen Wirkstoffen
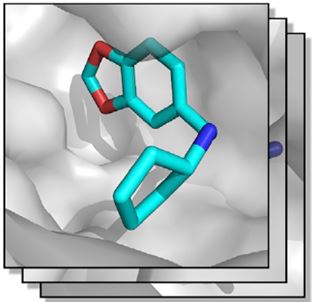
Nach vorgegebenen Kriterien identifiziert das Expertensystem Kandidaten, die am Zielprotein anbinden. Structure 2016: doi: 10.1016/j.str.2016
Die Suche nach neuen Wirkstoffen gegen ein Krankheitsbild gleicht im ersten Schritt dem Aufknacken eines Hochsicherheitsschlosses: Der Wirkstoff muss wie ein Schlüssel in das Zielmolekül hineinpassen. Das Zielmolekül ist dabei ein Protein (Eiweißmolekül), das im Organismus eine bestimmte Funktion erfüllt. Mit dem passenden Wirkstoff lässt sich beeinflussen, ob und wie gut es diese Funktion ausüben kann. So lässt sich im Krankheitsfall mit dem geeigneten Wirkstoff eine aus den Fugen geratene Funktion korrigieren. Allerdings muss man den richtigen Wirkstoff zunächst identifizieren.
Und diese Suche kann sehr langwierig sein: Bildlich gesprochen gibt es unendlich viele Schlüssel, und es kostet Zeit, sie alle auszuprobieren. Ein effizienter Panzerknacker sucht daher nicht lange in einem Berg von Schlüsseln herum, sondern tastet Zacken um Zacken des Schlosses ab, bis er die richtige Arretierung einstellen kann.
Ein ähnliches Verfahren hat sich mit dem Fragmentscreening auch in der Strukturbiologie etabliert: Um rascher den passenden „Schlüsselrohling“ zu finden, setzt man ihn aus einzelnen „Fragmenten“ zusammen. Dafür testet man Hunderte von sehr kleinen Molekülen (Fragmente) darauf, ob sie an das Zielprotein andocken. Aus den passenden Fragmenten wird dann die „Leitstruktur“ für den Wirkstoff zusammengesetzt.
Hochautomatisiert und stark nachgefragt: Die MX-Beamlines an BESSY II
An den MX-Beamlines von BESSY II läuft dieses Verfahren bereits seit drei Jahren hochautomatisiert und ermöglicht einen großen Durchsatz von Proben. Die MX-Beamlines sind in Deutschland mit bisher über 2000 bestimmten Proteinstrukturen mit Abstand am produktivsten, sie sind stark überbucht und die Nachfrage kommt dabei sowohl aus der Grundlagenforschung als auch von Nutzergruppen aus der Industrie.
Neues Expertensystem automatisiert auch die Auswertung
Nun ist es im Rahmen eines Verbundforschungsprojekts zwischen der Philipps-Universität Marburg und dem HZB gelungen, diese Automatisierung noch einen Schritt weiter zu führen: „Mit dem Fragmentscreening können wir Hunderte von Proben durchmessen, sodass die Auswertung von Hand eigentlich nicht mehr sinnvoll machbar ist“, sagt Dr. Manfred Weiss, der am HZB das MX-Team leitet. Mit der Gruppe des Kooperationspartners Prof. Dr. Gerhard Klebe, Uni Marburg, entwickelten beide Teams ein Computerprogramm (Expertensystem), in das die Expertise der Wissenschaftlerinnen und Wissenschaftler eingeflossen ist. Dieses Expertensystem wertet die Rohdaten einer Röntgenstrukturanalyse mit einer Kaskade von hintereinandergeschalteten Auswerteroutinen aus und identifiziert diejenigen Fragmente, die an das Protein anbinden. Dabei stellte die Gruppe um Klebe die Protein-Kristalle und eine ganze Bibliothek unterschiedlicher Fragmentmoleküle zur Verfügung.
Verfeinerung identifiziert zusätzliche Kandidaten
Insgesamt testeten die Kooperationspartner mit dem neu konzipierten Expertensystem 364 Kristalle aus einem bestimmten Protein, die mit unterschiedlichen Fragmentmolekülen getränkt waren. Dabei ist das Expertensystem auch in der Lage, die Strukturanalysen zu verfeinern: „Ohne die Verfeinerung fällt ein Teil der geeigneten Fragmente durchs Raster“, erklärt Weiss. „So hatten wir im ersten Durchgang nur etwa 50 Kandidaten identifiziert, die Verfeinerung hat dann zusätzlich weitere 32 Kandidaten aufgezeigt.“ Die Arbeit zeigt, dass das neue Expertensystem präzise Aussagen ermöglicht, welche Fragmente als Bestandteile der gesuchten Leitstruktur infrage kommen. Dadurch kann sich die Suche nach Wirkstoffen beschleunigen.
Anmerkung: Die Kooperation wird als Verbundforschungsprojekt im Rahmen der Fördermaßnahme „Erforschung Kondensierter Materie an Großgeräten“ (2016-2019) durch das BMBF gefördert. Am Ende des Projekts werden alle Nutzer der MX-Beamlines auch auf die Bibliothek an Fragmentmolekülen sowie das Expertensystem zugreifen können.
Zur Publikation: High-Throughput Crystallography: Reliable and Efficient Identification of Fragment Hits, Structure, (2016) DOI: 10.1016/j.str.2016.06.010
Media Contact
Weitere Informationen:
http://www.helmholtz-berlin.de/pubbin/news_seite?nid=14523&sprache=de&typoid=1Alle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Diamantstaub leuchtet hell in Magnetresonanztomographie
Mögliche Alternative zum weit verbreiteten Kontrastmittel Gadolinium. Eine unerwartete Entdeckung machte eine Wissenschaftlerin des Max-Planck-Instituts für Intelligente Systeme in Stuttgart: Nanometerkleine Diamantpartikel, die eigentlich für einen ganz anderen Zweck bestimmt…
Neue Spule für 7-Tesla MRT | Kopf und Hals gleichzeitig darstellen
Die Magnetresonanztomographie (MRT) ermöglicht detaillierte Einblicke in den Körper. Vor allem die Ultrahochfeld-Bildgebung mit Magnetfeldstärken von 7 Tesla und höher macht feinste anatomische Strukturen und funktionelle Prozesse sichtbar. Doch alleine…

Hybrid-Energiespeichersystem für moderne Energienetze
Projekt HyFlow: Leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem für moderne Energienetze. In drei Jahren Forschungsarbeit hat das Konsortium des EU-Projekts HyFlow ein extrem leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem entwickelt, das einen…