Revolutioniert DNA-Barcoding die Gewässergüteanalyse?
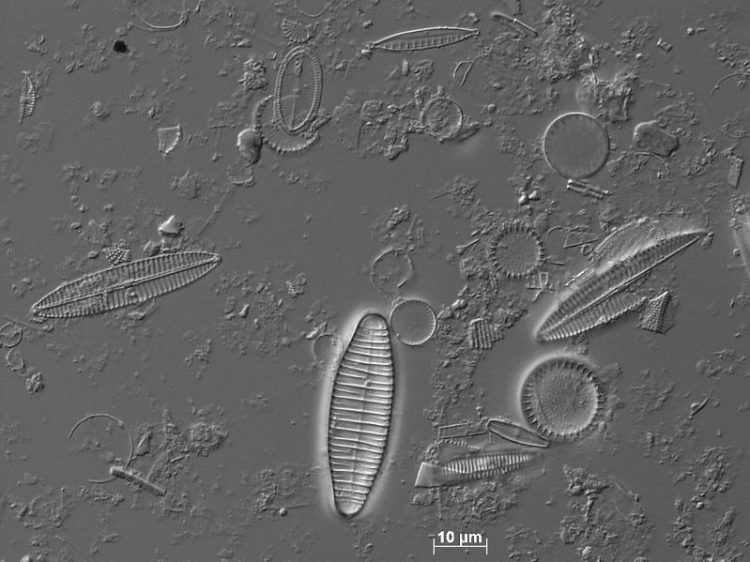
Eine Gewässerprobe der Lausitzer Neiße unter dem Lichtmikroskop: Die Artenfülle von Kieselalgen wird deutlich. Forschungsgruppe Diatomeen, Botanischer Garten und Botanisches Museum Berlin-Dahlem
Die moderne Bestimmung von Kieselalgenarten anhand ihrer Erbinformation, das DNA-Barcoding, ist demzufolge fast dreimal so genau wie die sehr detaillierte morphologische Untersuchung: Während molekulargenetisch 270 Taxa (d.h. Arten und Unterarten) entdeckt wurden, konnten morphologisch nur 103 Taxa identifiziert werden. Kieselalgen werden routinemäßig als Bioindikatoren innerhalb der EU-Wasserrahmenrichtlinie zur Bestimmung der Gewässergüte untersucht.
Die Untersuchung wird entscheidende Auswirkung für die Gewässergüteanalyse in Europa haben. Die verblüffenden Ergebnisse wurden gerade online in der renommierten Zeitschrift Molecular Ecology Resources veröffentlicht und wird in einer der nächsten gedruckten Ausgaben erscheinen. Beteiligt waren Forscherinnen und Forscher des Botanischen Gartens und Botanischen Museums Berlin-Dahlem der Freien Universität Berlin, des Leibniz-Institut für Gewässerökologie und Binnenfischerei, der Universität Köln und der Justus-Liebig-Universität Gießen.
Kieselalgen: Wichtige Bioindikatoren für die Gewässergüte
Kieselalgen (Diatomeen) sind in nahezu allen Gewässertypen zu finden. Die verschiedenen Kieselalgenarten reagieren empfindlich und spezifisch auf Änderungen der Umwelt wie Verschmutzung, Nährstoffversorgung, Säure und Salzgehalt und sind daher wichtige Bioindikatoren. Kieselalgen werden routinemäßig als Bioindikatoren innerhalb der EU-Wasserrahmenrichtlinie und global zur Bestimmung der Gewässergüte untersucht.
Dabei werden vorrangig substratbewohnende Kieselalgen untersucht, da sie im Gegensatz zu freischwimmenden Kieselalgen nicht durch Strömung oder andere Einwirkungen verdriftet werden was zur Verfälschung der Ergebnisse führen kann. Um die Kieselalgen eines Gewässers für eine Gewässergüteanalyse zu untersuchen, werden sie von Steinen oder anderen Untergründen abgekratzt und anschließend im Labor untersucht. Diese bodenlebenden Kieselalgen kennt fast jeder als rutschigen Schleimfilm auf Steinen.
Schwächen der bisherigen Gewässergüteanalyse
Bei aktuellen Gewässergüteanalysen werden die darin lebenden Kieselalgen nur lichtmikroskopisch untersucht, um Aussagen über die Wasserqualität zu treffen. Tatsächlich sind jedoch viele für die Bestimmung wichtige Merkmale lichtmikroskopisch nicht oder nur schwer zu erfassen. Zudem hängt die routinemäßige Bestimmung der im Wasser lebenden Kieselalgen von den taxonomischen Fähigkeiten und Augen derjenigen ab, die die Proben auswerten.
DNA-Barcoding: Die konkurrenzfähige neue Methode
Die Bestimmung von Kieselalgenarten anhand ihrer Erbinformation, das DNA-Barcoding, ist sehr viel objektiver und feiner als die herkömmliche lichtmikroskopische Untersuchung. Langfristig wird im DNA-Barcoding die neue Methode zur Gewässergüteanalyse gesehen. Aktuell ist das DNA-Barcoding noch teurer und zeitaufwändiger als die klassische Untersuchungsmethode. Wird das Verfahren jedoch automatisiert, lassen sich Zeit und Kosten deutlich reduzieren.
Langfristig könnte DNA-Barcoding die Gewässergüteanalyse somit revolutionieren. Wichtige Vorbedingung für erfolgreiches DNA-Barcoding ist die Erarbeitung einer Referenz-Datenbank der DNA -Sequenzen aller Kieselalgen eines Gewässersystems, da nur mit diesem die analysierten DNA -Sequenzen im Vergleich zugeordnet werden können.
Die Untersuchung an der Oder und Lausitzer Neiße ist ein Modellfall und auf andere Gewässer in Mitteleuropa übertragbar. Die Kieselalgen von sieben Standorten der beiden Flüsse wurden untersucht. Die molekulargenetische Untersuchung zeigte insgesamt 28.000 DNA -Sequenzen von Kieselalgen, die 270 Taxa (Arten und Unterarten) zugeordnet werden konnten. Der Vergleich erfolgte mithilfe einer Referenz-Datenbank mit Daten aus eigener Forschung und Daten anderer WissenschaftlerInnen.
Interessant ist, dass etwa 70 Prozent der DNA -Sequenzen bis auf die Art genau bestimmt werden konnten, etwa 30 Prozent jedoch nur bis zur Gattung, entweder weil die Art noch unbekannt und neu ist oder weil sie bisher in der Referenzdatenbank nicht vorhanden ist. Das ist umso erstaunlicher, da die Norddeutsche Tiefebene eines der am besten untersuchten Gebiete der Erde ist in Bezug auf Kieselalgen. Die WissenschaftlerInnen konnten zeigen, dass selbst hier noch vieles unerforscht ist und von der angewendeten Methodik abhängt.
Kieselalgen sind die Lunge und Nahrung der Erde
Kieselalgen sind einzellige Algen von meist nur einem Zwanzigstel Millimeter Durchmesser, für deren Beobachtung ein leistungsstarkes Mikroskop erforderlich ist. Sie leben in großer Zahl in Seen, Flüssen und Meeren und besiedeln selbst kleinste feuchte Lebensräume wie Baumrinden und Erde. Die Zahl der Diatomeenarten wird auf mehrere 100.000 geschätzt, wobei gegenwärtig erst 30.000 Kieselalgenarten beschrieben sind. Trotz ihrer geringen Größe kommt Kieselalgen eine herausragende ökologische Bedeutung zu. Der dank ihrer Photosyntheseaktivität freigesetzte Sauerstoff macht etwa 25 % der weltweiten Sauerstoffproduktion aus. Sie leisten 25 % der Kohlendioxid-Fixierung der Erde, stehen am Anfang der Nahrungskette und tragen bis zu 25-45 % zur globalen Primärproduktion bei.
Charakteristische Kieselschale
Charakteristisch für den Aufbau der Diatomeen sind ihre gläsernen Schalen aus Kieselsäure, weswegen sie auch Kieselalgen genannt werden. Die Schalen umgeben die Zelle schützend, sind sehr vielfältig gestaltet und symmetrisch durchbrochen. Die Form der strukturierten Schalen ist artspezifisch und wurde schon früh in der Naturwissenschaft systematisch erfasst.
Publikation des Artikels:
Zimmermann, J., Glöckner, G., Jahn, R., Enke, N. und Gemeinholzer, B. (2014), Metabarcoding vs. morphological identification to assess diatom diversity in environmental studies. Molecular Ecology Resources.
doi: 10.1111/1755-0998.12336
Online http://onlinelibrary.wiley.com/doi/10.1111/1755-0998.12336/abstract
Weitere Informationen geben Ihnen gern:
Dr. Regine Jahn, Leiterin der Forschungsgruppe Diatomeen,
Freie Universität Berlin, Botanischer Garten und Botanisches Museum Berlin-Dahlem,
Telefon: 030 / 838-50142, E-Mail: r.jahn@bgbm.org
Jonas Zimmermann, Forschungsgruppe Diatomeen,
Freie Universität Berlin, Botanischer Garten und Botanisches Museum Berlin-Dahlem
und Systematische Botanik, Justus-Liebig-Universität Gießen,
Telefon: 030 / 838-50144, E-Mail: j.zimmermann@bgbm.org
Weitere Informationen:
http://www.bgbm.org/de/presse/pressefotos#Kieselalgen_Oder – Pressefotos
http://onlinelibrary.wiley.com/doi/10.1111/1755-0998.12336/abstract – Artikel
Media Contact
Alle Nachrichten aus der Kategorie: Ökologie Umwelt- Naturschutz
Dieser Themenkomplex befasst sich primär mit den Wechselbeziehungen zwischen Organismen und den auf sie wirkenden Umweltfaktoren, aber auch im weiteren Sinn zwischen einzelnen unbelebten Umweltfaktoren.
Der innovations report bietet Ihnen interessante Berichte und Artikel, unter anderem zu den Teilbereichen: Klimaschutz, Landschaftsschutzgebiete, Ökosysteme, Naturparks sowie zu Untersuchungen der Leistungsfähigkeit des Naturhaushaltes.
Neueste Beiträge

Neue universelle lichtbasierte Technik zur Kontrolle der Talpolarisation
Ein internationales Forscherteam berichtet in Nature über eine neue Methode, mit der zum ersten Mal die Talpolarisation in zentrosymmetrischen Bulk-Materialien auf eine nicht materialspezifische Weise erreicht wird. Diese „universelle Technik“…

Tumorzellen hebeln das Immunsystem früh aus
Neu entdeckter Mechanismus könnte Krebs-Immuntherapien deutlich verbessern. Tumore verhindern aktiv, dass sich Immunantworten durch sogenannte zytotoxische T-Zellen bilden, die den Krebs bekämpfen könnten. Wie das genau geschieht, beschreiben jetzt erstmals…

Immunzellen in den Startlöchern: „Allzeit bereit“ ist harte Arbeit
Wenn Krankheitserreger in den Körper eindringen, muss das Immunsystem sofort reagieren und eine Infektion verhindern oder eindämmen. Doch wie halten sich unsere Abwehrzellen bereit, wenn kein Angreifer in Sicht ist?…





















