Strukturlosigkeit erleichtert Proteinsynthese
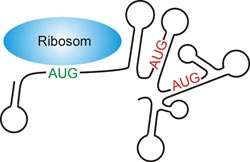
Mechanismus der Erkennung eines AUG-Startcodons durch das Ribosom (blau) in Abwesenheit einer Shine-Dalgarno-Sequenz. Das korrekte Start-AUG (grün) unterscheidet sich von allen anderen AUG-Tripletts (rot) durch seine Lage in einer einzelsträngigen (unstrukturierten) Region der Boten-RNA (schwarze Linie). Max-Planck-Institut für molekulare Pflanzenphysiologie, Potsdam<br>
Texte ohne Leerzeichen sind schwer lesbar, da der Leser Anfang und Ende eines Wortes kaum erkennen kann. Wenn in unseren Zellen die Erbinformation abgelesen und in Proteine übersetzt wird, werden die dafür verantwortlichen Enzyme mit einer ähnlichen Herausforderung konfrontiert. Sie müssen den richtigen Startpunkt für die Proteinsynthese finden.
Gene von Lebewesen ohne echten Zellkern besitzen deshalb kurz vor dem Lese-Start-Codon eine Stelle, an welche die Ablese-Proteine besonders gut binden und die somit beim Auffinden des Startpunkts behilflich ist. Doch auch Gene, die diese Sequenz nicht haben, werden trotzdem verlässlich in Proteine übersetzt. Wissenschaftler vom Max-Planck-Institut für molekulare Pflanzenphysiologie in Potsdam haben jetzt herausgefunden, dass dafür vermutlich die Struktur der Boten-RNA von entscheidender Bedeutung ist.
Die DNA von zellkernlosen Organismen besteht aus den vier Basen Adenin, Cytosin, Thymin und Guanin, die entsprechend mit den Buchstaben A, C, T, G abgekürzt werden. Bei der RNA ist Thymin durch Uracil (U) ersetzt. Die Basen sind über ihr Zucker-Phosphat-Rückgrat miteinander verknüpft. Sie sind mit den Buchstaben unserer Sprache vergleichbar, die man zu Wörtern zusammensetzen kann. In der DNA-Sprache kodieren jeweils drei Basen, sogenannte Tripletts, eine der 20 Aminosäuren, aus denen alle unsere Proteine aufgebaut sind. Da es zwischen den Tripletts in der DNA keine Leerzeichen gibt, ist es schwierig, die drei zu einem Triplett gehörenden Basen zu erkennen und vor allem den Startpunkt für die Proteinsynthese auf dem Nukleinsäurestrang auszumachen.
Bevor Proteine hergestellt werden können, wird die DNA in ihre Transportform, die Boten-RNA (mRNA), umgeschrieben und in das Zellplasma gebracht. Hier lagern sich kleine Proteinfabriken, die Ribosomen, an die mRNA an und beginnen mit ihrer Arbeit. Sie „lesen“ die Basenreihenfolge ab und übersetzen sie in Aminosäuren. Dabei starten sie weder direkt am Anfang der mRNA noch an einer beliebigen Stelle, sondern immer genau beim Basentriplett AUG, dem Start-Codon. Dieses Triplett kodiert die Aminosäure Methionin, die somit in jedem Protein die erste Aminosäure darstellt. Methionin kann jedoch auch noch an anderen Stellen mitten im Protein vorkommen. Die Frage lautet also: Woher wissen die Ribosomen, ob ein AUG-Codon ein Startsignal darstellt oder nicht?
Dabei kommt den einzelligen Lebewesen ohne echten Zellkern, den Prokaryoten, für gewöhnlich die Shine-Dalgarno-Sequenz (SD-Sequenz) zu Hilfe. Es handelt sich dabei um eine im Laufe der Evolution nahezu unveränderte Basenreihenfolge der mRNA, die sich in der Nähe des Start-Codons befindet. Die Ribosomen besitzen eine Anti-Shine-Dalgarno-Sequenz, die mit der SD-Sequenz eine starke Bindung eingehen kann. Wandert ein Ribosom die mRNA auf der Suche nach dem Start-Codon entlang, so wird es von der SD-Sequenz festgehalten und erkennt folglich auch den richtigen Startpunkt. Es gibt jedoch auch mRNAs ohne Shine-Dalgarno-Sequenz, bei denen die Ribosomen trotzdem das richtige AUG-Triplett aufspüren. Der Mechanismus, der die korrekte Identifikation des Startsignals ermöglicht, war bisher vollkommen unklar.
Neuesten Erkenntnissen nach scheint die Struktur – oder besser gesagt die Strukturlosigkeit – der mRNA dafür verantwortlich zu sein. Lars Scharff und Liam Childs vom Potsdamer Max-Planck-Institut für molekulare Pflanzenphysiologie haben mehrere zehntausend Gene von unterschiedlichen Prokaryoten und Zellorganellen auf das Vorhandensein einer Shine-Dalgarno-Sequenz hin untersucht. Sie fanden heraus, dass je nach Organismus zwischen 15 und 50 Prozent aller Gene keine SD-Sequenz besitzen. Dass die Ribosomen auch auf diesen Genen das Start-Codon erkennen, hängt vermutlich damit zusammen, dass es besonders gut zugänglich ist. Normalerweise liegt die mRNA nicht als langer Faden vor, sondern bildet Schlaufen und Haarnadelstrukturen aus. Ein Ribosom kann sich aber nur an unstrukturierte Bereiche der mRNA anlagern und genau hierin scheint das Geheimnis zu liegen. „Bei Genen ohne Shine-Dalgarno-Sequenz zeigt die mRNA um das Start-Codon herum kaum gefaltete Strukturen, anders als bei Genen mit einer Shine-Dalgarno-Sequenz“, erklärt Scharff die Erkenntnisse.
In einem Experiment zerstörten die Forscher die SD-Sequenz durch eine Mutation, wodurch sich die Rate, mit der die mRNA in Proteine übersetzt wurde, drastisch senkte. „Sobald wir eine zweite Mutation einfügten, die gleichzeitig die Struktur der mRNA am Start-Codon auflöste, verringerte sich dieser Effekt und es wurde wieder mehr Protein gebildet“, fasst Childs die Ergebnisse zusammen. Trotz fehlender SD-Sequenz wird das AUG-Codon vom Ribosom erkannt, weil es leichter zugänglich und nicht in Schlaufen und Windungen versteckt ist.
Aufbauend auf diesen Resultaten werden sich in Zukunft anhand von Strukturanalysen der mRNA bessere Vorhersagen über die Proteinsyntheserate machen lassen. Darüber hinaus könnte durch eine Änderung der mRNA-Struktur in die eine oder andere Richtung beeinflusst werden, welche Proteine gebildet werden.
Originalveröffentlichung
Lars B. Scharff, Liam Childs, Dirk Walther, Ralph Bock
Local Absence of Secondary Structure Permits Translation of mRNAs that Lack Ribosome-binding Sites
PLoS, 23. Juni 2011, DOI: DOI: 10.1371/journal.pgen.1002155
Ansprechpartner
Professor Dr. Ralph Bock
Max-Planck-Institut für Molekulare Pflanzenphysiologie, Potsdam
Telefon: +49 (0)331 567-8700
Fax: +49 (0)331 567-8701
E-Mail: rbock@mpimp-golm.mpg.de
Ursula Ross-Stitt
Max-Planck-Institut für Molekulare Pflanzenphysiologie, Potsdam
Telefon: +49 331 567-8310
Fax: +49 331 567-8983
E-Mail: ross-stitt@mpimp-gom.mpg.de
Media Contact
Weitere Informationen:
http://www.mpimp-gom.mpg.deAlle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Neue universelle lichtbasierte Technik zur Kontrolle der Talpolarisation
Ein internationales Forscherteam berichtet in Nature über eine neue Methode, mit der zum ersten Mal die Talpolarisation in zentrosymmetrischen Bulk-Materialien auf eine nicht materialspezifische Weise erreicht wird. Diese „universelle Technik“…

Tumorzellen hebeln das Immunsystem früh aus
Neu entdeckter Mechanismus könnte Krebs-Immuntherapien deutlich verbessern. Tumore verhindern aktiv, dass sich Immunantworten durch sogenannte zytotoxische T-Zellen bilden, die den Krebs bekämpfen könnten. Wie das genau geschieht, beschreiben jetzt erstmals…

Immunzellen in den Startlöchern: „Allzeit bereit“ ist harte Arbeit
Wenn Krankheitserreger in den Körper eindringen, muss das Immunsystem sofort reagieren und eine Infektion verhindern oder eindämmen. Doch wie halten sich unsere Abwehrzellen bereit, wenn kein Angreifer in Sicht ist?…





















