Protein-Arsenal der Hefe katalogisiert

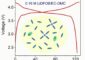
Verhältnis des Proteoms von haploiden zu diploiden Hefezellen. Die grün markierten Proteine werden verstärkt in haploiden Zellen gefunden (das Verhältnis ist 10-100), während die blau markierten Proteine vor allem in diploiden Zellen gefunden werden (Verhältnis unter 1). Bild: Max-Planck-Institut für Biochemie, Martinsried<br>
Schon seit 30 Jahren versuchen Forscher, die Gesamtheit der Proteine (Proteome) in einem Organismus zu erfassen. Doch technologische Grenzen machten dies bisher nicht möglich. Nun ist es Wissenschaftlern am Max-Planck-Institut für Biochemie in Martinsried gelungen, insgesamt 4399 verschiedene Proteine der Bäckerhefe zu identifizieren. Die Forscher zeigten zudem, wie sich das Protein-Set der Hefe im Laufe ihres Lebenszyklus ändert. In einer weiteren Veröffentlichung stellen die Wissenschaftler eine neue Methode vor, mit der sich Proteine, die von einem Gen reguliert werden, deutlich besser finden lassen als mit der herkömmlichen Micro-Array-Methode. (Nature Online, 29. September 2008 und Molecular Cell, 5. September 2008)
Seit der ersten Veröffentlichung des menschlichen Genoms im Jahr 2001 weiß man, dass nicht allein die Analyse von Genen für das Verständnis von Leben wichtig ist.
Wissenschaftler versuchen vielmehr, die Gesamtheit der Proteine eines Organismus – das Proteom – zu identifizieren. Dies ist der gesamte Proteinbestand in einem Lebewesen.
Die Forscher erhoffen sich so, die Entwicklung und Lebensfähigkeit von Organismen sowie die Entstehung von Krankheiten besser zu verstehen. Auf dieser Grundlage sollen dann spezifische Therapeutika entwickelt werden.
Bäckerhefe als ideales Versuchsobjekt
Neben Fruchtfliege, Zebrafisch und Maus zählt auch die Hefe zu den Modellorganismen der Zellbiologen. Wie menschliche Zellen besitzen auch Hefen doppelte Chromosomensätze. Da ihre Proteine denen in Säugerzellen sehr ähnlich sind, wird die Hefe in der Biotechnologie zur Produktion von Wirkstoffen eingesetzt.
Während ihres vegetativen Wachstums enthalten die Hefezellen, genau wie menschliche Ei- und Samenzellen, nur einen einfachen – haploiden – Chromosomensatz: Die Zellen besitzen dann von jedem Chromosomentyp jeweils nur ein Exemplar.
Hefe kann sich aber auch sexuell vermehren und besitzt in dieser Vermehrungsphase einen doppelten – diploiden – Chromosomensatz.
Als erstes Genom eines Eukaryonten wurde das Hefegenom sequenziert. Nunmehr ist auch das Proteom der Hefe vollständig aufgeklärt worden.
Identische Gene, aber unterschiedliches Protein-Set
Die Wissenschaftler verglichen das Proteom haploider mit dem diploider Hefezellen. Sie fanden heraus, dass die Proteine des Pheromonsignalweges verstärkt in den haploiden Zellen vorliegen. In den diploiden Zellen tauchten diese chemischen Botenstoffe jedoch nicht auf. Die Zellen benötigen die Pheromone zur Paarung. Sinnvollerweise setzt die Hefe nur im paarungsbereiten Stadium Energie für die Pheromon-Bildung ein.
Die Max-Planck-Forscher spürten noch weitere Unterschiede im Proteom der beiden Zellstadien auf. Der Vergleich ist deshalb interessant, weil beide Zellen identische Gene besitzen, jedoch für ihre unterschiedliche Lebensweise und Funktion ein völlig unterschiedliches Protein-Set benötigen. Auch Leber- und Muskelzellen eines Menschen weisen eine gleiche genetische Zusammensetzung auf, obwohl sie völlig unterschiedliche Funktionen im Körper ausüben.
„Wir haben bewiesen, dass es mit unserem Forschungsansatz möglich ist, dass gesamte Proteom eines Organismus aufzuklären. Jetzt müssen wir die Methoden verfeinern und unsere Analysen ausdehnen, um weitere Proteome zu entschlüsseln“, sagt Matthias Mann.
Der Vergleich des Proteoms der beiden Wachstumsstadien – diploid und haploid – eröffnet neue Einblicke in die Regulation der Zell-Differenzierung, die Entwicklung von Geweben und die Entstehung von Krankheiten.
Schwere und leichte Proteine
Wie bei der Bestimmung des Hefe-Proteoms, nutzten die Max-Planck-Forscher an der Fruchtfliege Drosophila die von Matthias Mann entwickelte sogenannte SILAC-Technik (Stable Isotope Labeling by Amino Acid by Cell Culture), die funktionale Proteom-Analysen ermöglicht.
Dabei werden einzelne Aminosäuren der Proteine im Nährmedium der Zellen oder im Futter der Modellorganismen mit schwereren Isotopen markiert. Dadurch ist es möglich, das Proteom von Zellen oder Organismen zu analysieren und zu vergleichen. Denn aus dem Verhältnis von schweren zu leichten Proteinen, die gebildet werden, lässt sich das Proteom quantitativ mit außergewöhnlicher Genauigkeit bestimmen.
Zugleich mit dieser quantitativen Analyse des Drosophila-Proteoms hemmten die Martinsrieder Wissenschaftler und ihre Kollegen von der Universität München die „Übersetzung“ bestimmter Gene in Proteine.
Dadurch konnten sie beispielsweise die Regulation eines Gens namens ISWI untersuchen, das bei allen höheren Organismen eine wichtige Rolle bei der Differenzierung von Eizellen spielt.
Veränderungen im Proteinmuster
Die Forscher identifizierten 4100 Proteine in den Zellen der Fruchtfliege. Nachdem sie das Gen ISWI gezielt ausgeschaltet hatten, veränderte sich die Synthese von mehr als 300 Proteinen. Diese Veränderungen im Proteinmuster korrelierten jedoch kaum mit denen, die mit der herkömmlichen Micro-Array-Methode gefunden wurden. „Die Ergebnisse zeigen, dass unser Verfahren besser geeignet ist, die Regulation einzelner Gene in verschiedenen Organismen wie etwa Fruchtfliege, Maus oder Mensch zu verstehen“, so Matthias Mann.
Die Wissenschaftler wollen nun auch bereits vorhandene Daten nutzen. In Kombination mit neuen Techniken könnten die Wissenschaftler so vielleicht ihrem Ziel näher kommen, das menschliche Proteom vollständig zu erfassen.
Hintergrund
Unter Leitung von Matthias Mann hat sich erst kürzlich ein internationales Konsortium zum größten EU-Forschungsprojekt auf dem Gebiet der Proteom-Forschung zusammengefunden.
Das Projekt PROSPECTS (Proteomics specification in Time and Space) wird durch das siebte Europäische Forschungsrahmenprogramm in den nächsten fünf Jahren mit insgesamt knapp 12 Millionen Euro gefördert. Das PROSPECTS-Konsortium plant, aktuelle Proteom-Forschung durch Entwicklung neuer Technologien und Instrumente voranzubringen. PROSPECTS soll für die biomedizinische Forschung neue Einblicke in die zelluläre Funktion von Proteinen und ihre Veränderung bei Krankheiten ermöglichen.
Originalveröffentlichung:
Lyris M. F. de Godoy, Jesper V. Olsen, Jürgen Cox, Michael L. Nielsen, Nina C. Hubner, Florian Fröhlich,Tobias C. Walther & Matthias Mann
„Comprehensive mass-spectrometry-based proteome quantification of haploid versus diploid yeast“
Nature Advance Online Publication 29. September 2008
Tiziana Bonaldi, Tobias Straub, Jürgen Cox, Chanchal Kumar, Peter B. Becker, Matthias Mann
„Combined use of RNAi and quantitative proteomics to study gene function in Drosophila“
Media Contact
Weitere Informationen:
http://www.mpg.deAlle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Diamantstaub leuchtet hell in Magnetresonanztomographie
Mögliche Alternative zum weit verbreiteten Kontrastmittel Gadolinium. Eine unerwartete Entdeckung machte eine Wissenschaftlerin des Max-Planck-Instituts für Intelligente Systeme in Stuttgart: Nanometerkleine Diamantpartikel, die eigentlich für einen ganz anderen Zweck bestimmt…
Neue Spule für 7-Tesla MRT | Kopf und Hals gleichzeitig darstellen
Die Magnetresonanztomographie (MRT) ermöglicht detaillierte Einblicke in den Körper. Vor allem die Ultrahochfeld-Bildgebung mit Magnetfeldstärken von 7 Tesla und höher macht feinste anatomische Strukturen und funktionelle Prozesse sichtbar. Doch alleine…

Hybrid-Energiespeichersystem für moderne Energienetze
Projekt HyFlow: Leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem für moderne Energienetze. In drei Jahren Forschungsarbeit hat das Konsortium des EU-Projekts HyFlow ein extrem leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem entwickelt, das einen…