Need for Speed: Bewegungsmessungen im Zellkern mit Hochgeschwindigkeit

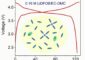
Mikroskopie-Aufnahmen nach Bleichen der Fluoreszenz in einem kreisförmigen oder rechteckigen Bereich. Die dunklen „Schatten“ stellen gebleichte Proteine dar, die sich während der Bildaufzeichnung bewegt hatten.<br><br>Abbildungsnachweis: Fabian Erdel und Karsten Rippe<br>
Mit einer neuen Messmethode ist es Heidelberger Wissenschaftlern gelungen, Wechselwirkungen zwischen Proteinen und DNA im Zellkern mit einer Zeitauflösung von einer tausendstel Sekunde zu verfolgen.
Gemessen werden konnte damit in lebenden Zellen die Bindung hochspezialisierter Proteinkomplexe, die die räumliche Struktur der Erbinformation gezielt verändern und so das Auslesen der DNA-Information kontrollieren.
Die Arbeiten von Privatdozent Dr. Karsten Rippe und seinem Team wurden am BioQuant-Zentrum der Universität Heidelberg und am Deutschen Krebsforschungszentrum durchgeführt. Ihre Forschungen haben gezeigt, dass die Positionierung von Nukleosomen – dies sind Komplexe aus DNA-Strängen und speziellen Proteinen – ein genau geregelter molekularer Prozess ist. Eine Fehlregulation kann mit verschiedenen Krebsarten in Verbindung gebracht werden. Die Ergebnisse dieser Untersuchungen wurden in der Fachzeitschrift PNAS veröffentlicht.
Im menschlichen Genom sind die DNA-Stränge um bestimmte Verpackungsproteine – die Histone – gewickelt. Zwischen diesen als Nukleosomen bezeichneten Komplexen befinden sich DNA-Bereiche, die frei von Histonen sind und die die Nukleosomen wie auf einer Perlenkette verbinden. „Die Aktivierung eines Gens erfordert frei zugängliche DNA. Wird die entsprechende DNA im Nukleosom verdeckt, ist das Gen oft abgeschaltet“, erläutert Dr. Rippe. „So geben die Positionen der Nukleosomen das Auslesemuster der DNA-Sequenz vor. Die freie DNA zwischen zwei Nukleosomen ist leichter zugänglich als die DNA-Bereiche in einem Nukleosom.“ Molekulare Maschinen, sogenannte Chromatin Remodeler, können unter Energieverbrauch Nukleosomen entlang der DNA-Kette verschieben. Dadurch etablieren sie Auslesemuster, die zusammen mit anderen Faktoren das aktive DNA-Programm der Zelle bestimmen.
Die Wissenschaftler um Karsten Rippe untersuchen mit Hilfe von Fluoreszenzmikroskopie-Verfahren, wie die Chromatin Remodeler das Auslesen der Erbinformation steuern. Dabei konnten sie messen, dass in einer menschlichen Zelle die meisten der rund eine Million Chromatin Remodeler immer wieder nur kurz an ein Nukleosom binden, um zu testen, ob alle rund 30 Millionen Nukleosomen in der richtigen Position sind. Um die Arbeitsweise dieser molekularen Maschinen verstehen zu können, war eine neue Messmethode erforderlich.
„Wir mussten kurze Bindungsereignisse mit einer Auflösung von einer tausendstel Sekunde aufzeichnen und gleichzeitig auch die selten auftretenden Reaktionen mit einer Bindungszeit von mehreren Sekunden oder gar Minuten nachweisen“, erläutert Karsten Rippe. Aus einer Idee des Doktoranden Fabian Erdel entstand dafür die Methode 3PEA für „Pixel-wise Photobleaching Profile Evolution Analysis“, die für Messungen in lebenden Zellen eingesetzt werden kann.
In seinen Experimenten brachte Fabian Erdel die künstliche Fluoreszenzmarkierung der Chromatin Remodeler durch Laserbestrahlung zum Erlöschen. Dabei beobachtete er, dass durch die so „gebleichten“ Proteine ein „Schatten“ entstand, wenn sie sich während der Aufzeichnung des Bildes bewegten. Die Form dieses Schattens hing davon ab, wie stark die Bewegung der Chromatin Remodeler durch Bindung an Nukleosomen verlangsamt wurde. „Es war nicht einfach, das Schattenbild in die Bindungsdauer umzurechnen, aber die Mühe hat sich gelohnt: Unsere Methode hat neue vielversprechende Anwendungsmöglichkeiten, weil wir damit die Bindung von Proteinen in lebenden Zellen sehr schnell und sehr genau messen können“, erklärt Fabian Erdel.
Mithilfe von 3PEA-Messungen haben die Forscher gezeigt, dass sich ein einzelner Chromatin Remodeler innerhalb nur einer Sekunde fast durch den gesamten Zellkern bewegt und dabei über 300 Nukleosomen testet – meistens ohne aktiv zu werden. Nur manchmal bindet die molekulare Maschine für einige Sekunden oder sogar Minuten an ein Nukleosom, um es dann auf der DNA zu verschieben. In einem nächsten Schritt will die Forschungsgruppe von Dr. Rippe die Signale entziffern, die die Chromatin Remodeler an bestimmten Stellen des Genoms aktivieren.
Informationen im Internet sind unter http://malone.bioquant.uni-heidelberg.de zu finden.
Originalveröffentlichung:
F. Erdel, K. Rippe: Quantifying transient binding of ISWI chromatin remodelers in living cells by pixel-wise photobleaching profile evolution analysis, PNAS, 20 November 2012, vol. 109, no. 47, E3221-3230 (online 5 November 2012, doi:10.1073/pnas.1209579109).
Kontakt:
Privatdozent Dr. Karsten Rippe
BioQuant, Telefon (06221) 54-51376
karsten.rippe@bioquant.uni-heidelberg.de
Universität Heidelberg
Kommunikation und Marketing
Pressestelle, Telefon (06221) 54-2311
presse@rektorat.uni-heidelberg.de
Media Contact
Alle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Diamantstaub leuchtet hell in Magnetresonanztomographie
Mögliche Alternative zum weit verbreiteten Kontrastmittel Gadolinium. Eine unerwartete Entdeckung machte eine Wissenschaftlerin des Max-Planck-Instituts für Intelligente Systeme in Stuttgart: Nanometerkleine Diamantpartikel, die eigentlich für einen ganz anderen Zweck bestimmt…
Neue Spule für 7-Tesla MRT | Kopf und Hals gleichzeitig darstellen
Die Magnetresonanztomographie (MRT) ermöglicht detaillierte Einblicke in den Körper. Vor allem die Ultrahochfeld-Bildgebung mit Magnetfeldstärken von 7 Tesla und höher macht feinste anatomische Strukturen und funktionelle Prozesse sichtbar. Doch alleine…

Hybrid-Energiespeichersystem für moderne Energienetze
Projekt HyFlow: Leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem für moderne Energienetze. In drei Jahren Forschungsarbeit hat das Konsortium des EU-Projekts HyFlow ein extrem leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem entwickelt, das einen…