Ein Barcode zum Schreddern von Abfall-RNA
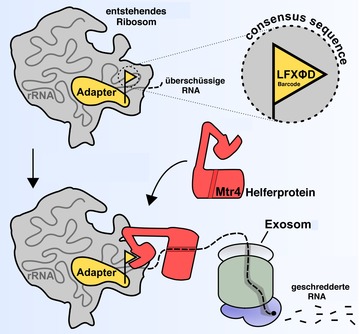
Die schematische Darstellung zeigt, wie das Exosom die überschüssige RNA am entstehenden Ribosom degradiert. Daran beteiligt ist ein Adapterprotein am Prä-Ribosom, das eine Konsensus Erkennungssequenz (Barcode) trägt, an die das Mtr4 Helferprotein binden kann. Dadurch wird das Exosom an die Ziel-RNA dirigiert. Copyright: Universität Heidelberg
Eine wachsende, sich teilende Zelle verwendet einen Großteil ihres Energiehaushalts für die Herstellung ihrer „Proteinfabriken“, der Ribosomen. Eine wichtige Rolle bei deren „Montage“ spielt das Exosom – eine molekulare Schredder-Maschine, die überschüssige Ribonukleinsäure (RNA) abbaut.
Wissenschaftler um Prof. Dr. Ed Hurt vom Biochemie-Zentrum der Universität Heidelberg (BZH) konnten nun aufklären, wie das Exosom seine Ziel-RNA erkennt: Sie identifizierten ein spezifisches Erkennungssignal, das – vergleichbar mit einer Postleitzahl oder einem Barcode – dem Exosom den Aufenthaltsort der zu entfernenden RNA signalisiert. Die Forschungsergebnisse wurden in der Fachzeitschrift „Cell“ veröffentlicht.
Die Herstellung von Ribosomen ist ein äußerst komplizierter Prozess, der einem strengen Bauplan mit zahlreichen Qualitätskontrollen unterliegt, wie Prof. Hurt erläutert. Die Proteinfabriken bestehen aus zahlreichen ribosomalen Proteinen (r-Proteinen) und ribosomaler Ribonukleinsäure (rRNA).
Um die r-Proteine und die verschiedenen rRNAs korrekt zusammenzusetzen, werden in eukaryotischen Zellen unter anderem mehr als 200 Helfer-Proteine, sogenannte Ribosomen-Biogenese-Faktoren, benötigt. Drei der insgesamt vier verschiedenen rRNAs werden aus einer großen Vorläufer-RNA hergestellt.
Sie müssen zu bestimmten Zeitpunkten des Herstellungsprozesses „zurechtgeschnitten“ werden, wobei nicht mehr benötigte Bereiche beseitigt werden. „Da diese Vorgänge unumkehrbar sind, ist dafür eine besondere Kontrolle nötig“, erklärt Ed Hurt.
Ein wichtiger Teil dieser überschüssigen ribosomalen RNA wird durch das Exosom abgebaut. Diese molekulare Maschine besteht aus mehreren Proteinuntereinheiten. Alle zusammen bilden eine Struktur ähnlich einem Fass, durch das die RNA geschleust wird. Am Ende dieses Kanals sitzt die Untereinheit des Exosoms, die die RNA in ihre Einzelbausteine zerlegt, die sogenannten Nukleotide. „Dieser Prozess ist bereits recht gut beschrieben – aber bisher haben wir noch nicht verstanden, wie das Exosom seine Ziel-RNA erkennt und von den zahlreichen RNA-Molekülen unterscheidet, die unversehrt bleiben müssen“, erläutert Ed Hurt.
Aus der Forschungsgruppe von Prof. Hurt konnten nun Matthias Thoms und Dr. Emma Thomson zwei Ribosomen-Biogenese-Faktoren ausmachen, die sich in räumlicher Nähe zur Ziel-RNA auf dem unfertigen Ribosom befinden und das Exosom zu seiner Ziel-RNA leiten. Beide Protein-Faktoren agieren während der Ribosomenherstellung, was aber räumlich und zeitlich voneinander getrennt abläuft.
Obwohl beide Proteine unterschiedlich aufgebaut sind, besitzen sie eine entscheidende Gemeinsamkeit: Die Heidelberger Wissenschaftler entdeckten bei beiden eine kurze Signalsequenz, die mit einem Barcode oder einer Postleitzahl vergleichbar ist. Durch dieses Erkennungssignal wird ein Helfer-Protein des Exosoms rekrutiert, das die Ziel-RNA präsentiert. Das Exosom kann dann mit seiner Aufgabe beginnen und die nicht mehr benötigte RNA schreddern.
Die Wissenschaftler der Hurt-Gruppe wollen nun weitere Proteine mit der beschriebenen Signalsequenz identifizieren, um herauszufinden, wie das Exosom ein derart breites Spektrum unterschiedlicher RNA eliminieren kann. „Das Exosom ist ein universaler Proteinkomplex, der für die ‚RNA-Homöostase‘, also das Gleichgewicht zwischen RNA-Aufbau und -abbau, in allen Zellen essentiell ist. Wir nehmen an, dass diese Art der Ziel-RNA-Erkennung, wie wir sie entdeckt haben, einen allgemeinen Mechanismus für die Regulation des Exosoms darstellt“, erklärt Prof. Hurt.
„Daher könnten unsere Erkenntnisse auch zu einem besseren molekularen Verständnis von Krankheiten führen, bei denen Defekte im Exosom beziehungsweise deren Helferproteinen nachgewiesen wurden.“ So seien Mutationen in Bestandteilen des Exosoms bekannt, was zu Autoimmunkrankheiten oder multiplen Myelomen beim Menschen führen könne.
Originalpublikation:
M. Thoms, E. Thomson, J. Baßler, M. Gnädig, S. Griesel and E. Hurt: The Exosome Is Recruited to RNA Substrates Through Specific Adapter Proteins. Cell 162, 1029-1038 (27 August 2015), doi: 10.1016/j.cell.2015.07.060
Kontakt:
Prof. Dr. Ed Hurt
Biochemie-Zentrum der Universität Heidelberg
Telefon (06221) 54-4781, -4173
ed.hurt@bzh.uni-heidelberg.de
Kommunikation und Marketing
Pressestelle, Telefon (06221) 54-2311
presse@rektorat.uni-heidelberg.de
http://www.uni-heidelberg.de//zentral/bzh/hurt
http://www.bzh.uni-heidelberg.de
Media Contact
Weitere Informationen:
http://www.uni-heidelberg.deAlle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Neue universelle lichtbasierte Technik zur Kontrolle der Talpolarisation
Ein internationales Forscherteam berichtet in Nature über eine neue Methode, mit der zum ersten Mal die Talpolarisation in zentrosymmetrischen Bulk-Materialien auf eine nicht materialspezifische Weise erreicht wird. Diese „universelle Technik“…

Tumorzellen hebeln das Immunsystem früh aus
Neu entdeckter Mechanismus könnte Krebs-Immuntherapien deutlich verbessern. Tumore verhindern aktiv, dass sich Immunantworten durch sogenannte zytotoxische T-Zellen bilden, die den Krebs bekämpfen könnten. Wie das genau geschieht, beschreiben jetzt erstmals…

Immunzellen in den Startlöchern: „Allzeit bereit“ ist harte Arbeit
Wenn Krankheitserreger in den Körper eindringen, muss das Immunsystem sofort reagieren und eine Infektion verhindern oder eindämmen. Doch wie halten sich unsere Abwehrzellen bereit, wenn kein Angreifer in Sicht ist?…





















