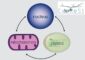
Die Nummer Eins aller Kolibakterien genetisch enträtselt

Rasterelektronenmikroskopische Aufnahme von Zellen des E. coli Typstamms DSM 30083T (Manfred Rohde, Helmholtz-Zentrum für Infektionsforschung; Christine Rohde, Leibniz-Institut DSMZ
Das Darmbakterium Escherichia coli ist der am besten untersuchte Modellorganismus der Lebenswissenschaften. Allerdings blieb der Referenzorganismus dieser Art, der sogenannte „Typstamm“, bisher von der mikrobiellen Genomik unberücksichtigt. Als Teil des „Genomic Encyclopedia of Bacteria and Archaea“-Projekts (GEBA) wurde jetzt das Erbgut des Typstamms DSM 30083T sequenziert und mit nahen Verwandten verglichen.
Die Untersuchungen erlauben nicht nur eine ganz neue Sicht auf die Vielzahl von medizinisch und biotechnologisch relevanten E. coli-Stämmen, einschließlich der pathogenen EHEC-Erreger und Shigellen, sondern lieferten auch eine allgemein anwendbare Methode, um bakterielle Arten in Unterarten einzuteilen. Die Forschungsarbeiten wurden am Leibniz-Institut DSMZ–Deutsche Sammlung von Mikroorganismen und Zellkulturen GmbH in Braunschweig und am Joint Genome Institute in Kalifornien durchgeführt.
Das Darmbakterium Escherichia (E.) coli, das „Haustier“ der Mikrobiologen und Biotechnologen, hat eine bewegte Geschichte. Vom Bakteriologen Theodor Escherich im Jahre 1886 erstmals unter dem Namen „Bacterium coli commune“ beschrieben, ging das ursprüngliche Isolat in den frühen 1920er Jahren verloren. Erst 1941 wurde es von Fritz Kauffmann am Staatlichen Serum-Institut in Kopenhagen erneut isoliert, wissenschaftlich beschrieben und in mehreren mikrobiellen Stammsammlungen hinterlegt. Heute ist E. coli der wohl am besten erforschte Mikroorganismus und ein wesentlicher Indikatorkeim für Badegewässer und das Trinkwasser.
„Es mutet sicher eigenartig an, dass gerade die ‚Nummer Eins‘, der Typstamm des Bakteriums, dem als Modellorganismus ganze Kongresse gewidmet sind, erst jetzt in seiner Sequenz vollständig aufgeklärt wird“, vermerkt Dr. Christine Rohde, Leiterin der E.coli-Stammsammlung an der DSMZ in Braunschweig. „Zuerst sequenzierte man vorrangig die Genome von pathogenen E. coli-Vertretern oder genetisch veränderten biotechnologisch relevanten Stämmen. Außerdem arbeiten Mediziner und Hygieniker in der Praxis eher mit Serotypen, die sich schnell durch einen Antikörpertest bestimmen lassen, um verschiedene E. coli-Stämme zu unterscheiden.“
Dr. Markus Göker, Bioinformatiker an der DSMZ führt weiter aus: „Vollständige Bakteriengenome sind für die humane Diagnostik, die Biotechnologie und die Suche nach antimikrobiellen Wirkstoffen von fundamentaler Bedeutung. Das gilt gerade heute, nachdem sich einige E. coli-Stämme zu gefährlichen Problemkeimen wie EHEC oder EAHEC entwickelt haben. Der E. coli-Typstamm wurde im Rahmen des GEBA-Projekts sequenziert, dessen Fokus auf Typstämmen mit außergewöhnlicher Physiologie oder einer Schlüsselstellung im Stammbaum liegt. Es handelt sich um den einzigen Mikroorganismus im Projekt, der aufgrund seiner Bedeutung als Modellorganismus aufgenommen wurde.“
Pathogenes Potential im Erbgut
Der E. coli-Typstamm unterscheidet sich physiologisch und genomisch stark vom ungefährlichen Laborstamm K-12. „Aufgrund seines Serotyps wurde der Typstamm in die biologische Sicherheitsstufe 2 eingeordnet, und die Genomsequenzierung bestätigte sein pathogenes Potential“, berichtet Dr. Jörn Petersen, Plasmidexperte an der DSMZ. „Im Gegensatz zum Laborstamm K-12 enthält der E. coli-Typstamm in seinem 5.038.133 Basenpaare langen Genom ein zusätzliches ringförmiges Plasmid mit 131.289 Basenpaaren, das eine Sequenzidentität von 99 Prozent zu bekannten Plasmiden aus pathogenen E. coli-Isolaten aufweist. Diese verursachen z.B. die ‚Colibacillose‘ bei Geflügel und Hirnhautentzündungen bei Neugeborenen, wobei das horizontal übertragbare Plasmid für die Virulenz dieser Stämme verantwortlich ist“, betont Jörn Petersen.
Moderne Stammbaumanalyse am Computer
Mit Hilfe der kompletten Genomsequenz des E. coli-Typstamms ist es den Forschern in Braunschweig gelungen, mit modernen taxonomischen Methoden zu prüfen, ob die große Anzahl der bisher sequenzierten E. coli-Isolate wirklich zur gleichen Art gehören. „Wir haben dazu über 250 E. coli-Stämme analysiert und auch ihre in der Literatur vorgeschlagene taxonomische Einteilung in Untergruppen, die ‚Phylotypen‘, überprüft. Die bioinformatische Analyse wurde mit der sogenannten „GGDC-Methode“ nach dem neuesten Stand der Technik durchgeführt. Diese Methode ist analog zur klassischen DNA-DNA-Hybridisierung im Labor, aber wesentlich exakter“, erklärt Markus Göker.
Die Ergebnisse belegen die Zugehörigkeit aller sequenzierten E. coli-Stämme zu einer Art. Eine neue Erkenntnis ist, dass E. coli in mehrere Unterarten aufgeteilt werden müsste, von denen eine sämtliche Stämme der Gattung Shigella enthält, bekannt als Erreger der Shigellenruhr. „Shigella ist jedoch ein aus medizin-historischen Gründen etablierter Name, so dass wir nicht auf taxonomische Änderungen abzielten“, fügt Markus Göker hinzu. „Viel wichtiger ist, dass sich die an E. coli erprobten Verfahren nutzen lassen, um generell bakterielle Arten in Unterarten einzuteilen.“
Originalpublikation:
Meier-Kolthoff JP et al. (2014). Complete genome sequence of DSM 30083T, the type strain (U5/41T) of Escherichia coli, and a proposal for delineating subspecies in microbial taxonomy. Stand Genomic Sci 9: 2 (http://dx.doi.org/10.1186/1944-3277-9-2)
Weiterführende Literatur:
Peigne C et al. (2009). The plasmid of Escherichia coli strain S88 (O45:K1:H7) that causes neonatal meningitis is closely related to avian pathogenic E. coli plasmids and is associated with high-level bacteremia in a neonatal rat meningitis model. Infect Immun 77: 2272-2284 (http://dx.doi.org/10.1128/IAI.01333-08).
Meier-Kolthoff JP et al. (2013). Genome sequence-based species delimitation with confidence intervals and improved distance functions. BMC Bioinformatics 14: 60 (http://dx.doi.org/10.1186/1471-2105-14-60).
GGDC-Methode:
Der GGDC (Genome-to-Genome Distance Calculator) ist ein bioinformatischer Ansatz, um Distanzen und Ähnlichkeiten zwischen Genomsequenzen zu berechnen. Diese können für die Erstellung von Stammbäumen verwendet werden, oder aber umgerechnet werden, um die traditionelle DNA-DNA-Hybridisierungs-Methode zu ersetzen. Das erlaubt es, Bakterien in Arten (und als Ergebnis der E. coli-Studie auch in Unterarten) einzuteilen. GGDC steht als Web-Service unter folgendem Link zur Verfügung: http://ggdc.dsmz.de.
GEBA:
Das Projekt GEBA (Genomic Encyclopedia of Bacteria and Archaea) und seine Folgeprojekte haben zum Ziel, systematisch die Lücken in den mikrobiellen Bereichen des Baums des Lebens mittels Genomsequenzierung zu füllen. Die DSMZ führt dieses Projekt in enger Kooperation mit dem Joint Genome Institute in Kalifornien durch. Leiter der Projekte an der DSMZ ist Dr. Markus Göker. Die annotierten Genome sind im Portal „GenBank“ (http://www.ncbi.nlm.nih.gov/genbank/) hinterlegt und interaktiv zugänglich unter https://img.jgi.doe.gov/cgi-bin/w/main.cgi.
Hinweise für die Medien:
Sie finden die Pressemitteilung in deutscher und englischer Sprache sowie Pressebilder auf unserer Webseite wwww.dsmz.de. (Link: http://www.dsmz.de/de/start/details/entry/the-number-one-of-en.html)
Über das Leibniz-Institut DSMZ
Das Leibniz-Institut DSMZ – Deutsche Sammlung von Mikroorganismen und Zellkulturen GmbH ist eine Einrichtung der Leibniz-Gemeinschaft und mit seinen umfangreichen wissenschaftlichen Services und einem breiten Spektrum an biologischen Materialien seit Jahrzehnten weltweiter Partner für Forschung und Industrie. Als einem der größten biologischen Ressourcenzentren seiner Art wurde der DSMZ die Übereinstimmung mit dem weltweit gültigen Qualitätsstandard ISO 9001:2008 bestätigt. Als Patenthinterlegungsstelle bietet die DSMZ die bundesweit einzigartige Möglichkeit, biologisches Material nach den Anforderungen des Budapester Vertrags aufzunehmen. Neben dem wissenschaftlichen Service bildet die sammlungsbezogene Forschung das zweite Standbein der DSMZ. Die Sammlung mit Sitz in Braunschweig existiert seit 42 Jahren und beherbergt mehr als 48.000 Kulturen und Biomaterialien. Die DSMZ ist die vielfältigste Sammlung weltweit: neben Pilzen, Hefen, Bakterien und Archaea werden dort auch menschliche und tierische Zellkulturen sowie Pflanzenviren und pflanzliche Zellkulturen erforscht und archiviert. www.dsmz.de
Die Leibniz-Gemeinschaft
Die Leibniz-Gemeinschaft verbindet 89 selbständige Forschungseinrichtungen. Deren Ausrichtung reicht von den Natur-, Ingenieur- und Umweltwissenschaften über die Wirtschafts-, Raum- und Sozialwissenschaften bis zu den Geisteswissenschaften. Leibniz-Institute bearbeiten gesellschaftlich, ökonomisch und ökologisch relevante Fragestellungen. Sie betreiben erkenntnis- und anwendungsorientierte Grundlagenforschung. Sie unterhalten wissenschaftliche Infrastrukturen und bieten forschungsbasierte Dienstleistungen an. Die Leibniz-Gemeinschaft setzt Schwerpunkte im Wissenstransfer in Richtung Politik, Wissenschaft, Wirtschaft und Öffentlichkeit. Leibniz-Institute pflegen intensive Kooperationen mit den Hochschulen – u.a. in Form der Leibniz-WissenschaftsCampi, mit der Industrie und anderen Partnern im In- und Ausland. Sie unterliegen einem maßstabsetzenden transparenten und unabhängigen Begutachtungsverfahren. Aufgrund ihrer gesamtstaatlichen Bedeutung fördern Bund und Länder die Institute der Leibniz-Gemeinschaft gemeinsam. Die Leibniz-Institute beschäftigen rund 17.500 Personen, darunter 8.800 Wissenschaftlerinnen und Wissenschaftler. Der Gesamtetat der Institute liegt bei 1,5 Milliarden Euro.
Pressekontakt:
Susanne Thiele
Leiterin Presse und Kommunikation
Leibniz-Institut DSMZ-Deutsche Sammlung von Mikroorganismen und Zellkulturen GmbH
Inhoffenstraße 7 B
38124 Braunschweig, Deutschland
Tel. ++49531-2616-300
Fax ++49531-2616-418
mobil ++49151-1409-2514
susanne.thiele@dsmz.de
http://www.dsmz.de
Social Web:
www.facebook.com/Leibniz.DSMZ
www.twitter.com/Leibniz_DSMZ_en
Webseite – wwww.dsmz.de
Link zur Pressemitteilung – http://www.dsmz.de/de/start/details/entry/the-number-one-of-en.html
Media Contact
Alle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Neuartiges Material für nachhaltiges Bauen
Innovativer Werkstoff für eine energieeffiziente Architektur: Forschende des Karlsruher Instituts für Technologie (KIT) stellen in der aktuellen Ausgabe der Fachzeitschrift Nature Communications ein polymerbasiertes Material mit besonderen Eigenschaften vor. Das…

Neues Antibiotikum gegen Erreger der Flussblindheit und Lymphatischen Filariose
Prof. Achim Hoerauf, Direktor des Instituts für Medizinische Mikrobiologie, Immunologie und Parasitologie des Universitätsklinikums Bonn (UKB), und seinem Team ist es in Kollaboration mit der Abteilung Pharmazeutische Technologie und Biopharmazie…
Evolutionäre Genomik: Folgen biodiverser Fortpflanzungssysteme
Die Deutsche Forschungsgemeinschaft (DFG) fördert die Einrichtung eines neuen Graduiertenkollegs (GRK) in der Biologie an der Universität Göttingen. Das GRK mit dem Titel „Evolutionary Genomics: Consequences of Biodiverse Reproductive Systems…