Bakterienprofile auf Knopfdruck

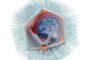
Die neue Software Traitar soll verschiedene Stämme des Erregers Pseudomonas aeruginosa auf neue Resistenzmechanismen gegen Antibiotika untersuchen. HZI/Manfred Rohde
Moderne Sequenziertechniken machen es heutzutage möglich, das gesamte Erbmaterial – das Genom – eines Bakteriums in kurzer Zeit zu entschlüsseln. Das Ergebnis ist eine riesige Datenmenge, in der sich Tausende Gene verbergen. Welche Eigenschaften diese Gene dem Bakterium verleihen, müssen Wissenschaftler jedoch aufwendig analysieren.
Bioinformatiker des Braunschweig Integrated Centre of Systems Biology (BRICS), einer gemeinsamen Einrichtung des Helmholtz-Zentrums für Infektionsforschung (HZI) und der Technischen Universität Braunschweig, haben nun eine Software entwickelt, die aus Genomdaten insgesamt 67 Eigenschaften des Bakteriums vorhersagen kann.
Zu diesen Merkmalen gehören zum Beispiel bevorzugte Nahrungsquellen, bestimmte Resistenzen oder die Beweglichkeit der Bakterien. Die Software mit dem Namen „Traitar“ haben die Wissenschaftler online frei zur Verfügung gestellt und sie kürzlich im Fachjournal mSystems beschrieben.
Die physiologischen Eigenschaften eines Organismus sind in kodierter Form in seiner Erbinformation, dem Genom, festgelegt. Es enthält Baupläne für eine Vielzahl von Proteinen, die allen Funktionen in den Zellen des Organismus zugrunde liegen und so auch seine charakteristischen Merkmale bestimmen.
Gerade bei krankheitserregenden Bakterien ist es wichtig, ihre spezifischen Merkmale zu identifizieren, um sie genauer erforschen und zum Beispiel bei einer Infektion eine passende medikamentöse Therapie entwickeln zu können. Dazu müssen die Forscher aber unter anderem wissen, welche Nahrungsquellen die Erreger nutzen, ob sie Sauerstoff benötigen oder ob sie Resistenzen gegen Antibiotika aufweisen. Um diese Informationen zu erhalten, bedarf es besonders bei noch nicht charakterisierten Bakterienstämmen oft langwieriger Laborexperimente.
Ein erster und mittlerweile schneller Schritt in diesem Prozess ist das Entschlüsseln des Genoms der Bakterien. Doch die Auswertung der erhaltenen Daten ist dafür umso aufwendiger. Die neue Software „Traitar“, die die Braunschweiger Bioinformatiker entwickelt haben, erleichtert den Wissenschaftlern nun die Charakterisierung von Bakterienstämmen wesentlich: Anhand der Genomdaten leitet das Programm in wenigen Minuten eine ganze Reihe relevanter Eigenschaften ab.
„Die aktuelle Version von Traitar testet die bakteriellen Genomdaten auf 67 verschiedene Phänotypen, also Eigenschaften, des jeweiligen Bakteriums“, sagt Aaron Weimann, der in der Abteilung „Bioinformatik der Infektionsforschung“ von Prof. Alice McHardy promoviert und mit dem Team die neue Software entwickelt hat. „Bakterien besitzen meist zwischen 3000 und 6000 Gene, die für die Ausprägung ihrer Eigenschaften verantwortlich sind. Aus den genetischen Daten identifiziert Traitar mittels maschineller Lernverfahren bestimmte Proteinfamilien und zeigt an, zu welchem Phänotyp diese Proteine führen.“
Darüber hinaus gibt die Software auch detailliert aus, welche Proteine oder Proteinfamilien zu den vorhergesagten Eigenschaften führen. Der Name der Software ist vom englischen Wort „trait“ – zu Deutsch Eigenschaft oder Merkmal – abgeleitet. Traitar ist im Internet frei verfügbar unter https://github.com/hzi-bifo/traitar. Dort lässt sich die Software kostenlos herunterladen und kann ohne weitere Programmierkenntnisse genutzt werden. Kleine Datenmengen können auch direkt per Online-Tool analysiert werden unter http://research.bifo.helmholtz-hzi.de/webapps/wa-webservice/pipe.php?pr=traitar.
Für die Entwicklung von Traitar haben Weimann und seine Kollegen zunächst in der Literatur und in Datenbanken Informationen über Phänotypen von Bakterien recherchiert, deren Genom bereits sequenziert ist. Die Genomdaten haben sie dann computergestützt mit den aufbereiteten Phänotypdaten verglichen und mithilfe maschineller Lernmethoden systematisch nach Mustern und Kombinationen von Proteinfamilien durchsuchen lassen, die eine genaue Vorhersage von Phänotypen ermöglichen.
„Für die Programmierung der Software haben wir Trainingsdaten verwendet, bei denen Genom und daraus resultierende Phänotypen bekannt waren“, erklärt Aaron Weimann. „Mit weiteren bekannten Datensätzen haben wir Traitar dann überprüft und anhand der richtigen und falschen Vorhersagen immer genauer programmieren können.“
Als Grundlage für das Training und die Überprüfung von Traitar dienten die Bakterienenzyklopädie Bergey's Manual of Systematic Bacteriology sowie die Datenbank GIDEON (Global Infectious Diseases and Epidemiology Online Network), die eine Vielzahl klinisch relevanter Eigenschaften von Bakterien zusammenfasst. Außerdem flossen Fördergelder des Deutschen Zentrums für Infektionsforschung (DZIF) in das Projekt.
„Die Vorhersage ist noch nicht für alle 67 Phänotypen gleich gut, weil die Qualität der zugrundeliegenden Daten in manchen Fällen besser ist als in anderen“, sagt Dr. Andreas Bremges, Wissenschaftler im Team von Alice McHardy. „Wenn neue Daten verfügbar werden, passen wir die Software im laufenden Prozess an. Und sie kann jederzeit um zusätzliche Phänotypen erweitert werden. Allerdings ist das Ziel von Traitar primär, Wissenschaftlern erste Hinweise zu geben, welche interessanten Eigenschaften die von ihnen untersuchten Bakterien haben können.“ So ermögliche die Software einen schnellen und gezielten Einstieg in die Charakterisierung und reduziere dadurch auch die Kosten für Laborexperimente.
In Forscherkreisen ist die Software bereits im Einsatz, so zum Beispiel in einem neu gestarteten Kooperationsprojekt mit Prof. Susanne Häußler vom HZI. In ihrer Abteilung „Molekulare Bakteriologie“ hat Häußler eine Vielzahl von Stämmen des Krankheitserregers Pseudomonas aeruginosa charakterisiert. Dieses Bakterium kann schwere akute Infektionen hervorrufen und zeichnet sich durch eine besonders hohe Resistenz gegenüber einer Vielzahl von Antibiotika aus. Die detaillierten Daten, die Häußler über diesen Erreger gesammelt hat, sollen nun in Traitar einfließen.
„Wir brauchen in Zukunft automatische Systeme, um genaue Vorhersagen für Antibiotikaresistenzen von Pseudomonas aeruginosa treffen und so eine passende Therapie für den Patienten wählen zu können“, sagt Häußler. „Gleichzeitig erhoffen wir uns, mit Traitar noch unbekannte Resistenzmechanismen aufzuklären.“
Originalpublikation:
Aaron Weimann, Kyra Mooren, Jeremy Frank, Phillip B. Pope, Andreas Bremges, Alice C. McHardy: From Genomes to Phenotypes: Traitar, the Microbial Trait Analyzer. mSystems, 2016, DOI: 10.1128/mSystems.00101-16
Die Pressemitteilung und Bildmaterial finden Sie auch auf unserer Webseite unter dem Link https://www.helmholtz-hzi.de/de/aktuelles/news/ansicht/article/complete/bakterie…
Das Helmholtz-Zentrum für Infektionsforschung:
Am Helmholtz-Zentrum für Infektionsforschung (HZI) untersuchen Wissenschaftler die Mechanismen von Infektionen und ihrer Abwehr. Was Bakterien oder Viren zu Krankheitserregern macht: Das zu verstehen soll den Schlüssel zur Entwicklung neuer Medikamente und Impfstoffe liefern. http://www.helmholtz-hzi.de
Das Braunschweig Integrated Centre of Systems Biology:
Das Braunschweig Integrated Centre of Systems Biology (BRICS) ist ein gemeinsames Forschungszentrum des HZI mit der Technischen Universität Braunschweig. Ziel des BRICS ist die Erforschung von Infektionen und Wirkstoffbildung sowie die Entwicklung biotechnologischer Prozesse mit Hilfe der Systembiologie. http://www.tu-braunschweig.de/brics
Ihre Ansprechpartner:
Susanne Thiele, Pressesprecherin
susanne.thiele@helmholtz-hzi.de
Dr. Andreas Fischer, Wissenschaftsredakteur
andreas.fischer@helmholtz-hzi.de
Helmholtz-Zentrum für Infektionsforschung GmbH
Presse und Kommunikation
Inhoffenstraße 7
D-38124 Braunschweig
Tel.: 0531 6181-1400; -1405
Media Contact
Alle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Diamantstaub leuchtet hell in Magnetresonanztomographie
Mögliche Alternative zum weit verbreiteten Kontrastmittel Gadolinium. Eine unerwartete Entdeckung machte eine Wissenschaftlerin des Max-Planck-Instituts für Intelligente Systeme in Stuttgart: Nanometerkleine Diamantpartikel, die eigentlich für einen ganz anderen Zweck bestimmt…
Neue Spule für 7-Tesla MRT | Kopf und Hals gleichzeitig darstellen
Die Magnetresonanztomographie (MRT) ermöglicht detaillierte Einblicke in den Körper. Vor allem die Ultrahochfeld-Bildgebung mit Magnetfeldstärken von 7 Tesla und höher macht feinste anatomische Strukturen und funktionelle Prozesse sichtbar. Doch alleine…

Hybrid-Energiespeichersystem für moderne Energienetze
Projekt HyFlow: Leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem für moderne Energienetze. In drei Jahren Forschungsarbeit hat das Konsortium des EU-Projekts HyFlow ein extrem leistungsfähiges, nachhaltiges und kostengünstiges Hybrid-Energiespeichersystem entwickelt, das einen…