Wie der Schnupfen in die Zelle kommt
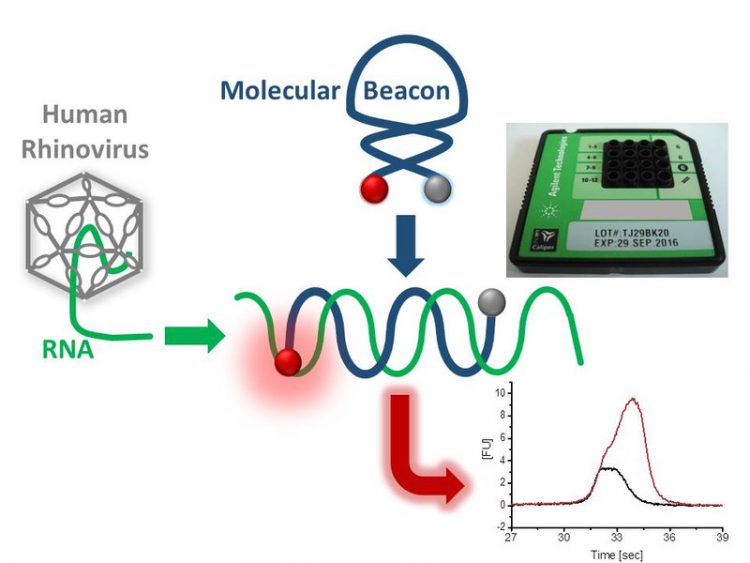
Das Messprinzip: Die RNA aus dem Virus wird mit molecular beacons markiert
Schnupfenviren verursachen uns Ärger, indem sie in unsere Zellen eindringen und dort die RNA aus ihrem Inneren in das Cytoplasma der infizierten Zelle transportieren. Erst dadurch können sie sich vermehren. Wie diese Ausschleusung der RNA aus dem Inneren des Virus im Detail abläuft, ist schwer zu untersuchen.
An der TU Wien wurden nun eine Methode entwickelt, mit der man diesen Prozess analysieren kann. Sie entstand aus der Kombination zweier etablierter Verfahren – sogenannten „Molecular Beacons (molekulare Leuchtfeuer)“ und der Kapillarelektrophorese im Chip-Format. Die neue Methode wurde nun publiziert und der Artikeltitel ziert das Cover des Fachjournals „Analytical and Bioanalytical Chemistry“.
Mini-Fußball mit Erbsubstanz
Das Schnupfenvirus, das Prof. Günter Allmaier und sein Team vom Institut für Chemische Technologien und Analytik studierten, ist relativ einfach aufgebaut. Es sieht aus wie ein Nano-Fußball mit einem Durchmesser von ungefähr 30 Nanometern. Seine Schale besteht aus vier verschiedenen Proteinen, die jeweils 60-fach vorhanden sind, im Inneren verbirgt sich die RNA, auf der die Erbinformation des Virus gespeichert ist.
„Bestimmte äußere Bedingungen können das Virus dazu bringen, seine RNA nach außen freizusetzen“, erklärt Victor Weiss, PostDoc von Günter Allmaier. „In unseren Zellen wird das durch einen niedrigeren pH-Wert ausgelöst, man kann denselben Effekt auch erzielen, indem man die Temperatur für zehn Minuten auf 57°C erhöht.“ In diesem Fall organisieren sich die Proteine um, die Schale des Virus bekommt Löcher, durch eines von ihnen wird dann der RNA-Strang freigegeben.
Für viele medizinische Fragen ist es wichtig, diesen Mechanismus genau zu verstehen – zum Beispiel für die künftige Entwicklung von Medikamenten, die genau diesen RNA-Transfer verhindern. Die Dynamik dieses Vorgangs konnte bisher nicht direkt beobachtet werden. In den Labors der TU Wien wird dieser Prozess aber nun experimentell zugänglich gemacht.
Fluoreszierende Marker und Elektrophorese
Man verwendet sogenante „Molecular Beacons“ – das sind maßgeschneiderte RNA (oder DNA-) Moleküle mit zwei verschiedenen Enden. An einem Ende sitzt ein Fluorophor, der aufleuchtet, wenn man ihn mit Laserlicht einer bestimmten Wellenlänge bestrahlt, am anderen Ende ein „Quencher“, der genau dieses Aufleuchten verhindert. „Anfangs ist das Molekül zusammengeklappt, Fluorophor und Quencher befinden sich ganz nahe nebeneinander, dann ist die Fluoreszenz sehr gering“, erklärt Victor Weiss.
Die Molecular Beacons können allerdings an eine ganz bestimmte RNA-Sequenz andocken. Wenn das passiert, klappt das Molekül auseinander, Fluorophor und Quencher sind plötzlich weit voneinander entfernt, und wenn man das Molekül dann mit dem passenden Laserlicht bestrahlt, fluoresziert es.
Man kann diese Molecular Beacons also verwenden, um bestimmte RNA-Sequenzen nachzuweisen. Diese Technik wurde an der TU Wien mit einer anderen bewährten Technik kombiniert – der Kapillarelektrophorese. Dabei trennt man die Komponenten einer Probe nach ihrer elektrophoretischer Mobilität (Wanderungsgeschwindigkeit in einem elektrischen Feld).
Eine kleine Flüssigkeitsprobe wird in einem Chip-Kanal platziert, und dort wir ein elektrisches Feld angelegt, in dem die unterschiedlichen Nanopartikel auf charakteristische Weise unterschiedlich schnell wandern. Nach einer Trennstrecke von etwa eineinhalb Zentimetern trifft dann ein Laserstrahl auf die Partikel. Dort werden dann die leuchtenden Fluorophore des ausgeklappten Molecular Beacons gemessen, die an der Viren-RNA andocken konnten.
„Die unterschiedlichen Bestandteile der Probe kommen zu unterschiedlichen Zeitpunkten beim Laser an, erst dadurch kann man sichergehen, dass man genau misst, was man eigentlich messen möchte“, erklärt Günter Allmaier. „Damit können wir nun beispielsweise zeigen, welches Ende der RNA zuerst aus dem Virus austritt, und wie dieser Prozess genau abläuft.“
Im Prinzip lässt sich die Methode, die im Rahmen eines FWF Projektes gemeinsam mit der Forschungsgruppe Dieter Blaas (Medizinische Universität Wien) entwickelt wurde, auch auf alle anderen Viren anwenden. „Uns geht es um die Entwicklung der Methode, als Testobjekt ist das Schnupfenvirus geradezu ideal“, meint Allmaier. „Wir hoffen aber natürlich, dass sich diese Methode in der medizinischen Forschung etabliert. Dass sie großes Potenzial hat, haben wir nun gezeigt und zeigt sich auch in der Kooperation mit der Firma Agilent Technologies.“
Originalpublikation: Analytical and Bioanalytical Chemistry, doi:10.1007/s00216-016-9459-2
Rückfragehinweis:
Prof. Günter Allmaier
Institut für Chemische Technologien und Analytik
Technische Universität Wien
Getreidemarkt 9/164, A-1060 Wien
T: +43-1-58801-15160
guenter.allmaier@tuwien.ac.at
Aussender:
Dr. Florian Aigner
Büro für Öffentlichkeitsarbeit
Technische Universität Wien
Operngasse 11, 1040 Wien
T.: +43-1-58801-41027
florian.aigner@tuwien.ac.at
http://link.springer.com/article/10.1007%2Fs00216-016-9459-2 Originalpublikation
Media Contact
Weitere Informationen:
http://www.tuwien.ac.atAlle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Ideen für die Zukunft
TU Berlin präsentiert sich vom 22. bis 26. April 2024 mit neun Projekten auf der Hannover Messe 2024. Die HANNOVER MESSE gilt als die Weltleitmesse der Industrie. Ihr diesjähriger Schwerpunkt…

Peptide auf interstellarem Eis
Dass einfache Peptide auf kosmischen Staubkörnern entstehen können, wurde vom Forschungsteam um Dr. Serge Krasnokutski vom Astrophysikalischen Labor des Max-Planck-Instituts für Astronomie an der Universität Jena bereits gezeigt. Bisher ging…

Wasserstoff-Produktion in der heimischen Garage
Forschungsteam der Frankfurt UAS entwickelt Prototyp für Privathaushalte: Förderzusage vom Land Hessen für 2. Projektphase. Wasserstoff als Energieträger der Zukunft ist nicht frei verfügbar, sondern muss aufwendig hergestellt werden. Das…