Was künstliche Intelligenz über Proteine verrät

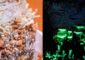
Das DeeProtein-Programm erlaubt es, Abschnitte in Proteinen zu erkennen, die wichtig (rot) oder weniger wichtig (blau) für die Funktion des Proteins sind. Dies können Forscher dazu verwenden, Proteine gezielt zu verändern. Im Bild: Das Protein ERK2, das bei Krebspatienten häufig mutiert ist. PDB 1GOL
Alexa, Siri und Google Assistant sind als intelligente, maschinelle Begleiter längst in den Alltag eingezogen. Und auch aus der Wissenschaft sind intelligente Rechenprogramme, so genannte Algorithmen, nicht mehr wegzudenken. Die großen Datenmengen, die in der biowissenschaftlichen Forschung auftreten, können mit Hilfe von Algorithmen effizient auf wiederkehrende Muster untersucht werden.
So können bestimmte Programme zum Beispiel erkennen, welche wiederkehrenden Strukturen in großen Eiweißmolekülen auftreten und daraus Rückschlüsse ziehen, welche Aufgaben diese in Zellen übernehmen, ob sie etwa als Genschalter, molekularer Motor oder als Signalmolekül aktiv sind. Die Vorhersagen, die solche Algorithmen auf Basis von Proteinsequenzen, also der perlenkettenartigen Abfolge von Proteinbausteinen, treffen, sind mittlerweile erstaunlich genau.
Ein entscheidender Nachteil bisheriger Verfahren ist allerdings, dass für Nutzer in keiner Weise nachvollziehbar ist, warum der Algorithmus bestimmten Proteinsequenzen eine bestimmte Funktion zuordnet. Das präzise Computerwissen über Proteine ist nicht direkt abrufbar, obwohl dieses Wissen für die Forschung als auch die Entwicklung von Wirkstoffen von großem Wert wäre.
Ein studentisches Team um Roland Eils und Irina Lehmann vom Berlin Institute of Health (BIH) und der Charité–Universitätsmedizin Berlin und ihrem Kollegen Dominik Niopek vom Institut für Pharmazie und Molekulare Biotechnologie (IPMB) an der Universität Heidelberg hat es sich zum Ziel gesetzt, dem Computer dieses Wissen zu entlocken.
Es arbeitet bereits seit 2017 an diesem Thema und entwickelte den Algorithmus „DeeProtein“, ein umfangreiches, intelligentes Neuronales Netzwerk, das die Funktion von Proteinen vorhersagen kann, basierend auf der Abfolge der einzelnen Proteinbausteine, der Aminosäuren.
Wie die meisten lernenden Algorithmen ist auch DeeProtein eine „Black Box“, wie es arbeitet, bleibt sowohl den Entwickler*innen als auch den Anwender*innen verborgen. Doch mit einem Trick gelang es nun den Student*innen, dem Netzwerk dieses Geheimnis zu entlocken.
Zunächst entwickelten die jungen Wissenschaftler*innen eine Möglichkeit, dem Programm während der Arbeit quasi über die Schulter zu schauen: „Bei der Sensitivitätsanalyse verdecken wir nacheinander jede einzelne Position in der Eiweißsequenz und lassen „DeeProtein“ aus dieser lückenhaften Information die Funktion des Eiweißes berechnen bzw. vorhersagen“, erläutert Julius Upmeier zu Belzen. Er ist Student im Masterstudiengang Molekulare Biotechnologie am IPMB und Erstautor der Publikation, die gerade in der Fachzeitschrift „Nature Machine Intelligence“ veröffentlicht wurde*.
„Im Anschluss geben wir „DeeProtein“ die vollständige Sequenzinformation und vergleichen die beiden Vorhersagen. So berechnen wir für jede einzelne Position in der Proteinsequenz, wie wichtig diese für die korrekte Vorhersage der Funktion ist. Das heißt, wir geben jeder Position bzw. Aminosäure innerhalb der Proteinkette einen Sensitivitätswert für die Proteinfunktion“.
Dieses neue Analyseverfahren nutzten die Wissenschaftler*innen anschließend dazu, diejenigen Bereiche in Proteinen zu identifizieren, die für ihre Funktion entscheidend sind. Das funktionierte für Signalproteine, die während der Krebsentstehung eine Rolle spielen, ebenso wie für die CRISPR-Cas9 Genschere, die bereits vielfach in präklinischen und klinischen Studien getestet wird.
„Mit der Sensitivitätsanalyse können wir Bereiche in Proteinen identifizieren, die Veränderungen besser oder weniger gut tolerieren. Das ist ein wichtiger erster Schritt, wenn wir Proteine gezielt verändern möchten, um ihnen neue Funktionen zu übertragen oder unerwünschte Eigenschaften auszuschalten“, sagt Dominik Niopek.
„Mit dieser Arbeit zeigen wir, dass nicht nur die Vorhersagen von neuronalen Netzwerken hilfreich sein können, sondern dass wir nun auch erstmals ihr implizites Wissen praktisch nutzen können“, erklärt Roland Eils. Dieser Ansatz ist für viele Fragestellungen in Molekularbiologie und Medizin relevant.
„Wenn wir zum Beispiel zielgerichtete Medikamente oder Gentherapien entwickeln möchten, müssen wir genau wissen, an welcher Stelle wir ansetzen können. Dabei kann uns nun „DeeProtein“ unterstützen.“
*Upmeier zu Belzen et al. (2019): Leveraging implicit knowledge in neural networks for functional dissection and engineering of proteins. Nature Machine Intelligence.
DOI: 10.1038/s42256-019-0049-9
Abbildungen zur Pressemitteilung finden Sie hier:
https://www.bihealth.org/fileadmin/artikel/pressemitteilungen/bilder/DeeProtein-…
BU: Das DeeProtein-Programm erlaubt es, Abschnitte in Proteinen zu erkennen, die wichtig (rot) oder weniger wichtig (blau) für die Funktion des Proteins sind. Dies können Forscher dazu verwenden, Proteine gezielt zu verändern. Im Bild: Das Protein ERK2, das bei Krebspatienten häufig mutiert ist. PDB 1GOL
https://www.bihealth.org/fileadmin/artikel/pressemitteilungen/bilder/DeeProtein-…
BU: Das DeeProtein-Programm erlaubt es, Abschnitte in Proteinen zu erkennen, die wichtig (rot) oder weniger wichtig (blau) für die Funktion des Proteins sind. Dies können Forscher dazu verwenden, Proteine gezielt zu verändern. Im Bild: Das Protein AcrIIA4, das die Genschere CRISPR-Cas9 stoppen kann. PDB 5VGB
Über das Berliner Institut für Gesundheitsforschung/Berlin Institute of Health (BIH)
Das Berliner Institut für Gesundheitsforschung | Berlin Institute of Health (BIH) ist eine Wissenschaftseinrichtung für Translation und Präzisionsmedizin. Das BIH widmet sich neuen Ansätzen für bessere Vorhersagen und neuartigen Therapien bei progredienten Krankheiten, um Menschen Lebensqualität zurückzugeben oder sie zu erhalten.
Mit translationaler Forschung und Innovationen ebnet das BIH den Weg für eine nutzenorientierte personalisierte Gesundheitsversorgung. Das BIH wird zu 90 Prozent vom Bundesministerium für Bildung und Forschung (BMBF) und zu zehn Prozent vom Land Berlin gefördert. Die Gründungsinstitutionen Charité – Universitätsmedizin Berlin und Max-Delbrück-Centrum für Molekulare Medizin in der Helmholtz-Gemeinschaft (MDC) sind im BIH eigenständige Gliedkörperschaften.
Kontakt
Dr. Stefanie Seltmann
Leiterin Kommunikation & Marketing
+49 (0) 30 450 543019
s.seltmann@bihealth.de
https://www.bihealth.org/de/aktuell/?L=0&tx_news_pi1%5Bnews%5D=2105&tx_n…
Media Contact
Weitere Informationen:
http://www.bihealth.orgAlle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Bakterien für klimaneutrale Chemikalien der Zukunft
Forschende an der ETH Zürich haben Bakterien im Labor so herangezüchtet, dass sie Methanol effizient verwerten können. Jetzt lässt sich der Stoffwechsel dieser Bakterien anzapfen, um wertvolle Produkte herzustellen, die…

Batterien: Heute die Materialien von morgen modellieren
Welche Faktoren bestimmen, wie schnell sich eine Batterie laden lässt? Dieser und weiteren Fragen gehen Forschende am Karlsruher Institut für Technologie (KIT) mit computergestützten Simulationen nach. Mikrostrukturmodelle tragen dazu bei,…

Porosität von Sedimentgestein mit Neutronen untersucht
Forschung am FRM II zu geologischen Lagerstätten. Dauerhafte unterirdische Lagerung von CO2 Poren so klein wie Bakterien Porenmessung mit Neutronen auf den Nanometer genau Ob Sedimentgesteine fossile Kohlenwasserstoffe speichern können…