Was einen kleinen Wurm zum gefragten Modell macht
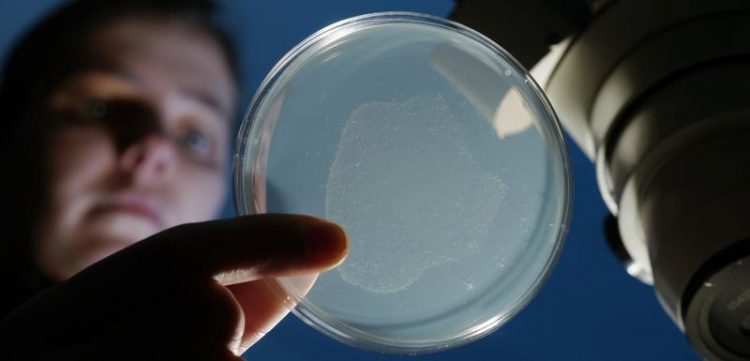
Mit dem bloßen Auge in der Petrischale kaum zu sehen, aber für die Forschung enorm wichtig: Der Fadenwurm Caenorhabditis elegans. Foto: Jan-Peter Kasper
Er ist nur etwa einen Millimeter lang, mit bloßem Auge also kaum zu erkennen. Sein kurzes Dasein von gerade einmal zwei bis drei Wochen fristet er verborgen im Boden. Auf den ersten Blick haben der winzige Fadenwurm Caenorhabditis elegans und der Mensch nicht viel gemeinsam. Und doch weist ihr Erbgut erstaunliche Ähnlichkeiten auf:
„Bis zu 80 Prozent der Gene von Fadenwurm und Mensch sind gleich“, sagt der Kieler Bioinformatiker Dr. Christoph Kaleta, Professor für Medizinische Systembiologie an der Medizinischen Fakultät der CAU und Mitglied im schleswig-holsteinischen Exzellenzcluster Entzündungsforschung. „Rund die Hälfte aller bekannten Gene, die an der Entstehung menschlicher Krankheiten beteiligt sind, liegen auch in C. elegans vor.“ Daher dient der Wurm als einfaches Modellsystem, um die Lebensprozesse sowie die Entstehung von Krankheiten – auch des Menschen – zu untersuchen und besser zu verstehen.
Dies ist für die Wissenschaftler weltweit nun auf einer neuen Basis möglich: Kaleta und sein Team haben gemeinsam mit Kollegen der Universität Jena und der ETH Zürich den kompletten Metabolismus des Wurms in einem mathematischen Modell zusammengefasst. Damit lassen sich sämtliche seiner Stoffwechselwege „in silico“ – also am Computer – nachvollziehen und untersuchen.
„Insbesondere können wir auch Vorhersagen treffen, wie sich bestimmte Änderungen – etwa in der Nahrungsaufnahme – auf das Wachstum, die Lebenserwartung oder die Entstehung von Krankheiten in diesem Organismus auswirken“, erläutert Kaleta, der bis 2014 als Juniorprofessor an der Uni Jena forschte und lehrte. Gemeinsam mit seiner früheren Arbeitsgruppe und Alternsforschern aus Jena und Zürich stellt er das Modell mit dem Namen „ElegCyc“ in der aktuellen Ausgabe des Wissenschaftsmagazins „Cell Systems“ vor (DOI: 10.1016/j.cels.2016.04.017).
In mühevoller Kleinarbeit haben die Forscher Datenbanken und Fachliteratur durchforstet und sämtliche bislang beschriebene Stoffwechselwege von C. elegans zusammengetragen. Insgesamt sind auf diese Weise knapp 2.000 Stoffwechselprozesse erfasst und miteinander verknüpft worden. Herausgekommen ist ein komplexes Netzwerk, das die Lebensprozesse des Wurms detailliert aufzeigt und das die Wissenschaftler nun nach einzelnen Genen, Stoffwechselprodukten oder Reaktionen durchsuchen können.
Der große Vorteil solch umfassender Computermodelle ist, dass sich damit auch sehr große Datenmengen analysieren lassen, sagt Juliane Gebauer vom Lehrstuhl für Bioinformatik der Uni Jena. „Moderne Hochdurchsatzverfahren, wie die RNA-Sequenzierung oder die Bestimmung der Konzentration von Stoffwechselprodukten liefern eine solche Fülle an Messdaten, die ohne entsprechende Modelle kaum sinnvoll zu nutzen wären“, so die Doktorandin, die Erstautorin der Veröffentlichung ist.
Dass ihr Modell diese Ansprüche erfüllt, konnten die Bioinformatiker in der nun vorgelegten Studie zeigen: Sie fütterten das Modell mit experimentellen Daten zum Alterungsprozess aus dem Jenaer Forschungskonsortiums JenAge. Vorausgegangen war ein Fütterungsexperiment mit C. elegans. Die Würmer erhielten drei unterschiedliche Substanzen zu fressen, von denen zwei bekanntermaßen die Lebensspanne verlängern.
Zu insgesamt vier Zeitpunkten wurde die Aktivität der rund 20.000 Gene von C. elegans bestimmt. Dank des Modells „ElegCyc“ spürten die Forscher aus dieser großen Zahl von Genen diejenigen Stoffwechselprozesse auf, die maßgeblich an der lebensverlängernden Wirkung der gefütterten Substanzen beteiligt sind.
In ihrer zukünftigen Arbeit wollen die Forscher ihr Modell verwenden, um die Wechselwirkung zwischen dem Wurm und seiner bakteriellen Flora im Darm besser zu verstehen. Da diese im Gegensatz zu der hochkomplexen Darmflora des Menschen bei dem Wurm sehr einfach ist, eignet sie sich besonders gut, um zu untersuchen, wie bestimmte Bakterienarten der Darmflora die Gesundheit als auch Krankheiten begünstigen. Aus diesen Arbeiten erhoffen sich die Forscher neue Einsichten in die Entstehung von chronisch-entzündlichen Darmerkrankungen, die mit einem starken Ungleichgewicht in der Darmflora einhergehen.
Das Metabolismus-Modell von C. elegans ist im Internet frei zugänglich unter: http://elegcyc.bioinf.uni-jena.de:1100
Original-Publikation:
Gebauer et al., A Genome-Scale Database and Reconstruction of Caenorhabditis elegans Metabolism, Cell Systems (2016), http://dx.doi.org/10.1016/j.cels.2016.04.017
Kontakt:
Juliane Gebauer
Lehrstuhl für Bioinformatik der Friedrich-Schiller-Universität Jena
Ernst-Abbe-Platz 2, 07743 Jena
Tel.: 03641 / 949594
E-Mail: juliane.gebauer[at]uni-jena.de
Prof. Dr. Christoph Kaleta
Exzellenzcluster „Entzündungsforschung“ und Institut für Experimentelle Medizin der Christian-Albrechts-Universität zu Kiel
Michaelisstr. 5, Haus 17, 24105 Kiel
Tel.: 0431 / 5973046
E-Mail: c.kaleta[at]iem.uni-kiel.de
Media Contact
Alle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Bakterien für klimaneutrale Chemikalien der Zukunft
Forschende an der ETH Zürich haben Bakterien im Labor so herangezüchtet, dass sie Methanol effizient verwerten können. Jetzt lässt sich der Stoffwechsel dieser Bakterien anzapfen, um wertvolle Produkte herzustellen, die…

Batterien: Heute die Materialien von morgen modellieren
Welche Faktoren bestimmen, wie schnell sich eine Batterie laden lässt? Dieser und weiteren Fragen gehen Forschende am Karlsruher Institut für Technologie (KIT) mit computergestützten Simulationen nach. Mikrostrukturmodelle tragen dazu bei,…

Porosität von Sedimentgestein mit Neutronen untersucht
Forschung am FRM II zu geologischen Lagerstätten. Dauerhafte unterirdische Lagerung von CO2 Poren so klein wie Bakterien Porenmessung mit Neutronen auf den Nanometer genau Ob Sedimentgesteine fossile Kohlenwasserstoffe speichern können…