Der Neandertaler in uns

Erstmals liegt eine Version der Genomsequenz einer ausgestorbenen Menschenart vor. Forscher des Max-Planck-Institutes für evolutionäre Anthropologie in Leipzig präsentieren gemeinsam mit einem internationalen Forschungsteam einen ersten Entwurf der Gensequenz des vor rund 30.000 Jahren ausgestorbenen Neandertalers. Erste Analysen von vier Milliarden Basenpaaren weisen darauf hin, dass Neandertaler im Genom einiger moderner Menschen Spuren hinterlassen haben. (Science, 7. Mai 2010)
Nach vier Jahren ist es geschafft: Ein Forscherteam unter der Leitung von Svante Pääbo, Direktor der Abteilung für Evolutionäre Genetik des Max-Planck-Institutes für evolutionäre Anthropologie in Leipzig veröffentlicht in der aktuellen Ausgabe des Fachmagazins Science eine erste Version der Genomsequenz des Neandertalers.
Es ist eine bisher einmalige wissenschaftliche Leistung: Fast zehn Jahre nach Entschlüsselung des Genoms des Homo sapiens präsentieren Forscher nun erstmals die Gensequenz eines ausgestorbenen Hominiden, der zudem der engste ausgestorbene Verwandte des Menschen ist. „Der Vergleich dieser beiden Gensequenzen gibt uns die Möglichkeit zu erfahren, wo wir uns in unserem Genom von unseren nächsten Verwandten unterscheiden“, sagt Svante Pääbo.
Die Version der Neandertaler-Sequenz basiert auf der Analyse von mehr als einer Milliarde DNA-Fragmente aus mehreren Neandertaler-Knochen aus Kroatien, Spanien, Russland und dem Neandertal in Deutschland. Unter den DNA-Fragmenten haben die Leipziger Forscher diejenigen identifiziert, die aus dem Neandertaler-Genom stammen und zusammen mehr als sechzig Prozent des Gesamtgenoms abdecken.
Ein erster Vergleich der beiden Sequenzen förderte bereits erste aufregende Entdeckungen zutage. Anders als von vielen Forschern vermutet, haben sich Neandertaler und der frühe moderne Mensch offensichtlich vermischt. Im Genom einiger heute lebender Menschen stammen nach Berechnungen der Forscher ein bis vier Prozent der DNA vom Neandertaler. „Diejenigen von uns, die außerhalb Afrikas leben, tragen ein kleines bisschen Neandertaler in sich“, sagt Svante Pääbo. Bei vorangegangen Untersuchungen der DNA von Mitochondrien der Neandertaler hatte man für eine Vermischung keine Hinweise gefunden.
Für die Analyse sequenzierten die Forscher zusätzlich fünf menschliche Genome europäischer, asiatischer und afrikanischer Abstammung und verglichen diese mit dem Neandertaler-Genom. Die Überraschung: Der Neandertaler hat ein wenig mehr genetische Gemeinsamkeiten mit den Menschen außerhalb Afrikas als mit den Afrikanern. Zugleich ähnelt das Neandertaler-Genom der Sequenz von Europäern im gleichen Ausmaß wie der von Ostasiaten. Das verwundert, denn bis heute wurden keine Überreste von Neandertalern in Ostasien gefunden. Sie lebten in Europa und Westasien.
Die Forscher haben aber eine einleuchtende Erklärung für ihre Ergebnisse. Svante Pääbo: „Neandertaler haben sich wahrscheinlich mit frühen modernen Menschen vermischt bevor Homo sapiens sich in Europa und Asien in verschiedene Gruppen aufspaltete.“ Dies war in einem Zeitraum zwischen 100.000 bis 50.000 Jahren im Mittleren Osten möglich, noch bevor die menschliche Population sich über Eurasien ausbreitete. Aus archäologischen Funden weiß man, dass damals Neandertaler und Menschen dieselbe Region bewohnten.
Abgesehen von der Frage, ob Neandertaler und Homo sapiens sich vermischt haben, gilt das Hauptinteresse der Forscher Genbereichen, die den Menschen von seinem nächsten Verwandten unterscheiden und ihm vielleicht Vorteile im Laufe der Evolution einbrachten.
Die Wissenschaftler um Pääbo haben bereits einige Regionen entdeckt, in denen sie Gene ausfindig machten, die möglicherweise eine wichtige Rolle in der menschlichen Evolution spielten. So fanden sie Gene, die mit kognitiven Funktionen, mit dem Stoffwechsel und mit der Entwicklung von Schädel, Schlüsselbein und Brustkorb zusammenhängen. Doch erst genauere Analysen werden Rückschlüsse über den tatsächlichen Einfluss dieser Gene zulassen.
Den Großteil der DNA für ihre Untersuchung gewann das Forschungsteam aus insgesamt 400 Milligramm Knochenpulver aus Knochen dreier weiblicher Neandertaler, die in einer Höhle in Kroatien ausgegraben wurden und dort vor mehr als 38.000 Jahren lebten.
Das Genom einer vor Zehntausenden von Jahren ausgestorbenen Art zu sequenzieren ist eine ganz besondere Herausforderung, denn die DNA ist im Laufe der Zeit zu winzigen Fragmenten zerfallen und zum Teil chemisch verändert. Hinzu kommt das Problem der Verunreinigung. „Mehr als 95 Prozent der DNA in einer Probe stammen von Bakterien und Mikroorganismen, die den Neandertaler nach seinem Tod besiedelten“, sagt Svante Pääbo. Auch menschliche DNA, die bei der Ausgrabung oder im Labor in die Probe gelangt, verfälscht die Ergebnisse. Pääbo und sein Team in Leipzig setzen verschiedene, zum Teil völlig neu entwickelte Techniken ein, um die zu sequenzierende DNA von Kontaminationen zu befreien. Sie bearbeiten die Proben in Reinsträumen und markieren jedes Sequenzstück eines Neandertalers mit einem kurzen Stück DNA als Etikett, um es von menschlicher DNA unterscheiden zu können.
Die technischen Herausforderungen haben die Forscher inzwischen im Griff. Jetzt schauen sie optimistisch in die Zukunft: „Wir werden auch die verbleibende Sequenz des Neandertalers entschlüsseln und noch viel mehr über uns und unseren nächsten Verwandten erfahren“, sagt Svante Pääbo.
Originalveröffentlichung:
Richard E. Green, Johannes Krause, Adrian W. Briggs, Tomislav Maricic, Udo Stenzel, Martin Kircher, Nick Patterson, Heng Li, Weiwei Zhai, Markus Hsi-Yang Fritz, Nancy F. Hansen, Eric Y. Durand, Anna-Sapfo Malaspinas, Jeffrey D. Jensen, Tomas Marques-Bonet, Can Alkan, Kay Prüfer, Matthias Meyer, Hernán A. Burbano, Jeffrey M. Good, Rigo Schultz, Ayinuer Aximu-Petri, Anne Butthof, Barbara Höber, Barbara Höffner, Madlen Siegemund, Antje Weihmann, Chad Nusbaum, Eric S. Lander, Carsten Russ, Nathaniel Novod, Jason Affourtit, Michael Egholm, Christine Verna, Pavao Rudan, Dejana Brajkovic, Željko Kucan, Ivan Gušic, Vladimir B. Doronichev, Liubov V. Golovanova, Carles Lalueza-Fox, Marco de la Rasilla, Javier Fortea, Antonio Rosas, Ralf Schmitz, Philip L.F. Johnson, Evan E. Eichler, Daniel Falush, Ewan Birney, James C. Mullikin, Montgomery Slatkin, Rasmus Nielsen, Janet Kelso, Michael Lachmann, David Reich, Svante Pääbo
A draft sequence and preliminary analysis of the Neandertal genome
Science, 7. Mai 2010
Weitere Informationen erhalten Sie von:
Prof. Dr. Svante Pääbo
Max-Planck-Institut für evolutionäre Anthropologie, Leipzig
Tel.: +49 341 3550 – 500
Fax: +49 341 3550 – 555
E-Mail: paabo@eva.mpg.de
Sandra Jacob, Presse- und Öffentlichkeitsarbeit
Max-Planck-Institut für evolutionäre Anthropologie, Leipzig
Tel.: +49 341 3550 – 122
Fax: +49 341 3550 – 119
E-Mail: info@eva.mpg.de
Media Contact
Alle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Entscheidender Durchbruch für die Batterieproduktion
Energie speichern und nutzen mit innovativen Schwefelkathoden. HU-Forschungsteam entwickelt Grundlagen für nachhaltige Batterietechnologie. Elektromobilität und portable elektronische Geräte wie Laptop und Handy sind ohne die Verwendung von Lithium-Ionen-Batterien undenkbar. Das…

Wenn Immunzellen den Körper bewegungsunfähig machen
Weltweit erste Therapie der systemischen Sklerose mit einer onkologischen Immuntherapie am LMU Klinikum München. Es ist ein durchaus spektakulärer Fall: Nach einem mehrwöchigen Behandlungszyklus mit einem immuntherapeutischen Krebsmedikament hat ein…
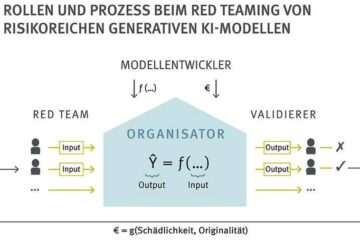
So soll risikoreiche generative KI geprüft werden
Die beschlossene KI-Verordnung der EU sieht vor, dass Künstliche-Intelligenz-Modelle (KI) „für allgemeine Zwecke mit systemischem Risiko“ besonders strikt überprüft werden. In diese Modellkategorie gehören auch populäre generative KI-Modelle wie GPT4…