Einzelne Moleküle auf Nanoebene abbilden? Neue Software simuliert superhochaufgelöste Lichtmikroskopie und ermöglicht die Planung von Laborversuchen
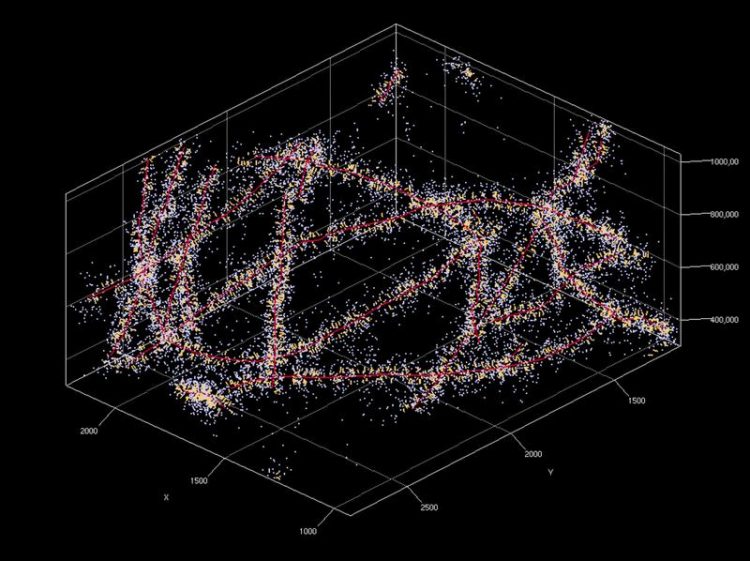
Mikrotubuli des Zytoskeletts einer Nervenzellle, röhrenähnliche Strukturen welchen den Zellen mechanische Stabilität geben, in räumlicher Anordnung wurden hier für eine Aufnahme mit der Einzelmolekül-Fluoreszenzmikroskopie simuliert: Die roten Linien stellen die Kurve der Mikrotubuli in räumlicher Anordnung dar, die gelben Stifte die Orientierung von Fluoreszenzmarkern und die weißen Punkte zeigen simulierte Lokalisationen. Die Lokalisationen skizzieren die Ansiedlung der Mikrotubuli mit höchster Präzision (Zahlen sind Entfernungen in Nanometern). Foto: Varun Venkatamarani und Frank Herrmannsdörfer, Abteilung Funktionelle Neuroanatomie, Institut für Anatomie und Zellbiologie.
Wissenschaftler am Institut für Anatomie und Zellbiologie der Mediznischen Fakultät Heidelberg haben die Opensource Software SuReSim (Super Resolution Simulation) entwickelt: Dieses Programm ermöglicht Anwendern Voraussagungen darüber zu treffen, ob eine biologische Struktur mit der Einzelmolekül-Fluoreszenzmikroskopie, einer Technik der superhochaufgelösten Lichtmikroskopie für welche 2014 der Nobelpreis verliehen wurde, dargestellt werden kann.
Die Forschungsgruppe hat damit in zweierlei Hinsicht einen neuen, optimierten Ansatz geschaffen: Zum einen ermöglicht die Software, Bildgebungsversuche mit Einzelmolekül-Fluoreszenzmikroskopie zu planen und zu validieren; zum anderen kann sie unnötige Versuchsreihen vermeiden und hilft so, wertvolle Zeit und Ressourcen zu sparen und eine Unter- oder Überinterpretation von Bilddaten zu vermeiden. Die Ergebnisse der Arbeit wurden vorab online in „Nature Methods“ veröffentlicht.
Einzelmolekül-Fluoreszenzmikroskopie, eine spezifische Form der superhochauflösenden Lichtmikroskopie, wird zunehmend genutzt, um molekulare und zelluläre Strukturen auf Nanoebene zu entschlüsseln. In der Regel gehen Wissenschaftler davon aus, dass die meisten molekularen Strukturen auf dieser Skala aufgelöst werden können.
Dennoch bleibt das Planen und Validieren von Versuchen, welche es zum Ziel haben molekulare Strukturen und deren Anordnung aufzulösen, eine grundlegende Herausforderung. SuReSim ist ein Softwareprogramm, das von den Mitgliedern des Exzellenzclusters CellNetworks der Universität Heidelberg, Prof. Dr. Thomas Kuner (Institut für Anatomie und Zellbiologie) und Prof. Dr. Mike Heilemann (Institut für Physikalische und Theoretische Chemie, Goethe Universität Frankfurt), entwickelt wurde.
Auf der Grundlage von Modellen aus elektronenmikroskopischen Daten simuliert das Programm Daten zur Anordnung beliebiger dreidimensionaler Strukturen. Anwender können so systematisch erforschen, wie sich Änderungen in den Versuchsparametern auf die potenziellen Bildgebungen auswirken, also ob die molekularen Strukturen dann mikroskopisch darstellbar sind.
Kontaktstellen zwischen Nervenzellen erfolgreich im Detail dargestellt und vorab simuliert
„Wir haben mit der Einzelmolekül-Fluoreszenzmikroskopie die Proteinzusammensetzung an Kontaktstellen zwischen Nervenzellen, der so genannten aktiven Zone von Synapsen, untersucht“, erläutert Prof. Dr. Kuner. „Daraus ist das Forschungsprojekt zur neuen Software entstanden. Mit Hilfe der Simulation haben wir verstanden, dass nicht nur die primäre Auflösung des Mikroskops eine Rolle spielt, sondern eben auch die Dichte und Anordnungen der Proteine im Präparat entscheidend dafür ist, ob Strukturen geeignet dargestellt werden können oder nicht.“
SuReSim nutzt einen Ansatz zur Bildsimulation, der auf detaillierten Strukturmodellen basiert. Solche Modelle können für viele Versuche entwickelt werden, weil die zelluläre Ultrastruktur und die Strukturen vieler Proteine gut erforscht sind, auch wenn Details der molekularen 3D-Nanostruktur von Zellen meist noch unklar bleiben.
Der SuReSim-Workflow startet mit einem Strukturmodell, welches je nach Vorwissen spezifiziert werden kann. In einem zweiten Schritt lassen sich fluoreszierende Farbstoffe in einer bestimmten Dichte und Orientierung hinzufügen. Die Software simuliert die Anordnung der Marker und gibt schließlich ein Bild aus. So kann eine Vielzahl wichtiger Versuchsparameter für die Einzelmolekül-Fluoreszenzmikroskopie angewandt und ihr Einfluss auf das daraus entstandene Bild ausgewertet werden.
Die volle Funktion von SuReSim liegt in der Hypothesenprüfung und der Versuchsplanung. Um die Funktionalität des Programms zu zeigen, nutzten Varun Venkataramani und Frank Herrmannsdörfer, Doktoranden aus der Forschungsgruppe von Prof. Dr. Kuner, bestimmte Teile von Nervenzellen, die synaptischen Bläschen. Sie besitzen eine eindeutige Größe und molekulare Struktur und sind dafür bekannt, dass sie sich dicht gedrängt in Gruppen an den Kontaktstellen von Nervenzellen – den Synapsen- sammeln.
Dennoch bleiben ihre Interaktionen untereinander sowie mit ihrer Umgebung, z.B. Elementen des so genannten Zytoskeletts oder der aktiven Zone der Synapsen, wenig erforscht. Das gleiche gilt für die molekulare Struktur. SuReSim wurde eingesetzt, um zu testen, ob die 3D-Einzelmolekül-Fluoreszenzmikroskopie einzelne Bläschen und die Moleküle, die die synaptischen Bläschen in Gruppen organisieren, visualisieren kann. Das Versuchsergebnis sagte voraus, dass einzelne synaptische Bläschen in einem Bläschencluster tatsächlich dargestellt werden können, wenn die fluoreszierenden Farbstoffe als Marker an spezifischen Stellen gesetzt und ideale Bedingungen für die Aufnahme der Bilder gegeben sind.
„SuReSim ist ein vielseitiges Programm mit den unterschiedlichsten Anwendungen“, fügt Prof. Dr. Kuner hinzu. „Einerseits kann es Vorhersagen über die Visualisierung der Zellstruktur machen, wie sie mit der Einzelmolekül-Fluoreszenzmikroskopie dargestellt werden. Aber es können auch Auswertungen von strukturellen Hypothesen gemacht werden. Daher kann die Software zu einem nützlichen Programm für die effiziente Planung von Versuchen mit Einzelmolekül-Fluoreszenzmikroskopie werden, womit wertvolle Ressourcen eingespart werden können.“
Literatur:
Varun Venkataramani, Frank Herrmannsdörfer, Mike Heilemann & Thomas Kuner: SuReSim: simulating localization microscopy experiments from ground truth models. Nature Methods (2016), doi:10.1038/nmeth.3775
Kontakt:
Prof. Dr. Thomas Kuner
Abteilung Funktionelle Neuroanatomie
Institut für Anatomie und Zellbiologie
Universität Heidelberg
06221-54-8678
kuner@uni-heidelberg.de
http://www.ana.uni-heidelberg.de/index.php?id=164 Abteilung Funktionelle Neuroanatomie
http://www.ana.uni-heidelberg.de/?id=198 SuReSim Software Projekt
http://www.cellnetworks.uni-hd.de/ Exzellenzcluster CellNetworks
Media Contact
Weitere Informationen:
http://www.klinikum.uni-heidelberg.de/Alle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Nanofasern befreien Wasser von gefährlichen Farbstoffen
Farbstoffe, wie sie zum Beispiel in der Textilindustrie verwendet werden, sind ein großes Umweltproblem. An der TU Wien entwickelte man nun effiziente Filter dafür – mit Hilfe von Zellulose-Abfällen. Abfall…

Entscheidender Durchbruch für die Batterieproduktion
Energie speichern und nutzen mit innovativen Schwefelkathoden. HU-Forschungsteam entwickelt Grundlagen für nachhaltige Batterietechnologie. Elektromobilität und portable elektronische Geräte wie Laptop und Handy sind ohne die Verwendung von Lithium-Ionen-Batterien undenkbar. Das…

Wenn Immunzellen den Körper bewegungsunfähig machen
Weltweit erste Therapie der systemischen Sklerose mit einer onkologischen Immuntherapie am LMU Klinikum München. Es ist ein durchaus spektakulärer Fall: Nach einem mehrwöchigen Behandlungszyklus mit einem immuntherapeutischen Krebsmedikament hat ein…





















