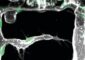
Computational Biology ermöglicht atomistische Einblicke in Proteinstrukturen

Struktur der dimeren Transmembran-Segmente des Rezeptors für das Blutplättchen-Wachstumshormon (grün), eingebettet in eine Lipidmembran (blau).<br>Foto: Universität Wien, KIT<br>
Aus den daraus gewonnenen Erkenntnissen können neue Strategien zur gezielten Ansteuerung von Rezeptoren durch pharmazeutische Wirkstoffe entwickelt werden. Die WissenschafterInnen publizieren dazu aktuell im renommierten Fachmagazin „The Journal of Biological Chemistry“.
Jede Zelle steht in ständigem Informationsaustausch mit ihrer Umgebung. Üblicherweise tragen Hormone als molekulare Botenstoffe die entsprechenden Signale von außen an die Zelle heran, z.B. um sie zum Wachsen anzuregen. Membranständige Rezeptoren – d.h. Proteinmoleküle, die die Zellmembran durchspannen – leiten die Information in das Innere der Zelle weiter. Dort wird die Antwort in einer weiteren Reaktionskaskade umgesetzt. Typische Zelloberflächenrezeptoren sind die sogenannten Tyrosinkinasen, die eine entscheidende Rolle in der Zellentwicklung, Zellteilung, Wundheilung und zellulären Homöostase – dem Gleichgewichtszustand in einer Zelle – spielen.
Eine fehlerhafte Weiterleitung der Signale kann u.a. zur Krebsentstehung führen. Aus pharmakologischer Sicht bieten diese Rezeptoren einen Hauptangriffspunkt für maßgeschneiderte pharmazeutische Wirkstoffe.
Computergestützte Strukturberechnungen zur Analyse zellulärer Signal-Prozesse
Über die molekulare Funktionsweise der Tyrosinkinase-Rezeptoren ist bekannt, dass sie sich bei ihrer Aktivierung paarweise zusammen lagern, also dimerisieren. Da dies in der Membran erfolgt, ist es nahe liegend, dass auch die umgebenden Lipidmoleküle dabei eine Rolle spielen. Zellmembranen bestehen aus vielen verschiedenen Lipiden, die eine Doppelschicht ausbilden. Aufgrund ihrer heterogenen Zusammensetzung enthält diese nicht nur dünne weiche Regionen, in denen die eingelagerten Proteine frei herum schwimmen können, sondern auch dicke zähflüssige Bereiche, die als Lipid-Flöße bezeichnet werden. In letzteren Mikrodomänen finden viele Signal-Prozesse statt, da sich bestimmte Rezeptoren dort offensichtlich bevorzugt aufhalten. Es ist jedoch nicht bekannt, welchen Einfluss die Membrandicke auf die Fähigkeit zur Dimerisierung hat und ob dadurch die Signalübertragung gesteuert werden kann.
Um die Stabilität der Rezeptoren in verschiedenen Lipiden zu untersuchen und im monomeren und dimeren Zustand miteinander zu vergleichen, müssen ihre dreidimensionalen Strukturen aufgeklärt werden. Die Membran-durchspannenden Protein-Segmente bestehen jeweils nur aus einer Alpha-Helix, die sich jedoch – aufgrund der relevanten Lipidumgebung – nicht mit konventionellen kristallographischen Verfahren untersuchen lassen. Stattdessen müssen moderne spektroskopische Methoden und computergestützte Strukturberechnungen eingesetzt werden.
Claudia Muhle-Goll und Silke Hoffmann vom Karlsruher Institut für Technologie (KIT) haben in Kooperation mit der Universität Wien aktuell eine Untersuchung über die Rolle der Membrandicke bei der Rezeptor-Dimerisierung durchgeführt. Die dimere Struktur des Transmembran-Segments vom -Rezeptor des Blutplättchen-Wachstumshormons wurde unter der Leitung von Anne S. Ulrich, Professorin am Institut für Biologische Grenzflächen des KIT, aufgeklärt. Dazu wurden hochauflösende Kernspinresonanz (NMR) und orientierte Festkörper-NMR Messungen in unterschiedlichen Membranumgebungen eingesetzt sowie eine spezielle Form des orientierten Zirkulardichroismus.
Computational Biology ermöglicht Molekulardynamik-Simulationen
Anton Polyansky, Postdoc der Universität Wien, führte im Rahmen dieser Forschungsarbeit Molekulardynamik-Simulationen durch. Mit Hilfe der Supercomputer des Departments für Strukturbiologie und Computational Biology der Universität Wien konnte ein atomistischer Einblick in die Lipid-eingebettete Proteinstruktur erzielt werden. So zeigte sich in dicken Floß-ähnlichen Lipiden ein stabiles linksgängig gewundenes Dimer, welches sich jedoch zu neigen beginnt und auseinanderbricht, wenn die Doppelschicht die typische Dünne einer fluiden Zellmembran erreicht. Dieses Verhalten zeigt, dass die Wechselwirkung der Transmembran-Helix mit der Lipidumgebung einen entscheidenden Einfluss auf die Dimerisierung und Rezeptor-Aktivierung ausübt. Mit Hilfe dieser strukturellen Einblicke können nun neue Strategien zur gezielten Ansteuerung von Rezeptoren durch pharmazeutische Wirkstoffe entwickelt werden.
Publikation:
The Journal of Biological Chemistry: Hydrophobic Matching Controls the Tilt and Stability of the Dimeric Platelet-derived Growth Factor Receptor (PDGFR) β Transmembrane Segment. Silke Hoffmann; Claudia Muhle-Goll; Anton Polyansky; Sergii Afonin; Marcel Zeitler; Stephan L. Grage; Dirk Windisch; Jochen Bürck; Anne S. Ulrich. Vol. 287, Issue 31, 26178-26186, July 27, 2012.
http://www.jbc.org/content/early/2012/05/22/jbc.M111.325555
Max F. Perutz Laboratories (MFPL):
Die Max F. Perutz Laboratories (MFPL) sind ein gemeinsames Forschungs- und Ausbildungszentrum der Universität Wien und der Medizinischen Universität Wien am Campus Vienna Biocenter. An den MFPL beschäftigen sich rund 530 WissenschaftlerInnen aus 40 Nationen in über 60 Forschungsgruppen mit Grundlagenforschung im Bereich der Molekularbiologie.
Wissenschaftlicher Kontakt (in Englisch)
Dr. Anton Polyansky
Department für Strukturbiologie und Computational Biology
Zentrum für Molekulare Biologie
Universität Wien
1030 Wien, Campus-Vienna-Biocenter 5
T +43-1-4277-522 71
anton.polyansky@univie.ac.at
Rückfragehinweis
Mag. Veronika Schallhart
Pressebüro der Universität Wien
Forschung und Lehre
Universität Wien
T +43-1-4277-175 30
M +43-664-602 77-175 30
veronika.schallhart@univie.ac.at
Media Contact
Weitere Informationen:
http://www.univie.ac.atAlle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Ideen für die Zukunft
TU Berlin präsentiert sich vom 22. bis 26. April 2024 mit neun Projekten auf der Hannover Messe 2024. Die HANNOVER MESSE gilt als die Weltleitmesse der Industrie. Ihr diesjähriger Schwerpunkt…

Peptide auf interstellarem Eis
Dass einfache Peptide auf kosmischen Staubkörnern entstehen können, wurde vom Forschungsteam um Dr. Serge Krasnokutski vom Astrophysikalischen Labor des Max-Planck-Instituts für Astronomie an der Universität Jena bereits gezeigt. Bisher ging…

Wasserstoff-Produktion in der heimischen Garage
Forschungsteam der Frankfurt UAS entwickelt Prototyp für Privathaushalte: Förderzusage vom Land Hessen für 2. Projektphase. Wasserstoff als Energieträger der Zukunft ist nicht frei verfügbar, sondern muss aufwendig hergestellt werden. Das…