Tausende auf einem Chip: Neue Methode zur Erforschung von Proteinen
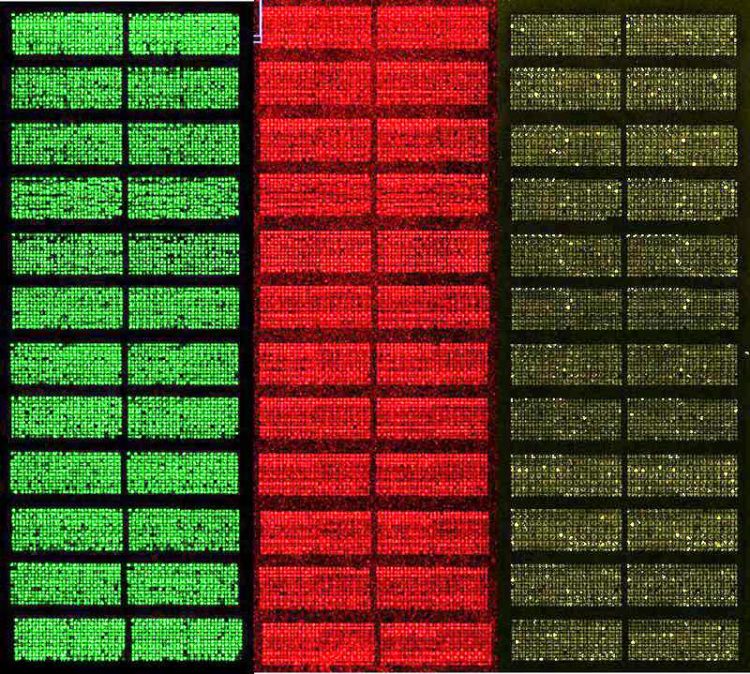
Protein-Microarrays wie hier zu sehen ermöglichen es, tausende Proteine in einem einzigen Experiment zu studieren. (Foto: TUM/Falter-Braun)
Die Genome des Menschen und der meisten gängigen Nutzpflanzen sind schon lange entschlüsselt. Bald wird es sogar möglich, sich sein persönliches Genom für weniger als 1000 Euro sequenzieren zu lassen.
Trotzdem gibt es noch zu lösende Rätsel: Für tausende der rund 20.000 bis 30.000 Gene ist nicht geklärt, was die von ihnen gebildeten Proteine im Körper machen oder welche Funktion sie ausüben. Dies macht es schwierig, viele der beim Forschen aufkommenden Daten zu interpretieren, und die zugrundeliegenden molekularbiologische Abläufe zu verstehen.
Die Funktion eines Proteins setzt sich aus vielen Aspekten zusammen: Mit welchen anderen Proteinen arbeitet es zusammen? Wie wird seine Funktion reguliert und welche Prozesse werden von ihm beeinflusst?
Selbst bei Modellpflanzen wie der Ackerschmalwand (Arabidopsis thaliana) ist die Rolle von rund 10.000 Proteinen nicht geklärt. Seit der Entschlüsselung des Genoms werden daher diese molekularen Funktionen intensiv untersucht. Jedoch mit gängigen Methoden kann diese Wissenslücke nur sehr langsam geschlossen werden.
Microarrays ermöglichen Untersuchung tausender Proteine
Protein-Microarrays (Abb.) ermöglichen es, tausende Proteine in einem einzigen Experiment zu studieren. Microarrays sind wenige Zentimeter groß und haben tausende Testfelder auf kleinstem Raum. Bei klassischen Protein-Microarrays werden in jedem Testfeld, sogenannten Spots, kleinste Protein-Mengen auf eine Glasplatte gedruckt und fixiert, wo sie für Experimente verfügbar sind.
Diese Methode erfordert jedoch, dass tausende Proteine vorab gereinigt und damit fürs Erforschen bereitgestellt werden. Das ist so zeit- und kostenaufwendig, dass Protein-Microarrays trotz ihres Potenzials bislang kaum verbreitet sind.
Pascal Falter-Brauns Arbeitsgruppe vom Lehrstuhl für die Systembiologie der Pflanzen an der TUM ist zusammen mit Wissenschaftlern aus den USA und Japan ein möglicherweise entscheidender Durchbruch gelungen: Statt Proteinen wird zunächst die einfacher verfügbare DNA (Desoxyribonukleinsäure) in hoher Dichte auf die Arrays gedruckt.
Nach dem Drucken wird der Array in einen Reaktionspuffer getaucht, also in eine Lösung, die bewirkt, dass die von der DNA gebildeten Proteine auf dem Array entstehen. Das passiert, weil die DNA die Informationen für den Aufbau von Proteinen enthält. Die so hergestellten Proteine werden sehr schnell und sehr fest von einem auf der Glasoberfläche befestigten Anker (Halo-Tag Ligand) ‚eingefangen’ und stehen nach dieser ‚Entwicklung‘ für funktionelle Untersuchungen bereit.
Das neue Verfahren wird im Englischen „nucleic acid programmable protein array“ (NAPPA – mit Nukleinsäure programmierbares Proteinarray) genannt und mit dem Namen für das Befestigungsverfahren kombiniert als HaloTag-NAPPA abgekürzt.
Durch das neue Verfahren konnten die Forscher darüber hinaus die Dichte der Proteine auf den Arrays derart erhöhen, dass es jetzt möglich ist, auf wenigen Arrays alle Proteine eines Organismus unterzubringen und ihre Funktionen zu untersuchen. Das Potenzial der neuen Proteinarrays konnten die Forscher an pflanzlichen Hormonsignalpfaden demonstrierten, die beispielsweise Reaktionen auf Trockenstress oder Schädlinge bestimmen.
1000 neue Protein-Protein-Interaktionen entdeckt
Für die jetzt in PNAS veröffentlichte Studie wurden Wechselwirkungen von 38 der wichtigsten Transkriptionsfaktor-Proteine der Ackerschmalwand untersucht. Transkriptionsfaktoren bestimmen, welche Gene zu welchem Zeitpunkt und unter welchen Bedigungen aktiv sind und haben dementsprechend eine große Bedeutung in Organismen. Diese Transktiptionsfaktoren werden ihrerseits durch Interaktionen mit anderen Proteinen aktiviert oder inaktiviert – in der vorliegenden Studie konnten mithilfe der Proteinarrays nahezu 1000 neue Proteininteraktionen für die untersuchten Faktoren identifiziert werden. „Viele der nun beobachteten Interaktionen sind bislang noch nie dokumentiert worden. Sie helfen uns dabei besser zu verstehen, wie biologische Systeme und die zugrundeliegenden molekularen Netzwerke funktionieren“, sagt Pascal Falter-Braun.
Protein-Netzwerke
Sowohl in Pflanzen als auch im Menschen agieren Proteine nicht unabhängig, sondern stehen in engen wechselseitigen Beziehungen und arbeiten zusammen in komplexen Netzwerken – der Forschungsschwerpunkt des Teams um Pascal Falter-Braun. In Organismen haben Proteine eine Schlüsselrolle und führen nahezu alle biologischen Prozesse aus. „Um zu verstehen, welche Proteine mit welchen anderen Proteinen oder aber mit anderen Molekülen der Zelle wechselwirken und kooperieren, dafür ist die neue Methodik möglicherweise ein Meilenstein“, sagt Falter-Braun. „Da sie kostengünstiger ist, kann nun eine breitere Masse an Forschern mit dieser Art von Microarrays arbeiten und die Rolle der Proteine genauer beleuchten.“
Dass die neue Methode dabei helfen wird, mit der Forschung zu Algen für erneuerbare Energien voranzukommen oder die Ursachen von Krankheiten zu beleuchten, davon ist Biochemiker Falter-Braun überzeugt.
Publikation:
Junshi Yazakia, Mary Gallia, Alice Y. Kima, Kazumasa Nitob, Fernando Alemand, Katherine N. Changb, Anne-Ruxandra Carvunise, Rosa Quana, Hien Nguyena, Liang Songb, José M. Alvarezh, Shao-shan Carol Huangb, Huaming Chena, Niroshan Ramachandrani, Stefan Altmannj, Rodrigo A. Gutiérrezh, David E. Hille, Julian I. Schroederd, Joanne Choryb, Joshua LaBaerl, Marc Vidale, Pascal Braunj and Joseph R. Eckera: Mapping transcription factor interactome networks using HaloTag protein arrays, PNAS June 2016.
DOI: 10.1073/pnas.1603229113
http://www.pnas.org/content/early/2016/06/28/1603229113.short?rss=1
Kontakt:
Dr. Pascal Falter-Braun
Technische Universität München
Lehrstuhl für die Systembiologie der Pflanzen
Emil-Ramann-Strasse 8
85354 Freising, Germany
Tel: 08161 71 5645
pbraun@wzw.tum.de
http://www.tum.de/die-tum/aktuelles/pressemitteilungen/kurz/article/33223/
Media Contact
Alle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Nanofasern befreien Wasser von gefährlichen Farbstoffen
Farbstoffe, wie sie zum Beispiel in der Textilindustrie verwendet werden, sind ein großes Umweltproblem. An der TU Wien entwickelte man nun effiziente Filter dafür – mit Hilfe von Zellulose-Abfällen. Abfall…

Entscheidender Durchbruch für die Batterieproduktion
Energie speichern und nutzen mit innovativen Schwefelkathoden. HU-Forschungsteam entwickelt Grundlagen für nachhaltige Batterietechnologie. Elektromobilität und portable elektronische Geräte wie Laptop und Handy sind ohne die Verwendung von Lithium-Ionen-Batterien undenkbar. Das…

Wenn Immunzellen den Körper bewegungsunfähig machen
Weltweit erste Therapie der systemischen Sklerose mit einer onkologischen Immuntherapie am LMU Klinikum München. Es ist ein durchaus spektakulärer Fall: Nach einem mehrwöchigen Behandlungszyklus mit einem immuntherapeutischen Krebsmedikament hat ein…





















