Proteinstruktur mittels Big Data vorhergesagt
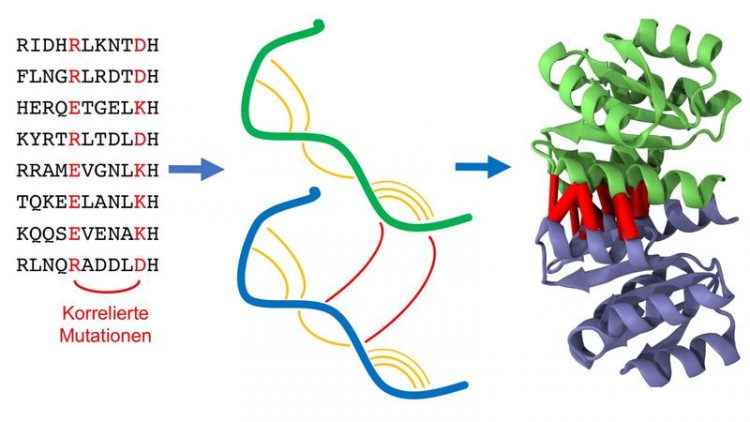
Homodimere sind identische Paare von Eiweißketten (Proteine, grün und blau), die aneinanderbinden. Die statistische Analyse der Proteinsequenzen sucht nach Mutationen, die auf eine räumliche Nähe von Proteinteilen hindeuten, sowohl innerhalb des gleichen Proteins (orange) wie auch mit dem Partnerprotein (rot). Diese Information ermöglicht es, die Proteinstruktur des Homodimers vorherzusagen. (Foto: KIT)
Wie die Forscher in der Zeitschrift Proceedings of the National Academy of Sciences of the United States of America (PNAS) berichten, ist es ihnen gelungen, auf Basis statistischer Analysen auch komplizierteste Proteinstrukturen unabhängig vom Experiment vorherzusagen.
Diese experimentell zu bestimmen ist sehr aufwändig und der Erfolg nicht garantiert. Proteine bilden die Grundlage des Lebens. Als Strukturproteine sind sie am Aufbau von Gewebe wie Nägeln oder Haare beteiligt. Andere Proteine arbeiten als Muskeln, steuern den Stoffwechsel und die Immunabwehr oder übernehmen den Sauerstofftransport in den roten Blutkörperchen
Die Grundstruktur von Proteinen mit bestimmten Funktionen ähnelt sich auch bei sehr verschiedenen Organismen „Ob Mensch, Maus, Walfisch oder Bakterium, die Natur erfindet Proteine für verschiedene Lebewesen nicht immer neu, sondern variiert sie lediglich durch evolutionäre Mutation und Selektion“, so Alexander Schug vom Steinbuch Centre for Computing (SCC). Solche Mutationen können beim Auslesen der Erbinformationen, aus denen die Proteine zusammengesetzt sind, leicht identifiziert werden.
Treten sie nun paarweise auf, liegen die beteiligten Proteinabschnitte meist räumlich nahe beieinander. Diese einzelnen Informationen vieler räumlich benachbarter Abschnitte können mit einem Computer wie ein großes Puzzle zu einer genauen Vorhersage der dreidimensionalen Struktur zusammengesetzt werden. Denn: „Um die Funktion eines Proteins wirklich im Detail zu verstehen und eventuell auch zu beeinflussen, muss man den Ort jedes einzelnen Atoms kennen“, sagt Schug.
Der gelernte Physiker nutzt in seiner Arbeit einen interdisziplinären Ansatz mit Methoden und Ressourcen der Informatik und Biochemie. Mit Hilfe von Hochleistungsrechnern hat er die frei verfügbaren Erbinformationen tausender verschiedener Organismen von Bakterien bis hin zum Menschen nach in Verbindung stehenden Mutationen durchsucht.
„Durch die Kombination von modernster Technik und einem wahren Schatz an Datensätzen konnten wir knapp 2000 verschiedene Proteine untersuchen – das ist eine völlig neue Größenordnung im Vergleich zu bisherigen Studien“, sagt Schug. Dies unterstreiche eindrucksvoll die Leistungsfähigkeit dieser Methodik und verspricht großes Potenzial für breite Anwendungen von der Molekularbiologie bis hin zur Medizin. Auch wenn die gegenwärtige Arbeit Grundlagenforschung sei, wie Schug betont, können die Ergebnisse in Zukunft beispielsweise in neue Behandlungsmethoden von Krankheiten einfließen.
Weiterer Kontakt:
Dr. Felix Mescoli, Pressereferent, Tel.: +49 721 608 48120, Fax: +49 721 608 43658, E-Mail: felix.mescoli@kit.edu
Das Karlsruher Institut für Technologie (KIT) verbindet seine drei Kernaufgaben Forschung, Lehre und Innovation zu einer Mission. Mit rund 9 300 Mitarbeiterinnen und Mitarbeitern sowie 25 000 Studierenden ist das KIT eine der großen natur- und ingenieurwissenschaftlichen Forschungs- und Lehreinrichtungen Europas.
KIT – Die Forschungsuniversität in der Helmholtz-Gemeinschaft
Das KIT ist seit 2010 als familiengerechte Hochschule zertifiziert.
Diese Presseinformation ist im Internet abrufbar unter: www.kit.edu
Media Contact
Alle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Neue Industrie-4.0-Lösung für niedrigschwelligen Zugang zu Datenräumen
»Energizing a Sustainable Industry« – das Motto der Hannover Messe 2024 zeigt klar, wie wichtig eine gleichermaßen leistungsstarke und nachhaltige Industrie für den Fertigungsstandort Deutschland ist. Auf der Weltleitmesse der…

Quantenpräzision: Eine neue Art von Widerstand
Physikforschende der Universität Würzburg haben eine Methode entwickelt, die die Leistung von Quantenwiderstands-Normalen verbessern kann. Sie basiert auf einem Quantenphänomen namens anomaler Quanten-Hall-Effekt. In der industriellen Produktion oder in der…

Sicherheitslücke in Browser-Schnittstelle erlaubt Rechnerzugriff über Grafikkarte
Forschende der TU Graz waren über die Browser-Schnittstelle WebGPU mit drei verschiedenen Seitenkanal-Angriffen auf Grafikkarten erfolgreich. Die Angriffe gingen schnell genug, um bei normalem Surfverhalten zu gelingen. Moderne Websites stellen…