Moleküle in Bewegung

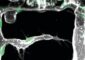
Riboschalter regulieren die Biosynthese und den Transport des Metaboliten Thiaminpyrophosphat (TPP). Grafik: Uni Innsbruck<br>
Forscher aus Innsbruck und New York setzen ein hochmodernes Verfahren ein, mit dem sie das dynamische Verhalten einzelner Biomoleküle sehr genau ermitteln können. Es liefert wichtige Einsichten in die Funktionsweise von Genschaltern.
Die DNA hat eine kleine Schwester, die Boten-RNA. Diese transportiert die Erbgut-Information in die Proteinfabriken der Zelle. Im Jahr 2000 entdeckten Wissenschaftler um Ronald R. Breaker, dass die Boten-RNA Kontrollelemente enthalten kann, mit denen diese Moleküle ihr eigenes Gen ein- oder ausschalten können. Bakterien dient dies zum Beispiel dazu, viele Stoffwechselvorgänge zu regulieren. So passen sie ihre Produktionsmaschinerie dem aktuellen Bedarf in einer Zelle an. Diese sogenannten Riboschalter haben auch das Interesse der Arbeitsgruppe um Prof. Ronald Micura am Institut für Organische Chemie und dem Centrum für Molekulare Biowissenschaften der Universität Innsbruck (CMBI) geweckt. Gemeinsam mit Wissenschaftlern des Weill Cornell Medical College in New York nutzen sie eine neue Technik, den Single-Molecule Fluorescence Resonance Energy Transfer (smFRET), um die Dynamik einzelner Riboschalter-Moleküle zu untersuchen. „Die biologische Aktivität eines Molekül erschließt sich selten nur aus der chemischen Struktur“, sagt Ronald Micura. „Entscheidend ist meist, wie sich diese Struktur im Laufe der Zeit ändert, also die Beweglichkeit des Moleküls.“
Know-how aus Tirol
Die Chemiker um Ronald Micura sind weltweit führend bei der synthetischen Herstellung und Modifizierung von Biomolekülen. Mit üblichen Syntheseverfahren ist es nämlich kaum möglich, mehr als 50 Basenbausteine gezielt zusammenzusetzen. Micura und seine Mitarbeiter haben ein raffiniertes Verfahren entwickelt, mit dem sie chemisch synthetisierte RNA-Teile nach Belieben kombinieren können. Sie greifen dabei auf einen Trick der Natur zurück: Bestimmte Enzyme können RNA-Strangbrüche reparieren, indem sie die Teile durch chemische Bindungen wieder aneinanderfügen. Bietet man diesen Enzymen die künstlich hergestellte RNA an, knüpfen sie auch daraus lange Ketten. So bilden die Chemiker natürliche Riboschalter nach und markieren diese an den entscheidenden Stellen mit Farbstoffen. Im Labor der New Yorker Forscher werden diese oft in monatelanger Feinarbeit hergestellten Riboschalter dann mit Hilfe von Laserlicht analysiert.
Auf größere biomolekulare Maschinen ausweiten
In der amerikanischen Fachzeitschrift Proceedings of the National Academy of Sciences (PNAS) haben die Forscher nun Ergebnisse der Untersuchung eines der verbreitetsten Riboschalter-Moleküle veröffentlicht. Diese kommen in Bakterien, Pflanzen und Pilzen vor und regulieren die Biosynthese und den Transport des Metaboliten Thiaminpyrophosphat (TPP), einem Abkömmling von Vitamin B1. Mit dem neuen Verfahren können die Forscher die Bewegungen einzelner Moleküle im Millisekundentakt beobachten. „Wir waren sehr überrascht zu sehen, wie beweglich die beiden Arme des Moleküls sind, welche in der Kristallstruktur eine starre Interaktion implizieren“, erzählt Micura. „Dort liegt auch die Bindetasche des Metaboliten, der an die RNA andockt und das entsprechende Gen abschaltet.“ Möglich ist diese genaue Beobachtung nur durch die selektive Modifizierung der RNA-Moleküle in den Innsbrucker Labors. Micura und sein amerikanischer Partner haben sich das entsprechende Verfahren in der Zwischenzeit auch patentieren lassen und wollen nun in einem gemeinsamen, von der National Science Foundation NSF und dem Wissenschaftsfonds FWF geförderten Projekt ihre Techniken auch auf noch größere Biomoleküle ausweiten. „Wir wollen den gesamten Translations-Mechanismus – als jene Maschinerie, die aus Erbgutinformation Proteine erzeugt – untersuchen“, blickt Micura bereits in die Zukunft.
Publikation: Folding and ligand recognition of the TPP riboswitch aptamer at single-molecule resolution. Andrea Haller, Roger B. Altman, Marie F. Soulière, Scott C. Blanchard, and Ronald Micura. PNAS 2013 DOI: 10.1073/pnas.1218062110
Rückfragehinweis:
Ronald Micura
Institut für Organische Chemie
Universität Innsbruck
Tel.: +43 512 507-57710
E-Mail: Ronald.Micura@uibk.ac.at
Christian Flatz
Büro für Öffentlichkeitsarbeit
Universität Innsbruck
Tel.: +43 512 507 32022
Mobil: +43 676 872532022
E-Mail: christian.flatz@uibk.ac.at
Media Contact
Weitere Informationen:
http://www.uibk.ac.atAlle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Ideen für die Zukunft
TU Berlin präsentiert sich vom 22. bis 26. April 2024 mit neun Projekten auf der Hannover Messe 2024. Die HANNOVER MESSE gilt als die Weltleitmesse der Industrie. Ihr diesjähriger Schwerpunkt…

Peptide auf interstellarem Eis
Dass einfache Peptide auf kosmischen Staubkörnern entstehen können, wurde vom Forschungsteam um Dr. Serge Krasnokutski vom Astrophysikalischen Labor des Max-Planck-Instituts für Astronomie an der Universität Jena bereits gezeigt. Bisher ging…

Wasserstoff-Produktion in der heimischen Garage
Forschungsteam der Frankfurt UAS entwickelt Prototyp für Privathaushalte: Förderzusage vom Land Hessen für 2. Projektphase. Wasserstoff als Energieträger der Zukunft ist nicht frei verfügbar, sondern muss aufwendig hergestellt werden. Das…