Bioinformatik-Strategie erfolgreich bei der Strukturaufklärung von Membranproteinen
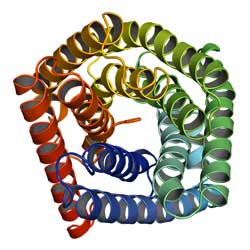
Durch Homology Modelling ermittelte Struktur des SLAC1-Anionen-Kanals. Yu-Hang Chen et al., Columbia University, New York<br>
Doch obwohl sie eine hohe biologische und pharmazeutische Bedeutung besitzen, gelang es bisher nur in wenigen Fällen, ihre genaue Struktur und Funktionsweise aufzuklären.
Mit einem neuen bioinformatischen Ansatz gelang es nun Forschern der Columbia University, New York, und der Technischen Universität München (TUM), über die Analyse von verwandten Proteinen die Struktur eines wichtigen Ionenkanals aufzuklären. Über ihre Ergebnisse berichtet die aktuelle Ausgabe des Journals Nature.
Proteine sind molekulare Maschinen, die Stoffe transportieren, chemische Reaktionen katalysieren, Ionen pumpen und Signalstoffe erkennen. Sie setzen sich aus einer Vielzahl von Aminosäuren zusammen. Die Abfolge der einzelnen Aminosäuren, die Aminosäuresequenz, ist bei einer Vielzahl von Proteinen bekannt. Doch welche Aufgaben ein Protein in der Zelle ausführen kann, entscheidet der dreidimensionale Aufbau des Proteins.
Die Bestimmung dieser so genannten Tertiärstruktur stellt Forscher vor große Herausforderungen. Deshalb gibt es bei der Strukturanalyse einen enormen Nachholbedarf. Um hier voranzukommen, investierte das National Institute of General Medical Sciences (NIGMS) der Nationalen Gesundheitsinstitute der Vereinigten Staaten (NIH) im Rahmen der Proteinstrukturinitiative in den letzten zehn Jahren mehr als 500 Millionen Dollar, da es sich davon entscheidende Fortschritte für die Medizin und die Biologische Forschung erhofft.
TUM-Informatikprofessor Burkhard Rost und Marco Punta, Carl von Linde Junior Fellow am Institute for Advanced Study (TUM-IAS) der TU München, beteiligen sich an diesem Großprojekt. Sie sind an das New York Consortium on Membrane Proteine Structure (NYCOMPS) angeschlossen, das zu einem der neun geförderten Membran-Forschungszentren gehört. Im besonderen Fokus der NYCOMPS-Wissenschaftler stehen Membranproteine. Der Grund: In der pharmakologischen Forschung nehmen sie eine Schlüsselrolle ein. Wenn Arzneiwirkstoffe in die Zellen eintreten, interagieren diese normalerweise zuerst mit Membranproteinen. Die Kenntnis der Proteinstruktur ist notwendig, um diese Wechselwirkung auf molekularer Ebene zu verstehen.
Allerdings ist die experimentelle Strukturaufklärung gerade für die so wichtigen Membranproteine extrem schwierig. Viele Membranproteine lassen sich nur schwer in Bakterien herstellen, auch die Reinigung und das Kristallisieren sind eine große Herausforderung. Die Folge: Einerseits sind etwa 25 Prozent aller Proteine Membranproteine, andererseits beträgt ihr Anteil an der Gesamtzahl der Proteine, deren Struktur aufgeklärt werden konnte, weniger als ein Prozent. Somit sind die Membranproteinstrukturen 25-fach unterrepräsentiert; eigentlich sollten sie auf Grund ihrer medizinischen Bedeutung besser bekannt sein.
Da die experimentelle Analyse eines Membranproteins teilweise mehrere Jahre dauert, nutzen die NYCOMPS-Wissenschaftler eine bioinformatische Methode, das sogenannte Homology Modeling. Die grundlegende Annahme dieser Methode ist, dass Proteine mit gemeinsamen evolutionären Vorfahren sich sowohl in ihren Aminosäuresequenzen als auch in ihrer dreidimensionalen Struktur gleichen. Kann eines dieser verwandten Proteine experimentell bestimmt werden, lassen sich die restlichen vorhersagen.
Im Fall des bakteriellen Membranproteins TehA konnten sie alle Puzzleteile zusammenfügen.
„In einem aufwändigen Screening haben wir durch den Vergleich Zehntausender Aminosäuresequenzen verwandte Membranproteine von TehA gesucht. In einem mehrstufigen Auswahlprozess wählten wir 43 verwandte Proteine von 38 verschiedenen Organismen aus“, sagt der TUM-Bioinformatiker Marco Punta.
Wissenschaftlern der Columbia Universität gelang es nun, die Tertiärstruktur des Membranproteins TehA des Bakteriums Haemophilus influenzae experimentell mittels Kristallstrukturanalyse bestimmten. Mit einer Auflösung von 0,12 Nanometern (1,2 Ångstrøm) ist die Struktur eine der besten Kristallstrukturen, die jemals für ein Membranprotein erzielt wurde. Darüber hinaus eröffnete das Experiment eine Überraschung: Das TehA-Membranprotein weist eine bislang völlig unbekannte Faltung auf.
Nachdem die „TehA-Familie“ bekannt war, konnten die Informatiker der Columbia University die Strukturen der einzelnen Proteine ableiten. Beispielhaft realisierten sie das für das pflanzliche Membranprotein SLAC1. Durch den Vergleich mit der experimentell ermittelten Proteinstruktur von TehA konnten sie eine Strukturvorhersage von SLAC1 treffen – ganz ohne Experiment, nur mit Hilfe von bioinformatischen Methoden.
„Durch dieses Verfahren haben wir eine hohe Durchsatzrate in der Strukturbestimmung. In kürzerer Zeit können wir mehr Proteine identifizieren – das war unser Ziel vor allem für die Membranproteine. Die vorliegenden Ergebnisse zeigen, dass wir mit unserer Strategie auch bei den Membranproteinen richtig liegen“, so Burkhard Rost.
Letztlich werden die dreidimensionalen Strukturen erfasst, um – mit Hilfe von Mutagenese-Tests – die Funktion der Proteine zu bestimmen. Obwohl die Membranproteine TehA und SLAC1 nur entfernt miteinander verwandt sind – die Übereinstimmung der Aminosäuresequenz liegt nur bei 19 Prozent vor – war die vorhergesagte SLAC1-Tertiärstruktur so gut, dass eine neue Hypothese über die Funktion des SLAC1-Membranproteins vorgelegt werden konnte.
SLAC1 befindet sich in den Spaltöffnungen (Stomata) der Pflanze Arabidopsis thaliana. Spaltöffnungen kontrollieren den Austausch von Wasserdampf und dem für die Photosynthese wichtigen Kohlendioxid zwischen der Pflanze und ihrer Umgebung. Dabei spielt auch das Membranprotein SLAC1 als Teil eines Anionenkanals eine Rolle: Es beeinflusst den Turgordruck – den Druck der Zellflüssigkeit auf die Zellwand – und darüber den Gasaustausch der Pflanzenzelle als Reaktion auf Umwelteinflüsse wie Trockenheit und hohe Kohlendioxid-Konzentrationen.
Ungeklärt war, wie die Ionen im Kanal transportiert werden. Insbesondere war mitten im Kanal eine Aminosäure entdeckt worden, die den Weg blockiert. Die Ergebnisse deuten darauf hin, dass diese Aminosäure aus dem Weg klappt, wenn der Kanal durch das Andocken eines weiteren Proteins aktiviert wird.
Original-Publikation:
Chen Y.-h. et al. Homologue structure of the SLAC1 anion channel for closing stomata in leaves Nature 467: 1074-1080, 2010 – DOI: 10.1038/nature09487
Kontakt:
Prof. Dr. Burkhard Rost
Technische Universität München
Institut für Informatik
Boltzmannstr. 3, 85748 Garching, Germany
Tel.: +49 89 289 17808 – E-Mail: rost@in.tum.de
Media Contact
Alle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Nanofasern befreien Wasser von gefährlichen Farbstoffen
Farbstoffe, wie sie zum Beispiel in der Textilindustrie verwendet werden, sind ein großes Umweltproblem. An der TU Wien entwickelte man nun effiziente Filter dafür – mit Hilfe von Zellulose-Abfällen. Abfall…

Entscheidender Durchbruch für die Batterieproduktion
Energie speichern und nutzen mit innovativen Schwefelkathoden. HU-Forschungsteam entwickelt Grundlagen für nachhaltige Batterietechnologie. Elektromobilität und portable elektronische Geräte wie Laptop und Handy sind ohne die Verwendung von Lithium-Ionen-Batterien undenkbar. Das…

Wenn Immunzellen den Körper bewegungsunfähig machen
Weltweit erste Therapie der systemischen Sklerose mit einer onkologischen Immuntherapie am LMU Klinikum München. Es ist ein durchaus spektakulärer Fall: Nach einem mehrwöchigen Behandlungszyklus mit einem immuntherapeutischen Krebsmedikament hat ein…





















