Ein Protein nimmt Maß: Wie Teile einer molekularen Maschine passgenau zusammengefügt werden

Der untersuchte Molekülkomplex ist Teil des „Spleißosoms“, das eine zentrale Rolle bei der Genexpression in höheren Lebewesen spielt. (Science, 6. April 2007)
Gemeinsamkeit macht stark – dieses Prinzip gilt auch für Moleküle in lebenden Zellen. Die meisten biochemischen Prozesse werden dort von Verbänden biologischer Makromoleküle vermittelt. Einige solcher Molekülverbände sind mit zahlreichen Komponenten wahre Giganten des zellulären Nanokosmos. Um die Funktionsweise dieser molekularen Maschinen zu verstehen, ist es notwendig, ihre Konstruktion im atomaren Detail aufzuklären, vergleichbar mit der Erstellung einer technischen Zeichnung.
Das Spleißosom ist eine solche molekulare Maschine, die sich aus über hundert Proteinkomponenten und einer kleineren Zahl von RNA-Molekülen zusammensetzt. Spleißosomen bewerkstelligen einen wichtigen Schritt bei der Genexpression in höheren Organismen. Hierbei müssen aus einer „prä-mRNA“, der Abschrift eines Gens der DNA, bestimmte Teile, die keine Information für die Proteinproduktion tragen, herausgeschnitten werden; die Protein-kodierenden Bereiche hingegen müssen präzise zusammengefügt werden. Durch dieses „prä-mRNA-Spleißen“ entsteht eine reife mRNA (für engl. messenger ribonucleic acid), die als Grundlage für die Proteinproduktion dient.
Eine Besonderheit des Spleißosoms ist, dass es für jeden Spleißvorgang auf einer prä-mRNA neu zusammengesetzt wird. Hierzu sind die Mehrzahl der spleißosomalen Protein- und RNA-Komponenten bereits im voraus zu Untereinheiten, sog. snRNPs (für engl. small nuclear ribonucleoprotein particles), zusammengefügt, die als Baumodule fungieren. Drei dieser Module sind im U4/U6-U5 tri-snRNP zusammengefasst, das die größte Baueinheit des Spleißosoms bildet. Wenn alle Untereinheiten auf einer prä-mRNA zusammengekommen sind, werden molekulare Umbaumaßnahmen vorgenommen. Erst in deren Folge entsteht eine funktionstüchtige Spleißmaschinerie. Insbesondere muss das U4 snRNP von den U6 und U5 snRNPs abgetrennt und entfernt werden. Das Spleißosom wird dabei gleichsam entriegelt. Auf diese Weise wird der Startschuss zur Spleißreaktion erst gegeben, wenn alle Komponenten ihre funktionalen Positionen eingenommen haben.
Bei der Aktivierung des Spleißosoms könnten zwei Proteine an der Schnittstelle der U4, U6 und U5 snRNPs eine wichtige Rolle spielen: das „15.5K“-Protein, das direkt an ein RNA-Molekül (die U4 snRNA) bindet, und das „Prp31“-Protein, das eine molekulare Brücke vom U4 zum U5 snRNP schlägt. Wissenschaftler am Göttinger Max-Planck-Institut für biophysikalische Chemie haben sich nun diesen Teil der Spleißmaschinerie genauer angeschaut. Die Abteilung von Prof. Reinhard Lührmann hatte durch biochemische Analysen herausgefunden, dass erst nach der Bindung des 15.5K-Proteins an die U4 snRNA der Einbau weiterer Proteinmoleküle, darunter Prp31, vonstatten gehen kann. Solche geordneten Aufbauwege waren bereits aus anderen Protein-RNA-Komplexen bekannt. Dort fungiert das zuerst eingebaute Protein als eine Art Ordnungshüter, der die RNA in eine bestimmte Form bringt. An diese formierte RNA können dann die später bindenden Proteine andocken.
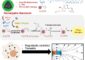
Erste Hinweise, dass im gegebenen Fall ein anderes Prinzip den Aufbau steuert, lieferte die Kernspinresonanzspektroskopie (NMR). Die Gruppe von Dr. Teresa Carlomagno stellte über dieses Verfahren fest, dass das 15.5K-Protein selbst einen Teil der Bindeoberfläche für das Prp31 zur Verfügung stellt. Die Arbeitsgruppe um Dr. Markus Wahl konnte anschließend ein genaues atomares Bild des Molekülverbandes über die Röntgenkristallographie anfertigen (siehe Abbildung). Details dieses Bildes wurden durch weitere biochemische Tests in der Lührmann-Gruppe, z.B. durch die Analyse gezielt veränderter Komponenten, untermauert. Nach den neuen Befunden wirkt das 15.5K-Protein (in der Abbildung rot) formgebend auf einen Teil der RNA (gelb) und präsentiert so dem Prp31 (blau) eine kombinierte Protein-RNA-'Landeplattform'. Im Kernbereich dieser Landeplattform passt das Prp31 zu der kombinierten Protein-RNA-Grundlage wie ein Schlüssel zum Schloss. Einen weiteren, zunächst unstrukturierten Bereich der RNA bringt das Prp31 hingegen erst bei seinem Einbau in eine stabile Form. Beim Andocken nimmt Prp31 genau Maß – es kann nur binden, wenn die RNA eine bestimmte Länge aufweist. So „weiß“ Prp31, wo genau in der Zelle es „hingehört“.
Die Aufklärung von Struktur-Funktionsbeziehungen zwischen biologischen Makromolekülen bildet eine wichtige Grundlage für unser Verständnis biochemischer Prozesse in lebenden Zellen. Forschungsergebnisse wie hier dargestellt sind aber nicht nur aus grundlagenwissenschaftlicher Sicht wichtig. Beispielsweise ist eine große Zahl menschlicher Erbkrankheiten auf fehlgeleitetes prä-mRNA-Spleißen zurückzuführen. Es besteht die Hoffnung, dass durch eine genauere Kenntnis der molekularen Maschinerie, die diesem zellulären Prozess zugrunde liegt, letztendlich auch ein besserer Einblick in Krankheitsmechanismen möglich wird.
Originalveröffentlichung:
Sunbin Liu, Ping Li, Olexandr Dybkov, Stephanie Nottrott, Klaus Hartmuth, Reinhard Lührmann, Teresa Carlomagno, and Markus C. Wahl: Binding of the Human Prp31 Nop Domain to a Composite RNA-Protein Platform in U4 snRNP. Science 316, 115-120 (6. April 2007)
Weitere Informationen:
Dr. Markus Wahl, Max-Planck-Institut für Biophysikalische Chemie, FG Makromolekulare Röntgenkristallographie, Am Faßberg 11, D-37077 Göttingen, Tel: 0551 201-1046, Fax: -1197, eMail: mwahl@gwdg.de
http://www.mpibpc.mpg.de/research/ags/wahl/
Dr. Teresa Carlomagno, Max-Planck-Institut für Biophysikalische Chemie, FG Flüssig-NMR-Spektroskopie, Am Faßberg 11, D-37077 Göttingen, Tel: 0551 201-2214, Fax: -2202, eMail: taco@nmr.mpibpc.mpg.de
http://www.mpibpc.mpg.de/research/ags/carlomagno/
Prof. Dr. Reinhard Lührmann, Max-Planck-Institut für biophysikalische Chemie, Abt. Zelluläre Biochemie, Am Faßberg 11, D-37077 Göttingen, Tel: 0551 201-1405, Fax: -1197, eMail: reinhard.luehrmann@mpi-bpc.mpg.de
http://www.mpibpc.gwdg.de/research/dep/luehrmann/
Media Contact
Alle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Merkmale des Untergrunds unter dem Thwaites-Gletscher enthüllt
Ein Forschungsteam hat felsige Berge und glattes Terrain unter dem Thwaites-Gletscher in der Westantarktis entdeckt – dem breiteste Gletscher der Erde, der halb so groß wie Deutschland und über 1000…

Wasserabweisende Fasern ohne PFAS
Endlich umweltfreundlich… Regenjacken, Badehosen oder Polsterstoffe: Textilien mit wasserabweisenden Eigenschaften benötigen eine chemische Imprägnierung. Fluor-haltige PFAS-Chemikalien sind zwar wirkungsvoll, schaden aber der Gesundheit und reichern sich in der Umwelt an….

Das massereichste stellare schwarze Loch unserer Galaxie entdeckt
Astronominnen und Astronomen haben das massereichste stellare schwarze Loch identifiziert, das bisher in der Milchstraßengalaxie entdeckt wurde. Entdeckt wurde das schwarze Loch in den Daten der Gaia-Mission der Europäischen Weltraumorganisation,…