Ein Fall von Zell-Spionage
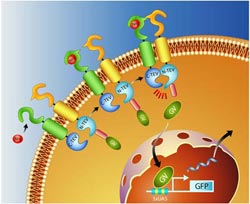
Schematische Darstellung der transkriptionsabhängigen Variante des Split-TEV Verfahrens. Der rote Auftraggeber "Nrg" dockt an einem grünen Transmembranprotein an, das sich daraufhin mit einem zweiten Membranprotein (gelb) verbindet, um die Information weiterzuleiten. Dadurch fügt sich auch das Späherenzym an der Zellinnenseite (blau) zusammen und setzen den Transkriptionsspion (GV) frei, der aus der DNA ein fluoreszierendes Molekül synthetisieren lässt. Das sorgt dann für das detektierbare Signal. Bild: Max-Planck-Institut für experimentelle Medizin
Ohne Richtmikrofon und Revolver, aber mit Methoden der Spionage erkunden Wissenschaftler des Max-Planck-Instituts für experimentelle Medizin in Göttingen und der Axaron Bioscience AG in Heidelberg die Informationswege in der Zelle. Mit Hilfe von Spähermolekülen übersetzen die Forscher die Interaktionen löslicher Proteine und komplexer Membranproteine in Lichtsignale, die leicht zu detektieren sind. Auf diese Weise können sie auch in bisher experimentell schwer zugänglich Zellen, etwa Neuronen oder Gliazellen, untersuchen, welche Proteine wann miteinander kommunizieren. Mit dem Verfahren lassen sich Krankheiten erforschen, die unter anderem durch fehlgeleitete Informationen in Zellen ausgelöst werden, wie beispielsweise Krebs oder neurodegenerative Erkrankungen (Nature Methods Dezember 2006, online: 29. Okt. 2006).
Häufig teilen Nachrichtenmoleküle Zellen ihre Aufgaben mit – beispielsweise sich zu teilen oder bestimmte Enzyme zu produzieren. Membranproteine an der Außenseite der Zelle empfangen diese Aufträge, lösliche Proteine leiten sie im Zellinneren weiter. Ist die Kommunikation in der Zelle oder zwischen Zellen gestört, können unter Umständen Krebs oder neurodegenerative Erkrankungen entstehen. Die Therapie dieser Krankheiten würde leichter, wenn Mediziner mehr über diese Informationswege wüssten. Ein Wissenschaftlerteam vom Max-Planck-Institut für experimentelle Medizin in Göttingen und der Axaron Bioscience AG in Heidelberg hat nun eine neue Methode namens Split-TEV entwickelt, um die Stillepost von Protein zu Protein abzuhören. Das Verfahren vereinfacht es enorm, die Interaktion sowohl von löslichen Proteinen als auch von komplexen Membranproteinen zu untersuchen.
Insbesondere die Membranproteine waren bisher nur sehr eingeschränkt zu analysieren. Sie sind besonders interessant, weil an ihnen viele wichtige Nachrichtenkaskaden beginnen und potenzielle Pharmazeutika besonders gut angreifen können. Die sehr empfindliche Methode eignet sich auch, um Zellen zu analysieren, die direkt aus lebendem Gewebe entnommen wurden. Diese sogenannten Primärzellen sind zwar aufwändiger zu handhaben als Zellen, die extra für die Forschung im Labor gezüchtet wurden. Sie stellen aber auch ein weitaus aussagekräftigeres biologisches System dar. Im Gegensatz zu früheren Methoden lassen sich mit der Split-TEV-Technik überdies in automatisierten Analyseverfahren (high-throughput screenings) viele der zahlreichen Proteine und externen Faktoren gleichzeitig testen, die an den Nachrichtenkaskaden in der Zelle beteiligt sind.
Die Forscher greifen bei ihren Untersuchungen auf Tricks der Spionagetechnik zurück: Sie schleusen zwei Spähermoleküle in die Zelle ein. Die beiden Moleküle bilden zusammen ein Enzym des Tabakvirus TEV, das erst aktiv wird, wenn die beiden Teile einander nahe kommen. Die Enzymhälften heften sich nun an zwei zuvor bestimmte Zielproteine. Sobald diese sich treffen, nähern sich auch ihre Späheranhängsel und werden dadurch zu einem funktionsfähigen Enzym. Das vollständige Enzym aktiviert anschließend ein weiteres Protein – ein fluoreszierendes Reportermolekül, das bis jetzt an einem der Agentenmoleküle hing, nun aber abgestoßen wird. Dabei fängt es an zu leuchten und signalisiert den Forschern so: Die Proteine reden miteinander!
Bei einer Variante des Verfahrens leuchtet das Reportermolekül nicht selber. Stattdessen wandert es in den Zellkern und aktiviert ein Gen, das ein fluoreszierendes Molekül kodiert. Die Forscher müssen dieses extra zu diesem Zweck schon vorher in die DNA eingebaut haben. Bei dieser Version gibt das Reportermolekül also nur indirekt ein Signal ab, weil das produzierte Fluoreszenzmolekül leuchtet. Dieses transkriptionsabhängige Verfahren, ist zwar etwas aufwändiger als das direkte, dafür aber erheblich empfindlicher. Es wandelt selbst vorübergehende, kurze Interaktionen in ein andauerndes, deutliches Signal um.
Die Wissenschaftler setzten viele verschiedene fluoreszierende und lumineszierende Reportermoleküle beziehungsweise Gene für zahlreiche Leuchtproteine ein. So können Forscher das Split-TEV-Verfahren sehr flexibel an die vorhandene Analysetechnik und das jeweils untersuchte Proteinsystem anpassen. „Außerdem stehen die für die Aufnahme und Analyse der Lichtsignale benötigten Geräte in den meisten Laboren ohnehin zur Verfügung – ein großer Kostenvorteil für das Verfahren“, sagt Moritz Rossner, der Projektleiter.
Seine Arbeitsgruppe will sich vor allem auf die Interaktionen von Neuronen und Gliazellen konzentrieren. Denn mit der Split-TEV Technik ist nun auch die intra- und interzelluläre Kommunikation dieser experimentell schwer zugänglichen Zelltypen gut zu untersuchen. „Außerdem wollen wir das System in Zukunft so weiterentwickeln, dass wir ausgedehnte Netze miteinander kommunizierender Proteine analysieren können“, sagt Moritz Rossner.
Originalveröffentlichung:
Michael C. Wehr, Rico Laage, Ulrike Bolz, Tobias M. Fischer, Sylvia Grünewald, Sigrid Scheek, Alfred Bach, Klaus-Armin Nave & Moritz J. Rossner
Monitoring regulated protein-protein interactions using split TEV
Nature Methods, Dezember 2006, online: 29. Okt. 2006
Media Contact
Weitere Informationen:
http://www.mpg.deAlle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Bakterien für klimaneutrale Chemikalien der Zukunft
Forschende an der ETH Zürich haben Bakterien im Labor so herangezüchtet, dass sie Methanol effizient verwerten können. Jetzt lässt sich der Stoffwechsel dieser Bakterien anzapfen, um wertvolle Produkte herzustellen, die…

Batterien: Heute die Materialien von morgen modellieren
Welche Faktoren bestimmen, wie schnell sich eine Batterie laden lässt? Dieser und weiteren Fragen gehen Forschende am Karlsruher Institut für Technologie (KIT) mit computergestützten Simulationen nach. Mikrostrukturmodelle tragen dazu bei,…

Porosität von Sedimentgestein mit Neutronen untersucht
Forschung am FRM II zu geologischen Lagerstätten. Dauerhafte unterirdische Lagerung von CO2 Poren so klein wie Bakterien Porenmessung mit Neutronen auf den Nanometer genau Ob Sedimentgesteine fossile Kohlenwasserstoffe speichern können…