Leben ohne Mund, Magen und Darm
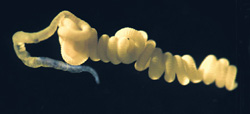
Olavius algarvensis unter dem Mikroskop <br>Bild: MPI Bremen und Hydra Institut, Elba, C. Lott
Zahlreiche Pflanzen und Tiere – einschließlich des Menschen – werden von Mikroorganismen besiedelt, die nützliche Funktionen (z.B. die Erschließung oder Verdauung von Nährstoffen) für ihre Wirte übernehmen. Im Gegenzug stellt ihnen der Wirt geeignete Lebensbedingungen zur Verfügung. Solche engen gegenseitigen Vorteilsgemeinschaften nennt man Symbiosen. Forschern vom Max-Planck-Institut für marine Mikrobiologie in Bremen und vom Joint Genome Institute in den USA ist es jetzt gelungen, die Genome von gleich vier bakteriellen Symbionten aus dem Inneren des marinen Wurms Olavius algarvensis mithilfe einer so genannten Metagenomanalyse zu entschlüsseln. Das ist die bislang größte Studie dieser Art an einer symbiontischen Lebensgemeinschaft. Sie bildet einen wichtigen Grundstein für die Analyse anderer komplexer Symbiosen, wie zum Beispiel der Endflora des menschlichen Darms (Nature, Advanced Online Publication, 17. September 2006).
Olavius algarvensis ist ein mariner Oligochaet (Wenigborster), der in den oberen zwanzig Zentimetern im sandigen Meeresboden der flachen Küstengewässer vor der Mittelmeerinsel Elba beheimatet ist. Nicole Dubilier und ihre Mitarbeiter aus der Symbiosegruppe vom Max-Planck-Institut in Bremen forschen seit Jahren auf diesem Gebiet. Die anatomische Besonderheit des Wurms ist, dass er nicht nur sein Verdauungssystem komplett reduziert hat – also weder Mund, Magen noch Darm hat – sondern auch keine nierenähnliche Organe (Nephridien) besitzt. Während die Reduktion des Verdauungssystems als Anpassung an symbiontische Mikroorganismen auch von anderen Tieren bekannt ist, sind darmlose Oligochaeten die einzige bekannte Wirtsgruppe, die auch ihre Ausscheidungssysteme reduziert haben. Das bedeutet für den Wurm, dass alle Prozesse, die mit Nahrungsaufnahme und Abfallentsorgung zu tun haben, von seinen Symbionten erledigt werden müssen. Die Forscher wollten nun herausfinden, wie diese wesentlichen Wirtsaufgaben an die Symbionten ausgelagert werden konnten, schließlich handelt es sich hierbei um ein wunderbares Beispiel für „Outsourcing“ von Energiegewinn und Abfallentsorgung.
Eine Symbiose detailliert zu untersuchen, ist oftmals eine besondere Herausforderung, da sich die meisten symbiontischen Mikroorganismen nicht isoliert züchten lassen. Mit der so genannten Metagenomanalyse ist es jedoch möglich, die Einzel-Genome (das Genom ist die Gesamtheit der Gene eines Organismus) verschiedener Organismen zu isolieren, ohne die Organismen selbst zu isolieren. Doch, wie lässt sich das Gemisch an Genomen aus der Umweltprobe einzelnen Arten zuordnen? In der klassischen Genomanalyse sequenziert man die Erbsubstanz einer bestimmten Art mithilfe etablierter Methoden, und jedes Jahr publizieren die Wissenschaftler in den Datenbanken hunderte verschiedener Genome. Der klassische Ansatz funktioniert jedoch nicht bei einem Gemisch von verschiedenen Organismen, denn die Zuordnung der Sequenzen ist nicht klar erkennbar. Dieses Problem lässt sich an einem Beispiel aus der Textanalyse veranschaulichen: Man stelle sich vor, die Bücher verschiedener Autoren seien hoffnungslos durcheinander geraten. Die Texte liegen nur noch in Bruchstücken vor. Aufgabe ist es nun, die ursprünglichen Werke wiederherzustellen. Da jeder Autor einen anderen Schreibstil bevorzugt, kann man die Ursprungstexte mithilfe einer statistischen Analyse der Bruchstücke rekonstruieren. Im Genom-„Text“ gibt es jedoch nur vier verschiedene Buchstaben A, G, C, T. Und diese Buchstaben hängen ohne „Punkt“ und „Komma“ aneinander.
Hanno Teeling aus der Arbeitsgruppe Mikrobielle Genomik von Prof. Frank Oliver Glöckner gelang es nun, mit einem neuen mathematischen Algorithmus, einem Binning-Verfahren, dieses Problem zu lösen. Die relativen Häufigkeiten aller 64 möglichen Dreiergruppen von A, G, C und T, aller 256 möglichen Viererkombinationen der Bausteine und die Häufigkeit von G und C innerhalb eines genormten Genomabschnitts unterscheiden sich deutlich je nach Organismenart. Damit ließen sich die Bruchstücke in einzelne Untergruppen, so genannte Bins, differenzieren. Die Fragmente konnten zusammengesetzt, gelesen und somit die einzelnen Genome rekonstruiert werden. Damit hatten die Forscher den Schlüssel in der hand, um die jeweiligen Stoffwechsel der Symbionten zu rekonstruieren und zu zeigen, welche Stoffwechselwege je nach Umwelteinfluss aktiviert werden können.
Das Ergebnis: Zwei Schwefelbakterien (Gammaproteobakterien) und zwei Sulfatreduzierer (Deltaproteobakterien) kommen gemeinsam in dem Wurm vor. Die Sulfatreduzierer produzieren reduzierte Schwefelverbindungen, die die Schwefeloxidierer als Energiequelle verwenden können. So füttern sich die Symbionten in einem syntrophen Schwefelzyklus gegenseitig. Überraschenderweise können alle vier Symbionten wie Pflanzen Kohlendioxid fixieren, der Wurm hat sich also ein regelrechtes endosymbiotisches Kraftwerk zugelegt. An der Zersetzung von giftigen Stoffwechselendprodukten wie Harnstoff und Ammonium sind auch alle vier Symbionten beteiligt und tragen damit zum Recycling von wertvollem Stickstoff bei.
„Der kleine Wurm macht vor, wie begrenzte Ressourcen durch das Zusammenwirken von aufeinander abgestimmten Mikrobengemeinschaften auf kleinstem Raum effizient genutzt werden können“ sagt Nicole Dubilier. So könnte die Olavius-Symbiose ein Modell für eine sich nahezu selbst erhaltende Biosphäre sein. Vergleichbare Systeme im größeren Maßstab werden intensiv erforscht, um zum Beispiel längere interplanetare Raumfahrten wie die geplante Reise zum Mars bewältigen zu können.
[MS/CB]
Originalveröffentlichung:
Tanja Woyke, Hanno Teeling, Natalia N. Ivanova, Marcel Hunteman, Michael Richter, Frank Oliver Gloeckner, Dario Boffelli, Iain J. Anderson, Kerrie W. Barry, Harris J. Shapiro, Ernest Szeto, Nikos C. Kyrpides, Marc Mussmann, Rudolf Amann, Claudia Bergin, Caroline Ruehland, Edward M. Rubin, Nicole Dubilier
Symbiosis insights through metagenomic analysis of a microbial consortium
Nature, Advanced Online Publication, September 17, 2006.
Media Contact
Weitere Informationen:
http://www.mpg.de/Alle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Merkmale des Untergrunds unter dem Thwaites-Gletscher enthüllt
Ein Forschungsteam hat felsige Berge und glattes Terrain unter dem Thwaites-Gletscher in der Westantarktis entdeckt – dem breiteste Gletscher der Erde, der halb so groß wie Deutschland und über 1000…

Wasserabweisende Fasern ohne PFAS
Endlich umweltfreundlich… Regenjacken, Badehosen oder Polsterstoffe: Textilien mit wasserabweisenden Eigenschaften benötigen eine chemische Imprägnierung. Fluor-haltige PFAS-Chemikalien sind zwar wirkungsvoll, schaden aber der Gesundheit und reichern sich in der Umwelt an….

Das massereichste stellare schwarze Loch unserer Galaxie entdeckt
Astronominnen und Astronomen haben das massereichste stellare schwarze Loch identifiziert, das bisher in der Milchstraßengalaxie entdeckt wurde. Entdeckt wurde das schwarze Loch in den Daten der Gaia-Mission der Europäischen Weltraumorganisation,…