Max-Planck-Wissenschaftler entdecken ungewöhnliche Dynamik von Aktivitätsmustern innerhalb skalenfreier Netzwerke

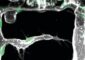
Drei aufeinander folgende Schnappschüsse von Aktivitätsmustern innerhalb eines relativ kleinen skalenfreien Netzwerks mit 31 Knoten und 50 Verbindungen. Die aktiven und inaktiven Knoten werden weiß bzw. schwarz dargestellt. Bei dem anfänglichen Muster auf der linken Seite sind die Hälfte der Knoten inaktiv (schwarz); bei dem abschließenden Muster auf der rechten Seite sind nahezu alle Knoten aktiv (weiß). Jeder Knoten des Netzwerks besitzt einen gewissen Verbindungsgrad, der der Anzahl der Verbindungen entspricht, die den Knoten angefügt sind. Diese Zahl wird für einige Knoten auf der linken Seite genau angegeben. Der Knoten mit dem höchsten Verbindungsgrad befindet sich in der Mitte des Netzwerks und besitzt 21 Verbindungen. Bild: Max-Planck-Institut für Kolloid- und Grenzflächenforschung
Unsere Biosphäre enthält verschiedene skalenfreie Netzwerke. Besonders interessant sind die funktionalen Netzwerke innerhalb des menschlichen Gehirns. Wissenschaftler am Max-Planck-Institut für Kolloid- und Grenzflächenforschung haben entdeckt, dass die Aktivitätsmuster derartiger Netzwerke ungewöhnliche dynamische Eigenschaften besitzen. Diese werden von nur wenigen Knoten mit einer hohen Anzahl von Verbindungen bestimmt. Geordnete Aktivitätsmuster sind deshalb sehr robust gegenüber zufälligen Störungen, reagieren jedoch sehr empfindlich, wenn die stark vernetzten Knoten selektiv gestört werden. Ungeordnete Muster sind andererseits immer instabil und wandeln sich sehr schnell in geordnete Muster um, und zwar selbst dann, wenn das Netzwerk unendlich groß ist. Die Netzwerke können zudem eine große Anzahl von vorgegebenen Mustern speichern und wieder abrufen (PNAS, Advanced Online Publication, 8. Juli 2005).
Das menschliche Gehirn besteht aus ca. 100 Milliarden Nervenzellen oder Neuronen, die so miteinander verbunden sind, dass sie ein riesiges Netzwerk bilden. Jedes einzelne Neuron ist in der Lage selbst aktiv zu werden, indem es ein Aktionspotential erzeugt. Wäre es möglich, einen Schnappschuss vom gesamten neuralen Netzwerk zu erstellen, würde zu jeder Zeit ein spezielles Muster von aktiven und inaktiven Neuronen erscheinen. Vereint man die Schnappschüsse zu einem Film, erhält man Aktivitätsmuster, die sich kontinuierlich mit der Zeit ändern (s. Abb. 1). Die Entwicklung dieser Muster repräsentiert die globale Dynamik des neuralen Netzwerks. Bislang ist es noch nicht möglich, Aktivitätsmuster auf der Ebene der einzelnen Neuronen zu beobachten. Moderne bildgebende Verfahren bieten jedoch die Möglichkeit, derartige Muster mit einer reduzierten räumlichen Auflösung darzustellen. Benutzt man zum Beispiel die Magnet-Resonanz-Tomographie, so kann man die Aktivität von etwa 100.000 Neuronencluster auflösen, die jeweils aus rund einer Million Neuronen bestehen. Auf diese Weise erhält man dynamische Aktivitätsmuster auf einem neuen, funktionalen Netzwerk, dessen Knoten durch die Neuronencluster gebildet werden.
In jedem Netzwerk können die Knoten durch ihren Verbindungsgrad charakterisiert werden, d.h. durch die Anzahl der Verbindungen zu anderen Knoten (vgl. Abb.1). Dieser Verbindungsgrad variiert von Knoten zu Knoten. Wenn man die Anzahl der Knoten innerhalb des Netzwerks zählt, die einen bestimmten Verbindungsgrad besitzen, erhält man ein Histogramm oder die genaue Verteilung des Verbindungsgrades im Netzwerk. Diese Verteilung bestimmt die Wahrscheinlichkeit P(k), dass ein zufällig gewählter Knoten des Netzwerkes k Verbindungen besitzt. Mittels Magnet-Resonanz-Tomographie wurde kürzlich ermittelt, dass die funktionalen Netzwerke im menschlichen Gehirn eine skalenfreie Wahrscheinlichkeits-Verteilung P(k) besitzen.
Um die Bezeichnung „skalenfrei“ zu verstehen, ist es aufschlussreich, die Wahrscheinlichkeiten P(10), P(100), P(1000), P(10.000) usw. zu betrachten, die angeben, dass ein zufällig herausgegriffener Knoten 10, 100, 1000, 10.000 usw. Verbindungen besitzt. Ein skalenfreies Netzwerk hat die Eigenschaft, dass das Verhältnis P(100)/P(10) genauso groß ist wie das Verhältnis P(1000)/P(100), das wiederum genauso groß ist wie das Verhältnis P(10.000)/P(1000) usw. Der Logarithmus dieses konstanten Verhältnisses definiert den so genannten Zerfallsexponenten. Für die funktionalen Netzwerke des menschlichen Gehirns, hat der Zerfallsexponent einen Wert von etwa 2.1.
Wissenschaftler des Max-Planck-Instituts für Kolloid- und Grenzflächenforschung haben jetzt ungewöhnliche dynamische Eigenschaften von Aktivitätsmustern auf derartigen skalenfreien Netzwerken entdeckt. Geordnete Aktivitätsmuster sind einerseits sehr robust gegenüber Störungen von zufällig ausgewählten Knoten, reagieren jedoch sehr empfindlich auf selektive Störungen der wenigen Knoten, die stark vernetzt sind. Mit einer derartigen selektiven Störung kann man ein bestimmtes geordnetes Aktivitätsmuster leicht in ein anderes geordnetes Muster umwandeln. Tatsächlich reagiert das skalenfreie Netzwerk auf solche selektive Störungen umso empfindlicher, je mehr sich der Zerfallsexponent dem Grenzwert 2 nähert. Ungeordnete Muster sind dagegen immer instabil und wandeln sich in relativ kurzer Zeit in geordnete Muster um. Diese Eigenschaft gilt sogar für unendliche Netzwerke, für die man eigentlich eine unendliche Zerfallszeit erwarten würde. Die relativ wenigen, stark vernetzten Knoten sind offenbar in der Lage, die relevanten Informationen über das gesamte Netzwerk zu streuen. Skalenfreie Netzwerke führen demnach zu geordneten Aktivitätsmustern, die sich trotz ihrer Stabilität gegenüber zufälligen Störungen relativ schnell ineinander umwandeln oder „umschalten“ lassen.
Derartige Netzwerke können auch benutzt werden, um eine bestimmte Anzahl von vorgegebenen Aktivitätsmustern zu speichern und wieder abzurufen. Die neue PNAS-Studie zeigt, dass skalenfreie Netzwerke mit einem Zerfallsexponenten, der größer ist als zwei, aber kleiner als 2,5, eine sehr große Anzahl von Mustern speichern können. Diese Anzahl erhöht sich kontinuierlich mit der Netzwerkgröße. Nähert sich der Zerfallsexponent dem Grenzwert 2, so wächst die Speicherkapazität des Netzwerks sogar proportional mit der Netzwerkgröße an. Übersteigt der Zerfallsexponent jedoch den Grenzwert von 2,5 bleibt die Zahl der gespeicherten Muster selbst für ein unendlich großes Netz klein. Skalenfreie biomimetische Netzwerke mit einem Exponenten größer 2 und kleiner 2,5 könnten so mögliche Designs für assoziative Speicher und Mustererkennung zur Verfügung stellen.
Die beschriebenen dynamischen Eigenschaften beschränken sich aber nicht auf neurale Netzwerke. In der Tat findet man in der Biosphäre eine ganze Hierarchie von skalenfreien Netzwerken, die typischerweise einen Zerfallsexponenten in diesem Bereich aufweisen. Diese Eigenschaft gilt zum Beispiel auch für metabolische Netzwerke innerhalb biologischer Zellen, wie bereits für 43 verschiedene Organismen aus allen drei Reichen des Lebens (Eukaryoten, Prokaryoten und Archäen) gezeigt wurde.
Auf der makroskopischen Ebene, die unserer Wahrnehmung direkt zugänglich ist, findet man Skalenfreiheit bei vielen sozialen Netzwerken, die auf unterschiedlichen Tätigkeiten beruhen wie der Zusammenarbeit von Schauspielern, der Co-Autorschaft von Wissenschaftlern oder häufiger Telefongespräche. Schließlich kann man die gleiche skalenfreie Struktur auch für die Netzwerke von Internet-Routern und Links auf dem World Wide Web finden. Alle diese Netze haben die Eigenschaft, dass ihre Aktivitätsmuster von relativ wenigen Knoten gesteuert werden, die eine hohe Anzahl von Verbindungen aufweisen.
Originalveröffentlichung:
Haijun Zhou and Reinhard Lipowsky
Dynamic pattern evolution on scale-free networks
PNAS, Advanced Online Publication, 8 July 2005
Media Contact
Weitere Informationen:
http://www.mpg.deAlle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Ideen für die Zukunft
TU Berlin präsentiert sich vom 22. bis 26. April 2024 mit neun Projekten auf der Hannover Messe 2024. Die HANNOVER MESSE gilt als die Weltleitmesse der Industrie. Ihr diesjähriger Schwerpunkt…

Peptide auf interstellarem Eis
Dass einfache Peptide auf kosmischen Staubkörnern entstehen können, wurde vom Forschungsteam um Dr. Serge Krasnokutski vom Astrophysikalischen Labor des Max-Planck-Instituts für Astronomie an der Universität Jena bereits gezeigt. Bisher ging…

Wasserstoff-Produktion in der heimischen Garage
Forschungsteam der Frankfurt UAS entwickelt Prototyp für Privathaushalte: Förderzusage vom Land Hessen für 2. Projektphase. Wasserstoff als Energieträger der Zukunft ist nicht frei verfügbar, sondern muss aufwendig hergestellt werden. Das…