Kein Stau am Golgi-Apparat
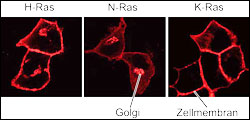
Lokalisation von drei verschiedenen Fluoreszenz-markierten Formen des Ras-Proteins. K-Ras ist nur an der Zellmembran und N-Ras hauptsächlich am Golgi-Apparat zu finden, während H-Ras etwa gleichmäßig zwischen Zellmembran und Golgi-Apparat verteilt ist. Bild: Max-Planck-Institut für molekulare Physiologie
Biochemiker des Max-Planck-Instituts für molekulare Physiologie in Dortmund zeichnen erstmals die Wanderwege von Ras-Proteinen in lebenden Zellen nach
Ras-Proteine, molekulare Schalter, die bei der Zellteilung und Tumorentstehung beim Menschen eine wichtige Rolle spielen, werden in menschlichen Zellen durch schnelle Transportprozesse zwischen dem Golgi-Apparat und der Zellmembran hin und her befördert. Verantwortlich hierfür ist die reversible Modifizierung dieser Proteine durch lipophile (fettliebende) Gruppen. Das haben jetzt Wissenschaftler des Max-Planck-Instituts für molekulare Physiologie in Dortmund unter der Leitung von Prof. Alfred Wittinghofer in Zusammenarbeit mit einer Arbeitsgruppe um Dr. Philippe Bastiaens vom European Molecular Biology Laboratory (EMBL) in Heidelberg nachgewiesen. Bisher ging man davon aus, dass Ras-Proteine nur in einer Richtung vom Golgi-Apparat zur äußeren Zellmembran transportiert werden und dort verbleiben. Für ihre Entdeckung haben die Zellbiologen ein neues Prinzip zur Lokalisierung von Proteinen in verschiedenen Membranen der Zelle entwickelt. Die Forscher brachten in einem interdisziplinären Ansatz chemisch-biochemisch-biophysikalische-zellbiologische Methoden und Erfahrungen zusammen, um der lang diskutierten Frage der Lokalisation von Ras-Proteinen in Zellen auf die Spur zu kommen (Science Express, 10. Februar 2005).
Zunächst ging es darum, zu klären, wie das Protein Ras gleichzeitig in zwei verschiedenen Substrukturen der Zelle, dem Golgi-Apparat und der äußeren Hülle der Zelle, der Plasmamembran, lokalisiert sein kann. Dazu wollten die Biologen wissen, warum verschiedene Formen des Ras-Proteins in unterschiedlichen Mengen in den jeweiligen Membransystemen vorkommen. Im nicht aktivierten Zustand liegen Ras-Proteine in der „Aus“-Form vor und gehen durch ein von außerhalb der Zelle kommendes Wachstumssignal in den „An“-Zustand über. Es gibt drei verschiedenen Formen von Ras-Proteinen, die man nach ihrem Vorkommen in bestimmten tierischen oder menschlichen Tumoren H-, N- oder K-Ras bezeichnet. Ras-Proteine sind mit der Zellmembran verankert, weil sie nach der Synthese im Zytoplasma der Zelle über mehrere Zwischenstufen mit ein, zwei oder drei lipophilen (fettliebenden) Gruppen verknüpft werden und sie dadurch eine hohe Affinität für die ebenfalls lipophilen Membranen erhalten.
Gängige Lehrmeinung war, dass Ras-Proteine nach der Synthese und nach Anknüpfung der lipophilen Gruppe im Golgi-Apparat zu finden sind, weil dieses Organell beim Transport der Ras-Proteine vom endoplasmatischen Retikulum zur Zellmembran als Zwischenstation fungiert. Danach sind die im Golgi-Apparat befindlichen Ras-Proteine dort sozusagen wegen eines Verkehrsstaus lokalisiert.
Die Mitarbeiter von Bastiaens und Wittinghofer haben nun mit Hilfe von ausgefeilten mikroskopischen Techniken und unter Verwendung von fluoreszent markierten Ras-Proteinen ihre Wanderwege in lebenden Zellen untersucht. Das Team konnte zeigen, dass das bisherige Modell so nicht richtig sein kann. Dazu verhinderten die Forscher die Neusynthese des Proteins und zerstörten gleichzeitig das im Golgi-Apparat befindliche Ras-Protein durch gezielte Verwendung eines Laserstrahls. Obwohl kein neu hergestelltes Ras-Protein mehr nachgeliefert wurde, kam es erstaunlicherweise nach kurzer Zeit zu einer Wieder-Anreicherung des Proteins am Golgi. Mit Hilfe molekularer Sonden entdeckten die Biologen, dass es einen kontinuierlichen Vor- und Zurück-Transport zwischen Golgi-Apparat und Zellmembran gibt, also genau das Gegenteil von einem Verkehrsstau.
Die Biochemiker vermuteten nun, dass diese schnellen Transportprozesse zwischen den Membransystemen dadurch zustande kommen, dass die Ras-Proteine am Golgi-Apparat mit einer lipophilen Gruppe versehen und nach erfolgter Modifizierung an die Plasma-Membran transportiert werden. Nach einer bestimmten Zeit wird diese lipophile Gruppe wieder abgespalten. Dadurch können die Proteine zurückgebracht und am Golgi-Apparat neu mit der lipophilen Gruppe verknüpft werden. Je nachdem, wie lange die Anheftung oder die Abspaltung dauern, hält sich das Protein mehr oder weniger lange in dem einen oder anderen Teil der Zelle auf.
Der endgültige Beweis für diese zunächst gewagte Hypothese konnte dann durch Zusammenarbeit mit Prof. Herbert Waldmann und Dr. Jürgen Kuhlmann vom Max-Planck-Institut für molekulare Physiologie erbracht werden. Dabei wurde durch chemische Synthese eine lipophile Gruppe an das Protein gebracht, die zwar genauso aussah wie die natürliche, aber nicht mehr abgespalten werden konnte. Dieses Variante zeigte dann in der Zelle keinen geregelten Vor- und Rücktransport mehr zwischen den Membranen.
Was bedeuten diese Befunde nun für die Funktion und Lokalisation von Membranproteinen? Die Wissenschaftler konnten zeigen, dass ihr neu entdecktes Prinzip der Modifizierung durch lipophile Gruppen für alle Proteine gilt, die eine abspaltbare lipophile Gruppe tragen. Dies erlaubt der Zelle über die Ausstattung mit lipophilen Ankern die Proteine an unterschiedliche Membransysteme zu adressieren. Je nach Geschwindigkeiten der Modifizierung erfolgt dann ein geregelter Austausch zwischen diesen Membransystemen.
Für die Signalweiterleitung mit Hilfe des Ras-Proteins bedeutet das zunächst, dass die verschiedenen Formen von Ras, die auch in verschiedener Weise an der Tumorentstehung beim Menschen beteiligt sind, sich über ihre spezifische Modifikation in ihren zugehörigen Membranstrukturen aufhalten. „Unsere Befunde erklären wahrscheinlich auch, warum in der Zelle zunächst das in der Zellmembran lokalisierte Ras-Protein durch das von außen kommende Wachstumssignal aktiviert wird und dann das aktivierte Ras-Protein in die Golgi-Membran transportiert werden kann“, sagt Alfred Wittinghofer. „Die unterschiedliche Lokalisation bewirkt dann, dass dort Ras-Proteine andere biologische Wirkungen hervorrufen als Ras-Proteine in der äußeren Membran der Zelle.“
Mit ihren Untersuchungen ist den Arbeitsgruppen damit ein wichtiger Schritt gelungen, das komplexe Muster der Signalweiterleitung von und die Unterschiede zwischen den Ras-Proteinen in lebenden Zellen zu erklären Die Studie wird auch dazu beitragen zu verstehen, warum unterschiedliche Ras-Proteine beim Menschen unterschiedliche Tumore hervorrufen.
Originalveröffentlichung:
O. Rocks, M. Kahms, C. Koerner, M. Lumbierres, J. Kuhlmann, H. Waldman and A. Wittinghofer
A De-/Reacylation Cycle Regulates Localisation and Activity of Palmitoylated Ras isoforms
Science Express, 10 February 2005
Media Contact
Weitere Informationen:
http://www.mpg.deAlle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Erstmals 6G-Mobilfunk in Alpen getestet
Forschende der Universität Stuttgart erzielen leistungsstärkste Verbindung. Notrufe selbst in entlegenen Gegenden absetzen und dabei hohe Datenmengen in Echtzeit übertragen? Das soll möglich werden mit der sechsten Mobilfunkgeneration – kurz…

Neues Sensornetzwerk registriert ungewöhnliches Schwarmbeben im Vogtland
Das soeben fertig installierte Überwachungsnetz aus seismischen Sensoren in Bohrlöchern zeichnete Tausende Erdbebensignale auf – ein einzigartiger Datensatz zur Erforschung der Ursache von Schwarmbeben. Seit dem 20. März registriert ein…

Bestandsmanagement optimieren
Crateflow ermöglicht präzise KI-basierte Nachfrageprognosen. Eine zentrale Herausforderung für Unternehmen liegt darin, Über- und Unterbestände zu kontrollieren und Lieferketten störungsresistent zu gestalten. Dabei helfen Nachfrage-Prognosen, die Faktoren wie Lagerbestände, Bestellmengen,…