Software-Neuheit für die mikrobiologische Genomforschung
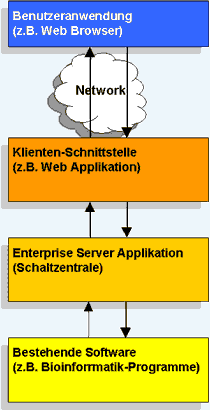
Die Bausteine der MiGenAS Workflow Engine (Microbial Genome Analysis System). Die Klientenschnittstelle kommuniziert mit der Benutzeranwendung, während die mittlere Schicht ("Enterprise Server Applikation") die Schaltzentrale darstellt. Die rechte Schicht stellt die Verbindung zu den bestehenden Software-Komponenten her. <br>Bild: Rechenzentrum Garching der Max-Planck-Gesellschaft
Billing-Preis für wissenschaftliches Rechnen 2004 geht an Entwickler einer innovativen Software-Umgebung für Bioinformatik-Problemstellungen
Für die Entwicklung einer integrierten Software-Umgebung für die Bioinformatik erhalten Markus Rampp und Thomas Soddemann vom Rechenzentrum Garching der Max-Planck-Gesellschaft den diesjährigen, mit 3.000 Euro dotierten Heinz-Billing-Preis. Die im Rahmen des Projekts MiGenAS (Microbial Genome Analysis System) der Max-Planck-Gesellschaft programmierte Workflow Engine macht es Wissenschaftlern wesentlich leichter, verschiedenste bioinformatische Software-Werkzeuge für die Analyse von Gen- und Proteinsequenzen anzuwenden und miteinander zu verknüpfen. Entstanden ist diese Innovation in enger Zusammenarbeit zwischen Biowissenschaftlern und Software-Ingenieuren. In die Endrunde der Billing-Preis-Verleihung kamen auch Johannes Wicht und Julien Aubert vom Max-Planck-Institut für Sonnensystemforschung in Katlenburg-Lindau mit ihrem Projekt „Dynamos in Action“ sowie Niko Beerenwinkel vom Max-Planck-Institut für Informatik in Saarbrücken mit dem Programmpaket „Computerwerkzeuge für die Analyse und Simulation der HIV-Resistenz gegen Wirkstoffe“. Die Preisverleihung erfolgt am 18. November 2004 in Göttingen.
Neben dem kürzlich „entschlüsselten“ menschlichen Genom ist die Erbinformation von inzwischen mehr als einhundert Organismen bekannt. Die meisten davon stammen aus dem Reich der Mikroorganismen. Das Identifizieren, Kategorisieren und Analysieren von Genen und ihren Produkten (Proteine) erfolgt dabei nicht nur in biochemischen Labors, sondern in zunehmendem Maße auch mit dem Computer („in silico“). Bioinformatiker entwickeln dafür Algorithmen und liefern geeignete Software-Werkzeuge, damit man die anfallende Datenflut effizient analysieren kann. Allerdings sind viele der eingesetzten Programme zueinander nicht kompatibel. Forscher sind vielmehr häufig gezwungen, ihre Daten zwischen vielen verschiedenen Formaten hin und her zu konvertieren. Dies ist erfahrungsgemäß sowohl mühsam als auch fehleranfällig.
Unter der Federführung des MiGenAS-Konsortiums (Microbial Genome Analysis System), das sich aus Arbeitsgruppen der Max-Planck-Institute für Biochemie (Abt. Oesterhelt), Entwicklungsbiologie (Abt. Lupas und AG Schuster), Informatik (Abt. Lengauer) und marine Mikrobiologie (Abt. Amann) sowie Partnern von den Universitäten Bielefeld und Tübingen zusammensetzt, wurde nun am Rechenzentrum Garching der MPG (RZG) eine in dieser Form einzigartige Softwarelösung entwickelt. Diese nimmt dem Forscher nicht nur alle Aufgaben bei der Umwandlung von Datenformaten für die eingebetteten Programme und Datenbanken ab, sondern erlaubt es auch, ganze Arbeitsabläufe, die aus der Verkettung einer Vielzahl von Programmen entstehen, in einer vereinheitlichten Software-Umgebung zu formulieren und durchzuführen, letzteres auch automatisch. Die Palette der eingebundenen Werkzeuge reicht von Programmen zur Suche in Sequenzdatenbanken über die Erstellung von Stammbäumen bis zur Vorhersage von Struktur und Funktion von Proteinen.
Die Architektur der Softwarelösung besteht aus der Benutzeranwendung und drei serverseitigen Schichten (vgl. Abb.). Während der Anwender direkt über einen Webbrowser mit der Web-Applikation kommuniziert, die ihm zum Beispiel Formulare zur Definition der Aufgabenstellung und Internetseiten für die Ergebnispräsentation zur Verfügung stellt, ist die so genannte „Enterprise Server Applikation“ die eigentliche Schaltzentrale des Systems. Hier werden Anwendereingaben interpretiert, Daten verwaltet, Dateiformate konvertiert und rechenintensive Aufgaben an andere Computer – unter Benutzung der dritten Schicht – delegiert. Hier ist auch die „Workflow Engine“ angesiedelt, welche die Arbeitsabläufe koordiniert und kontrolliert. Die dritte Schicht stellt unter anderem die Verbindung zur Farm der Auswertungsrechner her, wo alle rechenintensiven Aufgaben effizient abgearbeitet werden können. Diese Schicht enthält auch die Schnittstelle zu den verschiedenen, bereits existierenden bioinformatischen Software-Werkzeugen, die ihrerseits meist frei verfügbar sind oder von den Wissenschaftlern des MiGenAS-Konsortiums entwickelt und bereitgestellt werden.
Eine weitere wichtige Komponente im Gesamtkonzept sind die so genannten Web Service-Schnittstellen, die es dem Benutzer ermöglichen, einzelne Komponenten der Applikation flexibel in selbst entwickelte Programme einzubinden. Der Einsatz der gerade erst aufblühenden Web Service-Technologie eröffnet vielfältige Möglichkeiten für den Einsatz der MiGenAS Workflow Engine. So könnte das System beispielsweise Teil einer Grid-Anwendung werden, also einer Anwendung, die auf im Internet verteilten Diensten und Rechnerressourcen basiert. Bestehende Dienste können ihrerseits aber auch direkt in einen Arbeitsablauf mit eingebunden werden.
Bei Architektur und Design des Systems wurde darauf geachtet, dass es offen und dadurch einfach erweiterbar ist. Somit kann die Palette der zur Verfügung gestellten Werkzeuge mit der Zeit problemlos ergänzt werden. Daher stellt diese Software-Umgebung über die zuvor geschilderte konkrete Problemstellung hinaus eine allgemeine Plattform für zukünftige Anwendungen dar.
Die beiden anderen Finalisten für den Heinz-Billing-Preis 2004 kommen in diesem Jahr aus der Physik und der Bioinformatik
Der zweite Preis wird an Johannes Wicht und Julien Aubert, Max-Planck-Institut für Sonnensystemforschung, für das Projekt „Dynamos in Action“ verliehen. Die Methoden zur Simulation planetarer Magnetfelder werden immer komplexer und verlangen nach entsprechend weiter entwickelten Analyseverfahren. Mit dem neuen Tool wurde ein graphisches Analyseverfahren entwickelt, das die zeitabhängige Entwicklung von 3D-Vektorfeldern visualisiert und es ermöglicht, ihre Abhängigkeit von einzelnen Parametern zu identifizieren. Es leistet damit einen wesentlichen Beitrag zu einem besseren Verständnis und einer vereinfachten Analyse der Vorgänge in planetaren Magnetfeldern.
Niko Beerenwinkel, Max-Planck-Institut für Informatik, wird für das Programmpaket „Computational Tools for the Analysis and Simulation of HIV Drug Resistance“ mit dem dritten Preis ausgezeichnet. Ein zentrales Problem bei der Behandlung HIV-infizierter Personen ist die Wirkstoffresistenz der Viren. Das Programmpaket gibt Forschern jetzt Analyse-Werkzeuge an die Hand, die eine Suche nach geeigneten Wirkstoffkombinationen bei einer Resistenz wesentlich unterstützen. Damit ist man dem Ziel einer Entscheidungshilfe für die Medizin im Sinne einer „Personalized Medicine“ – im Gegensatz zum bisherigen universellen Ansatz – ein Stück näher gekommen.
Heinz-Billing-Preis zur Förderung des wissenschaftlichen Rechnens
Der „Heinz-Billing-Preis zur Förderung des wissenschaftlichen Rechnens“ wurde im Jahr 1993 zum ersten Mal verliehen. Mit dieser Ehrung werden herausragende Leistungen von Wissenschaftlern und Computerspezialisten gewürdigt, die in zeitintensiver und sehr kreativer Arbeit die notwendigen Hard- und Software-Voraussetzungen entwickeln, die für neue Vorstöße in der Forschung heute unverzichtbar sind. Der mit 3.000 Euro dotierte Preis ist benannt nach Prof. Heinz Billing, Emeritiertes Wissenschaftliches Mitglied des Max-Planck-Instituts für Astrophysik und langjähriger Vorsitzender des Beratenden Ausschusses für Rechenanlagen in der Max-Planck-Gesellschaft. Billing stand mit der Erfindung des Trommelspeichers und dem Bau der Rechner G1, G2 und G3 als Pionier der elektronischen Datenverarbeitung am Beginn des wissenschaftlichen Rechnens. Die für den Preis eingereichten Arbeiten werden jedes Jahr in der Buchreihe „Forschung und wissenschaftliches Rechnen“ der Gesellschaft für wissenschaftliche Datenverarbeitung mbH in Göttingen veröffentlicht.
Weitere Informationen erhalten Sie von:
Thomas Soddemann
Rechenzentrum Garching der Max-Planck-Gesellschaft, Garching
Tel.: 089 3299-2694
E-Mail: Thomas.Soddemann@rzg.mpg.de
Markus Rampp
Rechenzentrum Garching der Max-Planck-Gesellschaft, Garching
Tel.: 089 3299-1324
E-Mail: Markus.Rampp@rzg.mpg.de
Media Contact
Weitere Informationen:
http://www.billingpreis.mpg.de/Alle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Neue Industrie-4.0-Lösung für niedrigschwelligen Zugang zu Datenräumen
»Energizing a Sustainable Industry« – das Motto der Hannover Messe 2024 zeigt klar, wie wichtig eine gleichermaßen leistungsstarke und nachhaltige Industrie für den Fertigungsstandort Deutschland ist. Auf der Weltleitmesse der…

Quantenpräzision: Eine neue Art von Widerstand
Physikforschende der Universität Würzburg haben eine Methode entwickelt, die die Leistung von Quantenwiderstands-Normalen verbessern kann. Sie basiert auf einem Quantenphänomen namens anomaler Quanten-Hall-Effekt. In der industriellen Produktion oder in der…

Sicherheitslücke in Browser-Schnittstelle erlaubt Rechnerzugriff über Grafikkarte
Forschende der TU Graz waren über die Browser-Schnittstelle WebGPU mit drei verschiedenen Seitenkanal-Angriffen auf Grafikkarten erfolgreich. Die Angriffe gingen schnell genug, um bei normalem Surfverhalten zu gelingen. Moderne Websites stellen…