Unverzichtbare Rolle der dritten tRNA-Bindungsstelle des Ribosoms für fehlerfreie Übersetzung der Erbinformation nachgewiesen

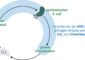
Kryo-Elektronenmikroskopische Rekonstruktion von tRNAs im Pre- und Post-Stadium, die an ein 70S-Ribosom (gelb: kleine Untereinheit, blau: große Untereinheit) gebunden sind (mRNA nicht mit dargestellt). PRE: tRNAs sind an die A- (pink) und P- (grün) Bindungsstellen des Ribosoms gebunden. Bei der Translokation wandern sie vom der A- in die Pund von der P- in die E-Stelle. Dabei ziehen sie die mRNA mit sich mit. POST: Nach der Translokation sind die tRNAs an die P- (grün) und E- (gelb) Bindungsstelle gebunden. Damit ist die A-Bindungsstelle frei und kann erneut eine tRNA binden. Abkürzungen: CP - central protuberance; ST - L7/L12 stalk base; lb - long bridge (helix 38 of 23 S rRNA); h - head; ch - mRNA channel; sh - shoulder region ; sp - spur. (Abbildung modifiziert nach Agrawal et al., J Cell Biol 2000)
Max-Planck-Forscher klären universellen Mechanismus für die Übersetzung der genetischen Information auf
Die Bildung von Proteinen am Ribosom als Ergebnis der exakten Übersetzung genetischer Information in eine definierte Kette von Aminosäuren ist eine der großartigsten Leistungen des Organismus. Ohne das strikte Einhalten des Lese-/Übersetzungsrasters von jeweils drei Basen in eine dadurch definierte Aminosäure ist die genetische Information jedoch wertlos. Wissenschaftler am Max-Planck-Institut für molekulare Genetik in Berlin konnten jetzt zeigen, dass die dritte tRNA-Bindungsstelle am Ribosom die Einhaltung des Leserasters der mRNA bei der Proteinbiosynthese kontrolliert. In der Fachzeitschrift Cell beschreiben sie die Funktion dieser dritten, bislang in ihrer Rolle umstrittenen tRNA-Bindungsstelle, deren Vorhandensein sie bereits 1980 nachgewiesen hatten [Cell 118: 45-55, 2004].
Die genetische Information aller Organismen ist als lineare Abfolge der vier Bausteine A, T (U bei RNA), G und C in den Nukleinsäuren DNA und RNA gespeichert. Damit die Information jedoch ihre Wirkung im Organismus entfalten kann, muss sie zunächst in Proteine, die Arbeits- und Funktionsstoffe der Zelle, übersetzt werden. Auch Proteine sind kettenförmige Moleküle aus einzelnen Bausteinen, in diesem Fall aus Aminosäuren. Zwanzig verschiedene existieren im Organismus, die für jedes Protein in festgelegter Reihenfolge aneinander gehängt werden müssen. Störungen bzw. Fehler in der Reihenfolge können zum Funktionsverlust des betroffenen Proteins führen. Die Anleitung für die richtige Reihenfolge verbirgt sich in der Abfolge der einzelnen Bausteine der DNA. Jeweils drei aufeinander folgende DNA-Bausteine (ein „Codon“) kodieren eine bestimmte Aminosäure.
Für die Übersetzung der DNA wird die enthaltene Information in Form der Boten-RNA (messengerRNA/mRNA) von der DNA zu den Ribosomen, den „Produktionsstätten“ der Proteine, transportiert. L-förmige Moleküle mit einem spezifischen Basen-Triplett („Anticodon“) an einem und einer dazu gehörigen Aminosäure am anderen Ende, die so genannten transfer-RNAs (tRNAs), „lesen“ die an das Ribosom gebundene mRNA über präzises Einpassen der jeweiligen Codon-Anticodon-Paare ab. Durch Verknüpfung der Aminosäuren der „lesenden“ tRNAs entsteht ein neues, genau definiertes Protein.
Für das Ablesen der mRNA ist es von größter Bedeutung, dass das Ribosom das richtige Leseraster auf der Nukleinsäurekette erkennt und einhält. Genau wie der Satz „die-kuhkam- auf-die-alm “ zu dem sinnlosen “ die-kuhk-ama-ufd-iea-lm “ wird, wenn das Leseraster nur um eine Stelle verschoben wird, verliert auch die Information in der mRNA auf diese Art ihre Bedeutung. Bereits seit langem ist bekannt, dass zwei funktionelle tRNABindestellen (A + P) am Ribosom für die Bindung der tRNA und das Ablesen der mRNA verantwortlich sind. Nierhaus und seine Mitarbeiter wiesen nun die Funktion einer dritten, ebenfalls universell vorhandenen Bindungsstelle (E-Stelle) nach, welche sie bereits 1980 entdeckt hatten. Sie konnten zeigen, dass die mRNA für das feste Einhalten des Leserasters jeweils über die Anticodone zweier benachbarter tRNAs auf dem Ribosom verbunden sein muss. Werden die tRNAs um eine Codonlänge im Ribosom verschoben (Translokation), wandern sie vom der A- in die P- und von der P- in die E-Stelle (siehe Abbildung 1) und ziehen die mRNA mit sich mit. Die Codon-Anticodon-Brücken zwischen tRNAs und mRNAs müssen dabei erhalten bleiben, um ein Verrutschen der mRNA und damit eine Verschiebung des Leserasters zu vermeiden. Die Bindung einer neuen tRNA an die nach der Translokation wieder freie A-Stelle erfolgt zeitgleich mit der Ablösung von der E-Bindungsstelle, d.h., während der Proteinsynthese befinden sich mindestens immer zwei tRNAs auf dem Ribosom. Wird die tRNA aus der E-Stelle jedoch vorzeitig aus dem Ribosom entfernt, so dass die mRNA nur noch über ein Anticodon gebunden ist, kommt es häufig zum „frame shift“, d.h., dem Weiterrücken der ablesenden tRNAs um eine Position auf der mRNA (siehe Abbildung 2). Dies führt zum Verlust des Leserasters. Wird dagegen die mRNA über zwei Anticodone verankert, verliert das Ribosom weniger als einmal bei 30.000 Übersetzungsschritten spontan den Leserahmen.
Die Aufklärung dieses Mechanismus zur Erhaltung des Leserahmens gelang den Berliner Forschern über einen Umweg. Bereits seit längerem ist bekannt, dass beispielsweise der bakterielle Proteinsynthese-Faktor RF2 für seine Bildung eine gezielte Verschiebung des Leserasters am 26. Codon benötigt. Nierhaus und seine Kollegen konnten zeigen, dass auch hier alle drei Bindungsstellen aktiv an der Übersetzung der mRNA beteiligt sind. Allerdings kommt es beim Einrücken des 26. Codons in die A-Stelle zu einer gezielten Verdrängung der tRNA an der E-Bindungsstelle (siehe Abbildung 2). Die Verdrängung erfolgt dabei mittels einer „Shine-Dalgarno“(SD)-Sequenz, die stark an einen definierten Teil des Ribosoms (Anti-SD) bindet.
Praktisch jede bakterielle mRNA enthält eine so genannte „Shine-Dalgarno“(SD)- Sequenz. Üblicherweise befindet sie sich vor dem Codon für die erste A-Bindungsstelle einer mRNA und sorgt dafür, dass die mRNA richtig innerhalb des Ribosoms positioniert wird. Die RF2 mRNA besitzt eine zweite SD-ähnliche Sequenz vor dem 26. Codon. Wenn diese an das Ribosom bindet, kommt es zu einer Überlappung mit der E-Bindungsstelle und die dort gebundene tRNA wird verdrängt. Dadurch erhält die mRNA Bewegungsfreiheit und die erforderliche Leserasterverschiebung kann stattfinden.
Fazit: Nach der Translokation müssen tRNAs in P und E Stelle die mRNA mittels Codon-Anticodon-Wechselwirkung verankern, um eine Leserasterverschiebung zu vermeiden. Der Regulationsmechanismus für die Synthese des RF2 Faktors nutzt die Bedeutung der tRNA in der E Stelle aus: Die SD Sequenz verdrängt die tRNA aus der E-Stelle und ermöglicht damit die hocheffiziente Leserasterverschiebung, die für die RF2 Synthese erforderlich ist.
Vollständiger Artikel: Viter Márquez, Daniel N. Wilson, Warren P. Tate, Francisco Triana-Alonso, Knud H. Nierhaus. Maintaining the Ribosomal Reading Frame: The Influence of the E site during Translational Regulation of Release Factor 2. Cell 2004 118: 45-55, July 9, 2004.
Weitere Informationen:
Prof. Dr. Knut Nierhaus
Max-Planck-Institut für molekulare Genetik
Tel.: 030-8413-1700, Fax: 030-8413-1690
AG Ribosomen Email:nierhaus@molgen.mpg.de
Ihnestrasse 63-73, 14195 Berlin
Media Contact
Weitere Informationen:
http://www.molgen.mpg.deAlle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Bakterien für klimaneutrale Chemikalien der Zukunft
Forschende an der ETH Zürich haben Bakterien im Labor so herangezüchtet, dass sie Methanol effizient verwerten können. Jetzt lässt sich der Stoffwechsel dieser Bakterien anzapfen, um wertvolle Produkte herzustellen, die…

Batterien: Heute die Materialien von morgen modellieren
Welche Faktoren bestimmen, wie schnell sich eine Batterie laden lässt? Dieser und weiteren Fragen gehen Forschende am Karlsruher Institut für Technologie (KIT) mit computergestützten Simulationen nach. Mikrostrukturmodelle tragen dazu bei,…

Porosität von Sedimentgestein mit Neutronen untersucht
Forschung am FRM II zu geologischen Lagerstätten. Dauerhafte unterirdische Lagerung von CO2 Poren so klein wie Bakterien Porenmessung mit Neutronen auf den Nanometer genau Ob Sedimentgesteine fossile Kohlenwasserstoffe speichern können…