Waffen von Killerzellen entschlüsselt
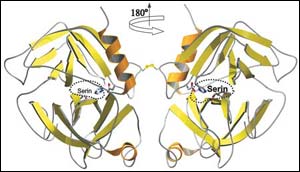
Abb.: Schematische Darstellung der dreidimensional gefalteten Aminosäuren(Polypeptid)kette des humanen Granzym A. Zwei Protease-Einheiten aus der gleichen Polypeptidkette sind in der Mitte fest miteinander verbunden, das katalytische Zentrum der linken Einheit zeigt nach hinten, das der rechten nach vorne zum Betrachter. Die beiden katalytischen Zentren um das Serin herum sind durch einen gestrichelten Kreis hervorgehoben. Die beiden Protease-Einheiten können durch eine 180 Grad-Drehung um die angegebene Vertikalachse miteinander überlagert werden. Vorder- und Rückseite verhalten sich funktionell identisch. <br> <br>Foto: Max-Planck-Institut für Neurobiologie <br>
Max-Planck-Forscher haben 3D-Struktur eines wichtigen Abwehrenzyms aufgedeckt und neues kostengünstiges Herstellungsverfahren entwickelt
Tumorzellen und viral infizierte Zellen bedrohen unser Leben Tag für Tag. Glücklicherweise verfügen wir in unserem Immunsystem über eine starke Leibwache von Killerzellen, die mit verschiedenen todbringenden Waffen – Proteasen oder Granzyme genannt – ausgerüstet sind und unerwünschte Zellen rechtzeitig eliminieren. Unter den circa 120 Proteasen des Menschen, die die Aminosäure Serin im ihrem Zentrum haben, verfügt das Granzym A als einzige über zwei identische katalytischen Köpfe. Wissenschaftler der Max-Planck-Institute für Biochemie und Neurobiologie in Martinsried bei München haben jetzt gemeinsam das Geheimnis dieses zweischneidigen Schwerts gelüftet und seine räumliche (dreidimensionale) Struktur bei hoher Auflösung analysiert (Nature Structural Biology 10, 535-540, Juli 2003). Diese Erkenntnisse sind wichtig für die Optimierung von Protease-Blockern, die bei Transplantationen, Autoimmunkrankheiten oder chronischen Infektionskrankheiten eingesetzt werden.
Die Granzyme A und B, zwei besonders intensiv erforschte Enzyme, sind hocheffiziente Katalysatoren des programmierten Zelltods und lösen eine explosionsartig ablaufende Kaskade von intrazellulären Prozessen aus, die zur Selbstauflösung und dem kompletten Abbau unerwünschter Körperzellen führen. Die selben lebensrettenden Proteasen können jedoch auch enorme destruktive Energien gegenüber Organtransplantaten, Spender-Stammzellen oder körpereigenen Geweben entfalten und verheerende Schäden an gesunden Zellen und Organen anrichten.
Granzym B erkennt besondere Sequenz-Motive an der Oberfläche von im Zytosol befindlichen Proenzymen, den Procaspasen, und wandelt diese Vorläufermoleküle durch selektiven Schnitt in der Nähe eines negativ geladenen Asparaginsäure-Restes in eine funktionell aktive Caspase um. Caspasen sind der intrazelluläre Motor eines universellen zellbiologischen Programms, das die Selbstzerstörung und geordnete Auflösung überflüssiger oder unerwünschter Zellen kontrolliert. Im Unterschied dazu erkennt Granzym A eine große Zahl von unterschiedlichen Oberflächen-Strukturen im Umgebungsbereich eines positiv geladenen Aminosäure-Restes und spaltet überwiegend Untereinheiten von großen Proteinkomplexen.
Die Max-Planck-Forscher Clara Hink-Schauer, Eva Estebanez-Perpina, Florian Kurschus, Wolfram Bode and Dieter E. Jenne haben nun mit Hilfe der Röntgenstrukturanalyse herausgefunden, dass die beiden miteinander verbundenen Molekülköpfe (-Einheiten) des Granzyms A in exakt entgegengesetzte Richtung zeigen und durch eine 180 Grad-Drehung ineinander überführt werden können (siehe Abbildung). Dadurch entstehen zwei funktionell gleichwertige Dimer-Oberflächen (Vorder- und Rückseite). Jede Moleküleinheit erfüllt eine doppelte Aufgabe: Mit der Rückseite des einen Moleküls wird die Beute festgehalten und dann dem Nachbarmolekül präsentiert. Dieses schneidet die Beute und gibt die beiden Bruchstücke rasch wieder frei. Wie ein zweischneidiges Schwert bahnt sich auf diese Weise das Granzym A seinen Weg durch komplexe Strukturen.
Zugleich ist es den Max-Planck-Forscher gelungen, ein neues, wesentlich kostengünstigeres Verfahren zur Herstellung von Granzym A zu entwickeln. Granzyme eigenen sich als Angriffspunkte für die Entwicklung neuartiger Medikamente, um eine Überreaktion des Immunsystems und die hierbei beobachteten Schäden durch Killerzellen zu verhindern. Das neue Herstellungsverfahren und die genaue Erfassung der dreidimensionalen Struktur des Granzym A sind wichtige Voraussetzungen, um vorhandene Protease-Blocker optimieren und neue entwickeln zu können. Derartige Inhibitoren könnten bei Transplantat-Abstoßung, Autoimmunkrankheiten, wie rheumatoider Arthritis oder Multipler Sklerose, und bei chronisch verlaufenden Infektionskrankheiten praktische Anwendung finden.
Originalveröffentlichung:
Clara Hink-Schauer, Eva Estébanez-Perpina, Florian C. Kurschus, Wolfram Bode & Dieter E. Jenne
Crystal structure of the apoptosis-inducing human granzyme A dimer
Weitere Informationen erhalten Sie von:
PD Dr. Dieter Jenne
Max-Planck-Institut für Neurobiologie, Martinsried bei München
Tel.: 089 8578 – 3588
Fax.: 089 89950180
E-Mail: djenne@neuro.mpg.de
Dr. Wolfram Bode
Max-Planck-Institut für Biochemie, Martinsried bei München
Tel.: 089 8578 – 2676
Fax.: 089 8578 – 3516
E-Mail: bode@biochem.mpg.de
Media Contact
Alle Nachrichten aus der Kategorie: Biowissenschaften Chemie
Der innovations-report bietet im Bereich der "Life Sciences" Berichte und Artikel über Anwendungen und wissenschaftliche Erkenntnisse der modernen Biologie, der Chemie und der Humanmedizin.
Unter anderem finden Sie Wissenswertes aus den Teilbereichen: Bakteriologie, Biochemie, Bionik, Bioinformatik, Biophysik, Biotechnologie, Genetik, Geobotanik, Humanbiologie, Meeresbiologie, Mikrobiologie, Molekularbiologie, Zellbiologie, Zoologie, Bioanorganische Chemie, Mikrochemie und Umweltchemie.
Neueste Beiträge

Bakterien für klimaneutrale Chemikalien der Zukunft
Forschende an der ETH Zürich haben Bakterien im Labor so herangezüchtet, dass sie Methanol effizient verwerten können. Jetzt lässt sich der Stoffwechsel dieser Bakterien anzapfen, um wertvolle Produkte herzustellen, die…

Batterien: Heute die Materialien von morgen modellieren
Welche Faktoren bestimmen, wie schnell sich eine Batterie laden lässt? Dieser und weiteren Fragen gehen Forschende am Karlsruher Institut für Technologie (KIT) mit computergestützten Simulationen nach. Mikrostrukturmodelle tragen dazu bei,…

Porosität von Sedimentgestein mit Neutronen untersucht
Forschung am FRM II zu geologischen Lagerstätten. Dauerhafte unterirdische Lagerung von CO2 Poren so klein wie Bakterien Porenmessung mit Neutronen auf den Nanometer genau Ob Sedimentgesteine fossile Kohlenwasserstoffe speichern können…